文献解读|Nature Immunology(30.5):区分自然遗传变异对Kupffer 细胞表观基因组和转录组的细胞内在和环境依赖性影响
✦ +
+
论文ID
原名:Discrimination of cell-intrinsic and environment-dependent effects of natural genetic variation on Kupffer cell epigenomes and transcriptomes
译名:区分自然遗传变异对Kupffer 细胞表观基因组和转录组的细胞内在和环境依赖性影响
期刊:Nature Immunology
影响因子:30.5
发表时间:2023.09.21
作者/通讯作者:Hunter Bennett/ Christopher K Glass
单位:加州大学圣地亚哥分校细胞和分子医学系
DOI号:10.1038/s41590-023-01631-w
背 景
非编码遗传变异的常见形式,包括单核苷酸多态性 (SNP) 和短插入和缺失 (indel),可以通过改变增强子和启动子内转录因子的 DNA 识别元件的序列来改变基因表达。例如,减少所需转录因子与细胞特异性增强子的结合的SNP可以减少该细胞类型中相应基因的表达。非编码遗传变异驱动表型多样性,但潜在机制和受影响的细胞类型尚不完全清楚。
实验设计
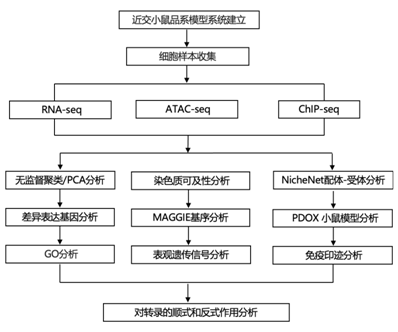
结 果
01
基因-环境相互作用影响基因表达
为了建立一个模型系统来分析自然遗传变异对Kupffer 细胞基因表达的影响,研究者团队选择了三种常见的近交小鼠品系,它们概括了在人类肝脏疾病中观察到的主要表型差异。他们生成了品系特异性骨髓源性巨噬细胞 (BMDM) 的转录组数据,以评估遗传变异对Kupffer 细胞影响的特异性。对这些数据进行的无监督聚类和主成分分析(PCA)表明,细胞类型是聚类的主要决定因素,但每个细胞类型根据品系分离(图1a)。配对比较各品系的Kupffer 细胞发现了194-362个差异表达基因(DEG),其中57-80%的品系差异调控基因仅在Kupffer 细胞或BMDM中发生了独特改变(图1b-d)。BMDM 中具有独特表达模式的转录本,包括显著的炎症调节因子(Rgs1、Ifi203和Aoah)、脂质反应(Ch25h、Abcg1和Sdc1)和极化因子(Arg2和Marco)(图1e-g)。同样,Kupffer 细胞中具有独特表达模式的转录本的例子包括转录(Atf5)和炎症(Cd300e、Irak3、Cxcl14、Cd40和Mefv)调节因子(图1-j)。Kupffer 细胞中的品系独特基因的表达与控制抗原加工和呈递、趋化因子信号传导和趋化性抑制的程序中的进化可塑性相关联(图1k-m)。
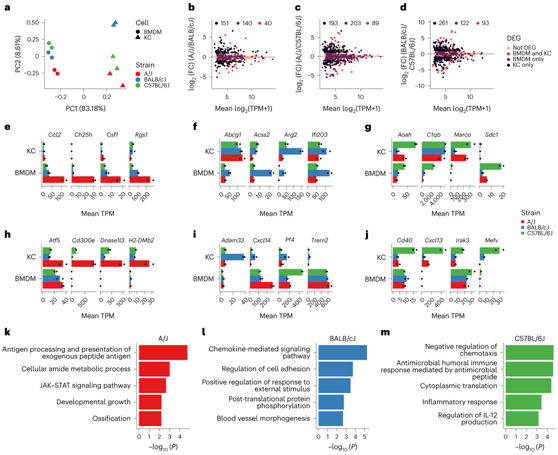
图1. 原代小鼠巨噬细胞的基因与环境转录调控。
(a) BMDM 和 Kupffer 细胞的 RNA-seq 数据的整体主成分分析。 (b-d) Kupffer 细胞的 RNA-seq 数据的平均 TPM 和 DeSeq2 log 2 (差异倍数) (e-j) BMDM和 Kupffer 细胞中代表性基因的表达。 (k-m) GO分析。
02
自然遗传变异对增强子景观的影响
为了研究遗传变异对 Kupffer 细胞中潜在 转录因子结合调控元件的影响,他们进行转座酶可及性染色质测序 (ATAC-seq)。成对比较了0-1000至>7000个差异ATAC-seq峰,发现差异峰的数量与遗传多样性水平成比例(图2a)。由于开放染色质不一定反映假定的调控元件的活性,因此他们对 H3K27ac 进行了染色质免疫沉淀,然后进行测序 (ChIP-seq),这与增强子和启动子活性高度相关。大约 5000–7000 个 ATAC-seq 峰按应变具有差异 H3K27ac 信号(图2b)。通过分析这些交叉数据,在约 66000 个假定的调控元件中,超过 10000 个受到遗传控制(图2c)。与品系特异性基因表达相关的ATAC-seq 和 H3K27ac ChIP-seq 的信号包括Cd300e、Trem2和Irak3(图2d)。
为了估计局部遗传变异对 ATAC-seq 和 H3K27ac 中品系特异性差异的影响程度,他们确定了与品系相似的 ATAC-seq 和 H3K27ac 峰处 ATAC 定义的开放染色质内 SNP 和插入缺失的频率ATAC-seq 或 H3K27ac 信号表现出大于两倍的变化。品系相似的峰表现出15-18%的背景SNP/插入缺失频率,而相差超过两倍的峰表现出46-56%的变异频率(图2e)。为了识别驱动品系特异性增强子选择的潜在转录因子,他们对品系特异性增强子进行了基序富集。在每种情况下,PU.1结合位点都是最富集的基序(图2f)。此外,假定的增强子在品系之间表现出优先的H3K27ac,包括富集了识别NFIL3、AP-1和ETS因子等的基序(图2f),并表明增强子活性和序列特异性转录因子之间可能存在因果关系。
为了进一步了解 SNP 和插入缺失对增强子选择和功能产生局部影响的机制,他们使用基序突变分析工具 MAGGIE 评估了这三种小鼠品系提供的遗传变异对开放染色质和 H3K27ac 的定量影响。MAGGIE 将同源序列的表观基因组特征的变化(例如增强子激活或增强子抑制)与遗传变异引起的基序突变联系起来,以优先考虑可能有助于局部调节功能的基序。MAGGIE 鉴定的许多基序是相关转录因子的结合位点(例如,ETS、AP-1 和 IRF 家族)(图2g)。总体而言,基序突变对开放染色质和 H3K27ac 的影响高度相关。

图2. 自然遗传变异对Kupffer 细胞表观基因组的局部和整体影响。
(a) 不可复制发现率(IDR) ATAC-seq峰联合集上ATAC-seq信号log2(标签计数)的散点图。 (b) H3K27ac ChIP-seq信号在所有品系的IDR ATAC-seq峰联合集的log2(标签计数)散点图。(c) 每个细胞系的活跃的和可及的基因组位点重叠。 (d) 与转录激活相关的细胞系特异性表观遗传信号。(e) 根据 ATAC-seq 和 H3K27ac ChIP-seq 数据将增强子分为细胞系相似性或特异性细胞系特异性。 (f) 与细胞系特异性活性增强子相关的基序,定义为在H3K27ac中具有品系特异性增加的位点。 (g) 细胞系差异可及性和活性增强子的MAGGIE基序突变分析。
03
环境对转录影响
利用来自 A/J、BALB/cJ 和 C57BL/6J 小鼠的肝细胞、星状细胞和肝窦内皮细胞 (LSEC) 新生成的 RNA 测序(RNA-seq)数据,他们确定了肝细胞是否表达具有同源受体的配体Kupffer 细胞中的表达可以预测品系特异性Kupffer 细胞基因表达(图3a)。利用NicheNet通过将特定配体诱导的受体细胞基因表达预测与一组品系特异性差异表达基因相关联,从而对每个配体进行评分,他们总结了得分最高的NicheNet配体和配体-靶基因连接(图3b-c)。肝细胞来源的配体包括ApoE,这是一种结合脂蛋白受体的载脂蛋白(例如LDLR和TREM2),预测它会诱导BALB/j特异性Kupffer细胞基因表达(例如Fads1和Cxcr4)(图3c)。
LSEC 衍生的配体包括骨形态发生蛋白 2 (BMP2),它是 BMP 配体家族成员,可控制 A/J 特异性Kupffer 细胞基因表达。LSEC 和肝星状细胞的预测生态位配体包括Adam17和App编码的蛋白质。App是编码淀粉样前体蛋白 (APP) 的基因,与 C57BL/6J 特异性基因表达相关,包括炎症反应基因Ccl5和Tnfaip3。Lep编码脂肪因子瘦素,预测它是BALB/cJ特异性Kupffer细胞基因表达的得分最高的配体,而Lepr编码瘦素受体,在BALB/cJ Kupffer细胞中表达最高(图3d)。总而言之,这些分析预测了调节品系特异性Kupffer 细胞基因表达的几种生态位配体-受体信号通路的表达或活性的改变。
他们研究了Lepr基因差异表达的重要性,在肝生态位的其他细胞类型中,Kupffer细胞表达的Lepr最高(图3d)。内源性瘦素完全来源于脂肪组织,并通过瘦素受体磷酸化和激活STAT3发出信号。在禁食小鼠中急性腹腔注射瘦素诱导C57BL/6J和BALB/cJ小鼠的肝组织中可检测到STAT3磷酸化,但在A/J小鼠中未检测到该修饰(图3e),在评估Kupffer 细胞中,A/J小鼠的Lepr表达最低(图3d)。此外,在实验NASH模型中,常驻(Tim4+)Kupffer 细胞的Lepr表达明显低于对照组小鼠,并且在C57BL/6J NASH小鼠的外周血单核细胞和单核细胞来源(Tim4-)Kupffer 细胞中Lepr表达极低(图3f-g)。
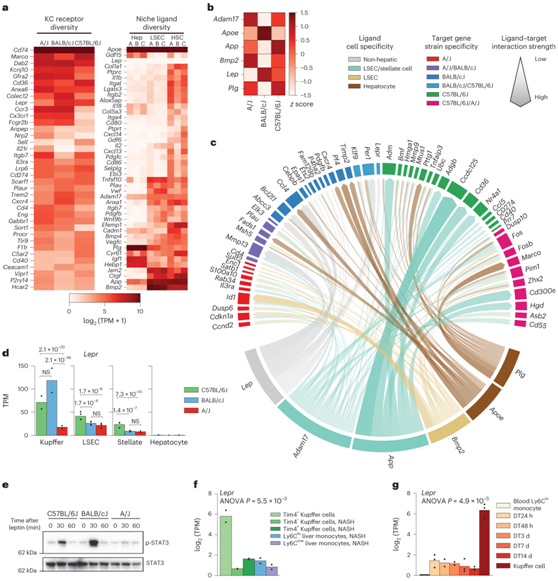
图3. 使用网络分析预测肝Kupffer 细胞生态位差异。
(a) Kupffer 细胞(左)的受体基因表达或来自指定细胞类型的生态位伴随细胞(右)的配体基因表达。 (b) 每个细胞系的最高 NicheNet 配体活性得分。(c) Circos 图展示了(b)中前 6 个 NicheNet 配体的基因靶标。 (d) 肝细胞中瘦素受体的品系特异性表达。(e)免疫印迹分析。(f) 编码瘦素受体的基因在健康C57BL/6J小鼠的Kupffer细胞(左)和骨髓细胞(包括从胰岛淀粉样蛋白肝NASH(AMLN)诱发NASH饮食20周的小鼠中分离的巨噬细胞和单核细胞)中的表达。(g) 编码瘦素受体的基因在胚胎来源的Kupffer 细胞(最右侧)和骨髓来源的单核细胞中的表达。
04
遗传变异对转录的顺式和反式影响
接下来,他们对第一代 C57BL/6J 雄性小鼠和 BALB/cJ 雌性小鼠杂交的 Kupffer 细胞进行 RNA -seq,以探索转录遗传变异的潜在机制。
他们在 F1 杂种中鉴定出了 245 个具有显著等位基因偏差的基因;在亲本 Kupffer 细胞的 RNA-seq 数据中,83 个基因对同一品系具有共同的基因表达偏差(图4a)。他们通过直接比较亲本细胞和等位基因数据之间的相对表达来定义顺式/反式表达模式。在等位基因水平,如果表达保守或不保守,则将品系特异性基因表达分别归类为顺式驱动或反式驱动。通过这种方法,他们发现,在健康Kupffer细胞中,反式基因的数量超过顺式基因(图4b),但反式基因在亲本品系中与较小的表达相关(图4c)。C57BL/6J和BALB/cJ的反式基因在C57BL/6J小鼠和BALB/cJ小鼠中的多种通路中富集,这些通路包括抗原呈递和toll样受体(TLR)信号通路,以及趋化和伤口愈合等通路(图4d)。反式基因启动子的从头基序分析确定了Kupffer 细胞中的不同转录调节因子,包括C57BL/6J小鼠中的NF-κB和BALB/cJ小鼠中的RXRα(图4e)。
由于反式效应可能由细胞自主和非细胞自主机制驱动,他们将来自C57BL/6J小鼠,BALB/cJ小鼠或CB6F1/J F1杂交小鼠的骨髓移植到Busulfan预处理的NOD scid γ (NSG)受体中来确定它们的相对贡献。Busulfan处理耗尽了驻留的Kupffer细胞,允许移植来自供体祖细胞的单核细胞来源的Kupffer细胞。F0-NSG和F1-NSG移植Kupffer 细胞具有相似的肝细胞外环境。因此,亲代Kupffer细胞中具有品系特异性基因表达偏向的基因,在F0-NSG和F1-NSG Kupffer细胞中具有等效表达的基因,可能是由亲代肝脏特有的环境差异驱动的(图4f)。来自亲代小鼠和F0-NSG移植模型的基因在Kupffer 细胞中表现出品系特异性表达,而在供体来源的F1-NSG模型中没有显著的等位基因失衡,这可能是由于细胞内信号传导或转录因子活性的差异。在品系特异性基因方面,F0和F0 - NSG小鼠比F1-NSG小鼠共有更多的差异基因,这与细胞内环境维持部分品系特异性基因表达的作用是一致的(图4g)。
在F1杂交和NSG模型中,大多数具有等位基因偏差的转录本是反式调节的,并且可能是由模型中丢失的亲代肝脏环境中的环境反式效应驱动的(图4h)。
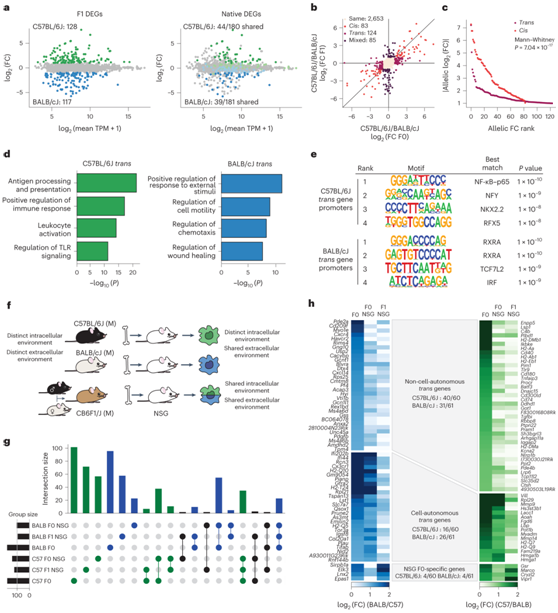
图4. 细胞自主和非细胞自主反式相互作用导致Kupffer 细胞中基因表达差异。
(a) F1 杂交小鼠中具有按品系着色的显著偏差基因的差异等位基因表达(左)或具有重叠的 F0 品系特异性基因的 F1 杂交小鼠中的等位基因表达(右)。 (b) 使用来自亲代小鼠的Kupffer 细胞(x轴)与来自F1小鼠的Kupffer 细胞(y轴)的基因表达比率的比较。 (c) 与顺式和反式基因相关的亲本系中等位基因差异倍数的累积分布。 (d) GO分析。(e) 与 BALB/cJ 或 C57BL/6J 特异性转基因相关的启动子中的 HOMER 从头基序富集。 (f) 实验示意图。 (g) UpSet图显示在亲本、F0-NSG移植和F1-NSG移植条件下BALB/cJ和C57BL/6J品系特异性基因的交叉。 (h) F1 和 NSG 模型中鉴定的转基因重叠。
05
遗传变异对增强子的顺式和反式影响
接下来,他们对从 CB6F1/J F1 杂交小鼠分离的 Kupffer 细胞中的 H3K27ac 进行了 ATAC-seq 和 ChIP-seq,并评估了通过遗传变异的存在来区分的等位基因差异。与基因表达结果一致,ATAC-seq 峰和叠加的 H3K27ac ChIP–seq 信号以反式效应为主(图5a)。总体而言,H3K27ac ChIP-seq 数据中的等位基因偏差与 ATAC-seq 和 RNA-seq 数据中的等位基因偏差呈正相关,表明表观遗传信号和基因表达中的等位基因特异性变化具有相关性(图 5b-c)。
为了鉴定驱动这些效应的反式调节的转录因子,他们对F1杂种小鼠的Kupffer 细胞中具有趋同的等位基因信号的反式调节的1184个ATAC峰和1493个H3K27ac位点进行基序富集分析。ETS因子基序优先富集在BALB/cJ Kupffer细胞特异的开放染色质的反式调节区域中,而CTCF和IRF基序富集在C57BL/6J Kupffer细胞特异的开放染色质中。
AP-1因子、MAF因子和FOXO1识别的基序在BALB/cJKupffer 细胞中富集于H3K27ac高水平的ATAC-seq峰,而IRF、LXR和NF-κB识别的基序在C57BL/6JKupffer 细胞中富集于H3K27ac区域(图5d)。
与反式基因相关的BALB/cJ增强子富集于SpiC、AP-1和MITF基序(图5f),而与反式基因相关的C57BL/6J增强子富集于SpiB、Rel/RelA NF-κB和异二聚体PU.1-IRF基序(图5e-f)。这些结果表明,在BALB/cJ Kupffer细胞中,AP-1上游的信号通路和因子具有品系特异性,而在C57BL/6J Kupffer细胞中,NF-κB具有品系特异性活性。
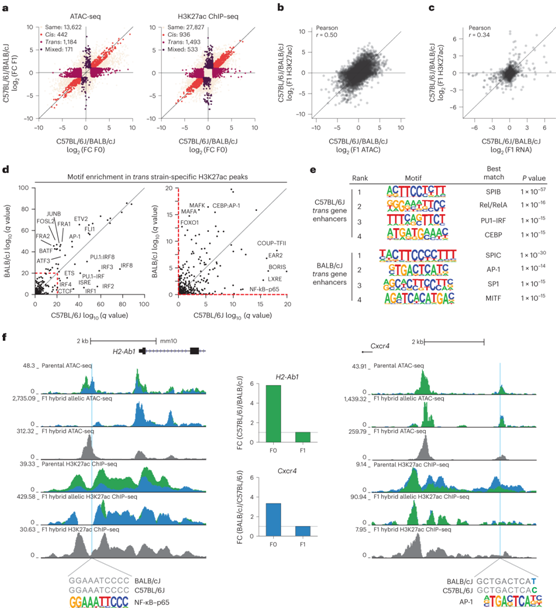
图5. 表观遗传位点的顺式和反式分析揭示了反式转录多样性的上游调节因子。
(a) 使用亲代小鼠(x轴)和 F1 杂交小鼠(y轴)的 Kupffer 细胞比较 ATAC-seq(左)和 H3K27ac ChIP-seq(右)的比率。 (b) IDR ATAC-seq 峰处的等位基因 ATAC-seq 和 H3K27ac ChIP–seq 读数的相关性。(c) 至少一个样本中 TPM > 4 表达的转录本的等位基因 RNA-seq 读数和启动子 H3K27ac ChIP-seq 读数的相关性以及具有超过 8 个 H3K27ac ChIP-seq 读数的启动子。(d) 反式BALB/cJ特异性增强子(y轴)和反式C57BL/6J H3K27ac增强子(x轴)中转录因子基序的富集得分。 (e) 与C57BL/6J或BALB/cJ转基因相关的活性增强子的从头基序富集。(f) 反式调节基因上游的反式调节表观遗传基因座的例子。
06
标题遗传变异影响脂多糖(LPS) 反应
为了深入了解遗传变异如何影响体内环境扰动的Kupffer 细胞反应,他们评估了 BALB/cJ 和 C57BL/6J 小鼠腹腔注射脂多糖(LPS) 2 小时后Kupffer 细胞转录组和开放染色质景观的变化。在两个亲本品系的 Kupffer 细胞中注射 LPS 后,共有 3268 个基因受到差异调节,其中 1885 个基因具有保守反应以及仅在一种品系中响应的 1383 个基因的品系分化(图6a)。两个亲本品系的Kupffer细胞在注射LPS后,6887个ATAC-seq峰的可及性发生了显著变化(图6b)。
由于差异倍数是由LPS和对照处理条件下的表达比决定的,因此品系特异性值中的任何一个或两个值的变化有助于检测品系特异性差异(图6c)。他们通过基础表达水平划分品系特异性基因来评估品系特异性转录和表观遗传机制。通过比较未处理的BALB/cJ或C57BL/6J小鼠的Kupffer细胞基因表达,将品系特异性LPS应答基因分为“低基础”(例如Slc7a2)、“等基础”(例如Med21)和“高基础”(例如Plac8)状态(图6c)。Slc7a2、Med21和Plac8启动子以及附近增强子的ATAC-seq峰分别说明了相应的低、等/高基础标准化标签计数(图6d)。等基础、高和低基础增强子分别占显示品系特异性 LPS 反应的 ATAC-seq 峰的77%、17% 和4%(图6e-f)。
将MAGGIE应用于这些基础状态峰值类别显示出明显的基序突变分离。信号依赖性转录因子(包括IRF、JUN/JUNB和NF-κB,它们在LPS介导的TLR4信号传导中起转录作用)的有害基序突变在无反应品系的相同基础ATAC-seq峰中富集(图6g)。
最后,他们将 LPS 处理峰与顺式调节、反式调节或混合调节与每个基础峰类别相交。辨别亲本特异性等位基因将品系特异性峰从 1261 减少到 459,其中 133 个峰表现出顺式调节,40 个表现出反式调节,22 个表现出混合调节(图6h)。这表明品系特异性 LPS 反应性主要是由信号依赖性转录因子结合位点的局部基序突变引起的。
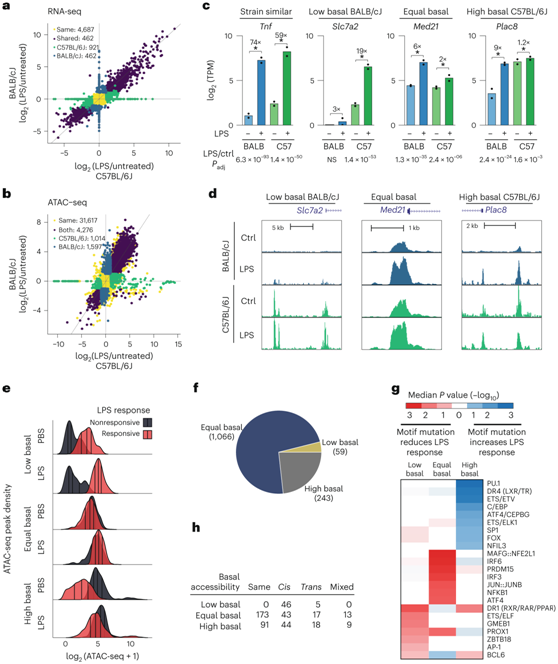
图6. 通过基序亲和力的顺式作用变化确定对LPS的品系特异性反应。
(a) C57BL/6J(x轴)和BALB/cJ(y轴)Kupffer细胞中LPS转录反应的比较。(b) C57BL/6J(x轴)和BALB/cJ(y轴)Kupffer细胞中对LPS的ATAC-seq反应比较。(c) 对LPS的品系相似和品系差异转录反应的例子。 (d) 与“低基础”、“等基础”和“高基础”基因相关的附近增强子处的 ATAC-seq 信号。 (e) “低基础”、“等基础”和“高基础”复合增强子中 ATAC-seq 信号的分布。(f) 每个基本可及性类别中的增强子数量。(g) MAGGIE基序突变分析,比较与每一类增强子相关的品系之间的基序得分。(h) 在从用LPS处理2小时的小鼠分离的CB6F1/J Kupffer细胞中进行顺式和反式增强子分析的三个增强子类别的重叠。
+ + + + + + + + + + +
结 论
本项研究通过研究自然遗传变异对近交小鼠品系的Kupffer 细胞表观基因组和转录组的影响,确定了影响Kupffer 细胞表型的品系特异性环境因素,包括来自脂肪性肝炎抗性品系的Kupffer 细胞中的瘦素信号传导。通过分析 F1 杂交小鼠和移植到免疫缺陷宿主中的细胞,探究了遗传变异的细胞自主和非细胞自主效应。在稳态期间,遗传变异的非细胞自主反式效应主导了Kupffer 细胞的控制,而对脂多糖(LPS)注射的品系特异性反应则由顺式作用效应的作用主导,这些顺式作用效应修饰了谱系决定和信号依赖性转录因子的反应元件。
+ + + + +



 English
English


