文献解读|BMC Genomics(4.547):高、低产长顺绿壳蛋鸡的卵巢组织转录组分析
✦ +
+
论文ID
原名:Transcriptome analysis of ovary tissues from low- and high-yielding Changshun green-shell laying hens
译名:高、低产长顺绿壳蛋鸡的卵巢组织转录组分析
期刊:BMC Genomics
影响因子:4.547
发表时间:2021.5.14
DOI号:10.1186/s12864-021-07688-x
背 景
鸡蛋价格便宜,且富含蛋白质、必需维生素和矿物质,是人们重要的食品来源。过去40年,全球鸡蛋消费量增长了2倍,因此,如何提高鸡蛋产量成为了一个重要的问题。提高鸡的遗传潜力是提高产蛋量的重要策略之一。然而,用蛋数量和产蛋率作为指标进行长期选择的常规育种技术,通常是费力和耗时的。近年来,高通量测序技术的发展为遗传选择提供了快速、准确的方法,已经越来越多地用于研究家禽繁殖。长顺绿壳鸡是贵州省的地方鸡种,是蛋肉兼用型鸡,其蛋具备外观好、蛋白质含量高、氨基酸组成更好、脂肪含量低等优点,具有极高的经济价值。因此,本研究旨在利用转录组测序技术分析高、低产长顺绿壳蛋鸡卵巢mRNA的差异,筛选出与产蛋率有关的通路和候选基因,为蛋鸡育种提供基础数据。
实验设计

结 果
01

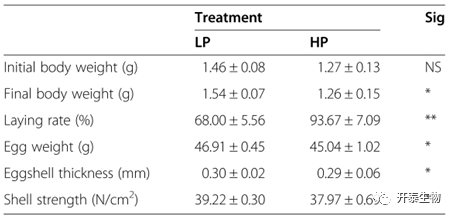
蛋鸡体重、产蛋量和蛋质量参数评估
在饲喂期间,高产组(HP)和低产组(LP)蛋鸡的体重、产蛋量和蛋质量参数详细信息见表1。HP组产蛋率极显著高于LP组(93.67 ± 7.09 vs 68.00 ± 5.56,p < 0.01)。然而,LP组的蛋重、蛋壳厚度和强度及终末体重均显著高于HP组(p < 0.05)。
图1 中国玉米育种中的根系结构变化
02

转录组测序质量评估
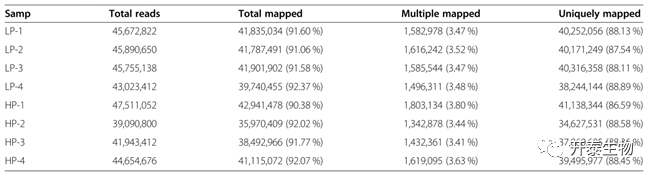
转录组测序数据质量指标如表2所示。本实验以长顺绿壳蛋鸡的卵巢为研究对象,构建了8个cDNA文库。除HP-2外(raw reads约为3920万,clean reads约为3900万),其余各文库的raw reads和clean reads分别在4200万和4100万以上。所有样品的转录本GC含量在49.11%~52.24%之间,Q20 > 97.79%,Q30 > 93.55%。因此,测序数据可用于后续数据分析。
表2 长顺绿壳蛋鸡卵巢转录本质量评估

03

转录组比对
参考基因组比对结果如表3所示。所有样品的reads与参考基因组之间的总比对率(Total mapped ratio)在90.30 ~ 92.37%之间。唯一比对率(Unique mapped ratio)在86.59 ~ 88.89%之间,表明转录组数据是可靠的,可用于后续分析。
表3 参考基因组比对结果
04

差异表达基因(DEGs)分析
PCA分析显示,HP和LP两组明显分为两个集群,表明HP和LP组之间转录谱存在差异。与LP组相比,HP组中共鉴定到142个DEGs,包括55个上调基因和87个下调基因(图2)。聚类分析直观地反映了LP和HP组之间基因表达模式的差异(图3)。

图1 卵巢转录组的PCA评分图
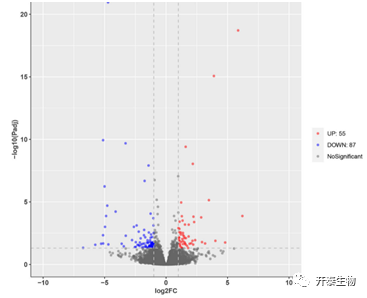
图2 所有表达基因的火山图
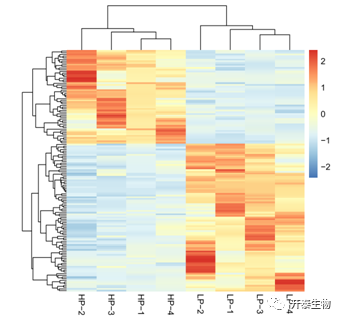
图3 DEGs的层次聚类分析
05

GO和KEGG分析
为了进一步阐明DEGs的生物功能,作者对DEGs进行了GO和KEGG通路分析。KEGG分析结果显示,DEGs显著富集到9条KEGG通路(p < 0.05,图4),包括神经活性配体-受体相互作用通路、补体和凝血级联通路、金黄色葡萄球菌感染通路、卵巢类固醇生成通路、催乳素信号通路、PI3K-Akt信号通路、cAMP信号通路、GnRH信号通路和TRP通道炎症介导调节通路(表4)。
GO分析表明,共220个GO条目被显著富集(FDR < 0.05)。生物过程(BP)的前25个显著富集GO条目以及分子功能(MF)和细胞组分(CC)的所有显著富集GO条目如图5所示。前25个显著富集的BP条目主要涉及:肽酶活性和内分泌过程的调节、分泌调节和细胞脂质输出;显著富集的CC条目为:含胶原的细胞外基质、分泌颗粒腔、胞质囊泡腔、囊泡腔;显著富集的MF条目包括:肽酶调节剂和抑制剂活性、受体配体活性、跨膜受体蛋白激酶活性和生长因子结合等。

图4 KEGG通路富集分析
表4 显著富集的KEGG通路
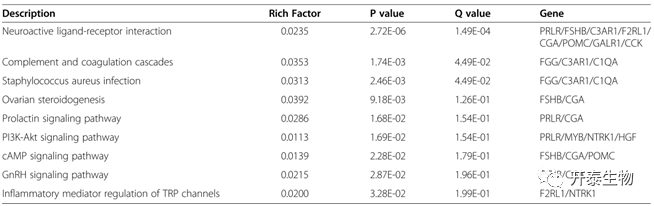

图5 GO富集分析
06

RT-qPCR验证
为了验证RNA-Seq结果,选择了6个DEGs(三个上调基因AMN、POMC和CGA;三个下调基因OVA、OVALX和OVALY)进行RT-qPCR。结果表明,RT-qPCR检测的mRNA表达趋势与RNA-Seq结果一致(图6),再次表明RNA-Seq结果的可靠性。

图6 转录组测序和RT-qPCR结果之间的一致性
+ + + + + + + + + + +
结 论
综上所述,本研究对高、低产长顺产蛋鸡的卵巢转录组进行了鉴定和评价。共鉴定出142个差异表达基因,可作为产蛋性状遗传改良的候选基因。GO和KEGG富集分析表明,神经活性配体-受体相互作用通路和跨膜受体蛋白酪氨酸激酶通路可能在卵巢功能和产蛋量的调控中发挥重要作用。
+ + + + +



 English
English

