文献解读|Cell Rep Med(14.3):肝胆肿瘤类器官的综合表征提供了药物基因组相互作用的潜在前景
✦ +
+
论文ID
原名:Integrated characterization of hepatobiliary tumor organoids provides a potential landscape of pharmacogenomic interactions
译名:肝胆肿瘤类器官的综合表征提供了药物基因组相互作用的潜在前景
期刊:Cell Reports Medicine
影响因子:14.3
发表时间:2024.01.17
DOI号:10.1016/j.xcrm.2023.101375
背 景
肝胆肿瘤是全球第四大癌症相关死亡原因,全球每年约有 70 万人死亡。肝胆肿瘤包括 20 多种不同的亚型,这些亚型在遗传、形态和临床上各不相同。患者来源的肝胆肿瘤类器官 (PDHO) 提供了研究肿瘤组织学和分子特征的机会。
实验设计
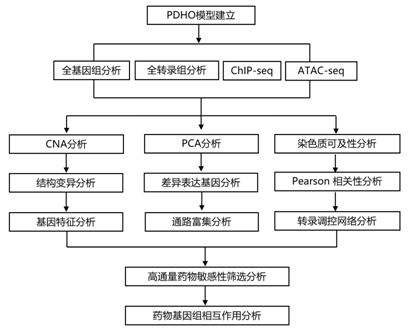
结 果
01
PDHO 保持亲本肿瘤特征
为了建立中国肝胆肿瘤的临床相关模型,研究者团队生成了包含 64 个 PDHO 细胞系的生物库,可以在 3D 培养中轻松繁殖(图 1A)。将分离的原代肿瘤细胞接种在基质胶滴中,并用优化的肝胆肿瘤类器官培养基覆盖。为了支持生长并维持长期扩张,多种小分子和生物制剂,包括表皮生长因子 (EGF)、成纤维细胞生长因子(FGF2 和 FGF10)、肝细胞生长因子 (HGF)、Wnt 激动剂 R-spondin1、添加转化生长因子 β (TGF-β) 抑制剂 Noggin 和 ROCK 抑制剂 Y-27632(图 1 A)。总共产生了64个来自不同原发性肝胆肿瘤患者的类器官,这其中来源于46例肝细胞癌(HCC)患者、8例肝内胆管癌(ICC)患者、4例肝细胞胆管癌(CHC)合并患者,4 名胆囊癌 (GBC),1 名肝门部胆管癌 (HCCA),1 名结肠癌转移癌(图 1 A)。GBC 类器官的管状结构,与其患者特异性和肿瘤特异性组织病理学特征一致(图 1 B)。一些 HCC 还形成假腺花结,这是 CHC 的典型特征(图 1 B)。
拷贝数改变 (CNA) 通常影响肝胆肿瘤基因组的很大一部分。他们首先比较了全基因组 CNA,正如预期的那样,发现了多个染色体重排。DNA 拷贝数的增加和丢失保留在整个基因组中,PDHO 通常显示出比原始肝胆肿瘤更清晰、更明显的拷贝数信号(图 1 C)。这些 CNA 广泛影响多种癌症基因。例如,包含典型癌症基因的区域中的局部扩增包括 11q13.3 (CCND1) 中的区域,而局部缺失涉及 9p21.3 (CDKN2A),与原发性肝胆肿瘤中的情况类似。结构变异 (SV) 涉及 DNA 片段的缺失、重复、倒置和易位,是体细胞遗传变异的主要来源,可能导致癌症相关通路失调。他们在 PDHO 中检测到 5845 个体细胞 SV,其中 733 个是染色体间的。正如预期的那样,大多数 SV 在肝胆肿瘤-类器官对之间是保守的(图 1 D)。乙型肝炎病毒(HBV)可以整合到人类基因组中,导致基因组不稳定和肝癌发生。他们在肿瘤和类器官模型之间的 HBV 整合优先位点中观察到类似的模式(图 1 E),这表明 PDHO 重现了癌症基因的 HBV 整合模式,并可能为 HBV 提供潜在的研究模型。相关肝胆肿瘤。
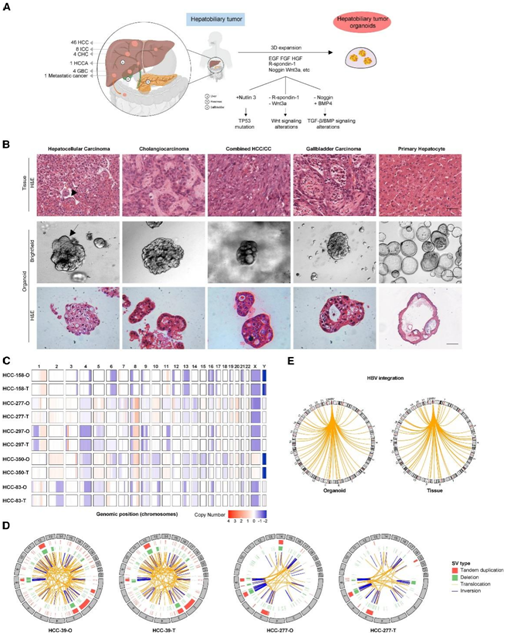
图1. 患者源性肝胆肿瘤类器官生物库的建立。
(A) 优化建立方法图和迄今为止为生物库建立的 64 个肝胆肿瘤类器官及其不同分子亚型范围的总结。(B) 健康肝组织和原发性肝胆肿瘤的代表性 H&E 染色(上)和相应的明视野显微镜图像(中)以及源自这些的类器官系的 H&E 组织学分析(下)。(C) 基因组图显示了肝胆肿瘤组织-类器官对的全基因组 CNA。(D) circos 图代表 HCC-39 和 HCC-277 配对组织/类器官样品中的所有 SV。(E) 组织和类器官样本中 HBV 整合事件的 Circos 图。
体细胞中出现的突变是癌症发展的驱动力。根据 COSMIC 和 OncoKB 数据库,总共有 5276 个基因具有非沉默突变,其中 243 个基因是癌症相关基因(图 2 A)。原发性肝胆肿瘤中与癌症相关的改变在患者来源的类器官模型中捕获,其中包括肿瘤抑制基因 TP53 (77%)、AXIN1 (25%)、CDKN2A (10%)、PTEN (10%) 和 RB1(9%)因突变而失活;WNT 通路癌基因 CTNNB1 (10%)和染色质重塑基因 ARID1A (14%)。这些结果概括了主要的基因组特征,并且与已发表的 TCGA 结果一致,但 TP53 突变除外,他们从中推断 TP53 突变的肝胆肿瘤更有可能生长成类器官(图2A)。
结果表明,特征B和C分别与COSMIC特征22和24具有较高的相似性(图2A)。例如,已知特征22与马兜铃酸暴露有关,而特征24与黄曲霉毒素暴露有关。基于本项研究的数据和COSMIC特征之间的相关性,分层聚类产生了四组肿瘤(突变特征1 [MS1-MS4])(图2A)。这些聚类的特点是富集了特定的突变特征和遗传改变。例如,MS1以TP53、CDKN2A、NTRK3和SDHA突变为特征。MS2与TERT启动子突变和AXIN1突变存在显著相关(图2A-B)。此外,MS3的肿瘤突变负担(TMB)高于其他聚类(图2A-B),这可能是由于BRCA1突变导致DNA损伤敏感性和基因组不稳定。
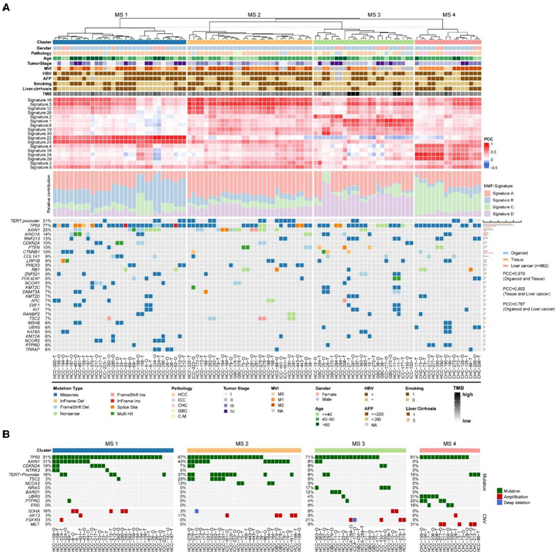
图2. 肝胆肿瘤类器官队列的基因组图谱。
(A) 上:基于突变特征的肝胆肿瘤组织和类器官分类以及相关的临床病理特征以及总突变负荷。中:显示了本项研究的数据的上下文特定突变谱与 COSMIC 数据库中的 MS 之间的皮尔逊相关系数 (PCC),并附有堆叠条形图,其中显示了每个患者的每个非负矩阵分解 (NMF) 特征的贡献。下:肝胆肿瘤组织和类器官中主要癌症相关基因的遗传图谱(左)。条形图显示了这些基因在肝胆肿瘤组织、类器官和 TCGA 中的改变频率。计算了肿瘤组织、类器官和 TCGA 之间基因频率的 Pearson 相关性(右)。(B) Oncoplot 显示了每个基于 MS 的肝胆肿瘤类器官中最重要的癌症相关基因的基因组特征。
02
PDHO 概括了原发肿瘤的转录组学概况
他们对进行了转录组分析(RNA-seq),并根据 16410 个基因组装了相关热图。为了阐明 PDHO 之间的转录组多样性,他们对 RNA-seq 数据进行了无监督聚类。通过主成分分析 (PCA) 和基于前 2000 个最可变基因的无监督聚类,他们进一步确定了类器官队列中的三个亚组(图 3 A)。亚组1的特点是增殖基因的增加,如TOP2A、MKI-67、CCNB1和CCND3,并表现出增殖相关通路的功能富集,包括E2F靶标、G2-M检查点和有丝分裂纺锤体。亚组2的特点是代谢相关基因水平最高,例如ADH1A、G6PC、CYP3A和CYP2E1,它们调节代谢重编程、过氧化物酶体和肝功能。亚组3表现出炎症和免疫相关基因的特异性表达,例如ITGB1、CD44、CD82、LAMB3和AGRN,主要调节细胞粘附和肿瘤免疫(图3B-C)。有趣的是,基因突变和基于拷贝数变异(CNV)的基因组不稳定性在三个亚组之间显示出显著差异:KMT2A和GNAQ仅在亚组1中突变,而AXIN1在亚组2中优先突变。此外,亚组的突变负荷1 普遍高于其他亚组(图 3 D)。非编码突变广泛存在于整个基因组中,根据 RNA 分类,他们鉴定出了大量亚组特异性非编码突变。这些非编码突变可能单独充当癌症中的“微型驱动因素”(图 3D)。
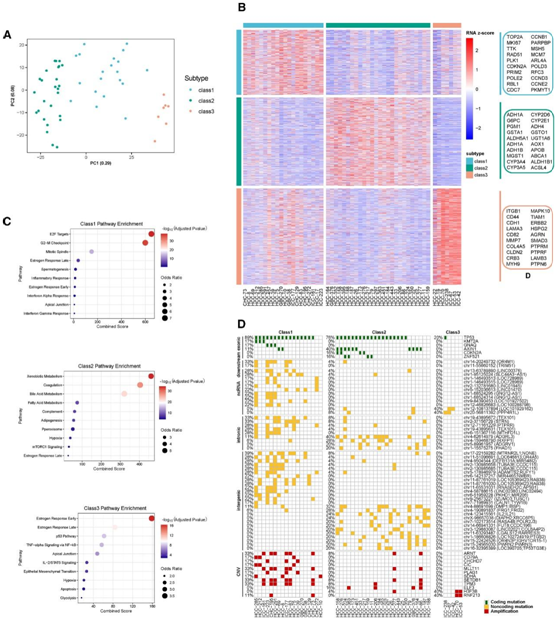
图3. 肝胆肿瘤类器官转录组分析。
(A) 对所有样本中 2000 个表达差异最大的蛋白质编码基因的 mRNA 表达值进行主成分分析 (PCA)。(B) 每个亚组差异表达基因的热图。(C) 每个亚组中显著差异表达基因的富集通路。(D) 条形图显示基于 mRNA 亚型的肝胆类器官中癌症相关基因的基因组特征。
03
染色质可及性图谱提供了对肝胆肿瘤基因调控网络的深入了解
他们通过 ATAC-seq 对 53 个 PDHO 模型的染色质可及性进行了全基因组分析。大多数 ATAC-seq 峰映射到远端基因间和内含子区域,但三种亚型之间发现的峰数量没有显著差异(图 4 A)。本研究队列中生成的肝胆肿瘤特异性峰集能够量化已识别的 DNA 调节元件的数量。ATAC-seq 定义的 DNA 调控元件与染色质免疫沉淀测序 (ChIP-seq) 定义的 ChromHMM 调控状态的重叠显示启动子和增强子区域中可及染色质位点的强烈富集。在 MYC 位点,他们鉴定出 chr8:128746252–128748838 是基因组中的一个活跃调控元件,具有广泛的染色质可及性,可促进广泛的基因表达并导致不受限制的细胞生长。这种趋势在不同的肝胆肿瘤样本中是一致的,如具有高增殖能力的亚组 1(图 3C-D)。
为了进一步进行生物学表征,他们将 ATAC-seq 数据与 RNA-seq 数据进行了整合。他们对 DNA 调控元件的基因表达和染色质可及性进行了 Pearson 相关性分析,发现远端元件表现出更大的特异性和与基因表达相关的更广泛的动态范围,而启动子元件的可及性特异性较低,并且两个元件都显示出模式相关性类似于整体基因表达(图 4 B)。他们研究了基于 RNA 分类的 ATAC-seq 可及性概况,以识别 4266 个亚组特异性峰(图 4 C)。
为了进一步预测与肝胆肿瘤分子亚型相关的相互作用,他们鉴定了 1256 个亚型特异性峰-基因链接,其基因和峰以亚型特异性方式连接。尽管不同亚组中的这些峰与基因的联系是由相似的调控区域驱动的,但大多数这些联系都是由一个聚类强烈驱动的(图4D)。此外,该分析还得出了每个亚组的监管峰与基因链接的最终列表,例如亚组 1 中的 MAP3K5 和 CDKN2A、亚组 2 中的 CP 和 ABCA1 以及亚组 3 中的 LAMC2 和 ITGB7(图 4 E)。
转录调控网络(trn)描述了转录因子(tf)与其靶基因(tgg)之间的调控相互作用。为了鉴定主转录调控因子,他们分析了通过基因激活或抑制促进亚群特异性基因表达的核心tf,发现了24个正调控的tf(图4F)。核心TRN分析正确地恢复了已知的亚群特异性调节因子:subgroup1中的FOXM1和FOSL1;亚组2中的DDIT3、XBP1、ZNF467;第3亚组为HOXA,trn还能准确预测特定的TF-TG边缘(图4F)。
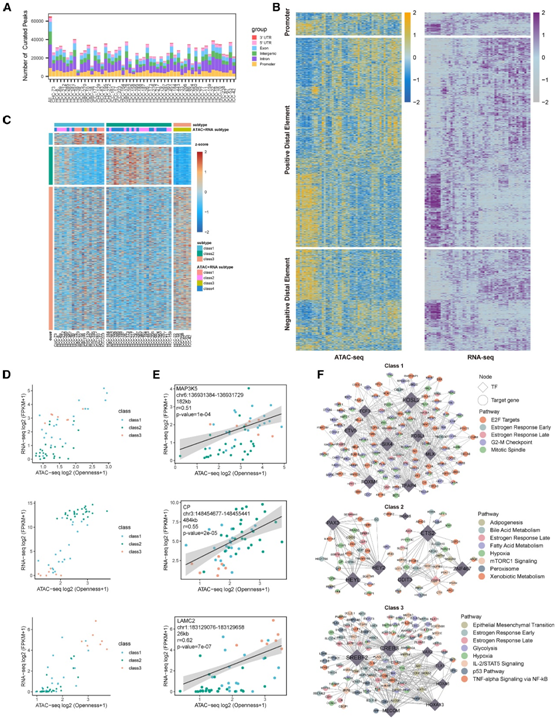
图4. 染色质可及性图谱揭示了肝胆肿瘤的不同分子亚型。
(A) 来自所有样本的 ATAC-seq 数据的精选峰调用数量。颜色表示峰重叠的基因组区域的类型。(B) ATAC-seq 启动子、ATAC-seq 远端元件(左)和所有基因的 RNA-seq(右)的 Pearson 相关热图。(C) 基于 mRNA 分类的 4266 个亚组特异性峰的热图表示。(D) 亚组特异性基因峰与基因链接的 ATAC-seq 可及性和 RNA-seq 基因表达的点图。(E) MAP3K5 基因、CP 基因或 LAMC2 基因的峰到基因链接的 ATAC-seq 可及性和 RNA-seq 基因表达的点图。(F) 亚组特异性转录因子 (TF)-靶基因 (TG) 基因网络,注释有每个亚组中丰富的通路。
04
PDHO 中的功能性高通量药物筛选鉴定出潜在的抗癌化合物
基于转录亚组,他们根据癌症生物学中的20个关键途径和细胞过程筛选出301种化合物,包括36种抗癌药物、36种表观遗传药物和229种代谢相关化合物(图5A)。首先,他们定义了药物反应评分(DRS),它是每种药物的 AUC 值的归一化Z评分,以反映整个 PDHO 的药物反应。
他们鉴定了 1082 个编码突变-药物对、68 个启动子突变-药物对、3417 个非编码突变-药物对、9573 个基因-药物对和 18191 个峰-药物对,中位数为 450 个特征,包括 78 个突变(16 个编码突变、1 个启动子突变和 61 个非编码突变)、139 个表达特征和 233 个染色质可及性特征确定为每种药物的预测(图 5 B)。值得注意的是,预测特征包括已知的基因药物关联,例如 KEAP1-β-Lapachone 的关联和 NF1-afatinib(图 5C)。他们还发现了影响 PDHO 对药物敏感性的启动子和非编码突变(图 5D)
在个体遗传特征与药物的关联中,他们发现了许多不同的药物基因组学关系。其中包括 NF1、AGRN 和 SETDB1 改变之间的详细描述的遗传相关关联;KMT2B、HLA-F-AS1 和 PCDHB7 的基因表达以及针对各自蛋白质产物或途径的临床药物的敏感;以及逐渐认识到的NR2F2启动子、RUNX1启动子、非编码突变、ATAC-seq可及性以及基于癌症发生和进展中的调节功能的药物敏感性之间的表观遗传学相关关联(图5E)。有趣的是,他们发现 RUNX1 启动子突变与药物敏感性相关(图 5D-E)。在这种情况下,该位点处的染色质发生了显著的偏差,该位点通常不可接近(灰点和轨迹),但在 RUNX1 启动子突变的情况下变得非常容易接近(图 5 F)。正如预期的那样,RUNX1 启动子可及性的增加与 RUNX1 的一种同工型的表达增加相关,发现一组药物在具有 RUNX1 启动子突变的样品中优先敏感(图 5G-H)。
与在肝胆肿瘤类器官中观察到的结果一致,短期生长抑制测定显示,与对照细胞相比,RUNX1 敲低细胞对伊立替康治疗更具抵抗力(图 5 I)。相反,RUNX1 的外源表达使 HCC-LM3 和 Huh-7 细胞对irinotecan处理更加敏感(图 5 J)。为了进一步探讨RUNX1启动子突变在药物脆弱性中的潜在调节作用,他们通过在已识别的RUNX1启动子基序中用鸟嘌呤(G)替换腺苷(A),进一步构建了突变的RUNX1启动子报告质粒(chr21_36262405)。双荧光素酶报告基因检测显示,与野生型相比,突变型RUNX1启动子的相对荧光素酶活性显著增加,证实了突变对RUNX1启动子活性的调节(图5K),这意味着RUNX1启动子活性的增强RUNX1启动子可能与肝胆肿瘤中伴随的基因表达增加和药物敏感性有关。这些结果揭示了与药物敏感性相关的这些基因组特征,并提供了肝胆肿瘤的潜在预测药物基因组景观。
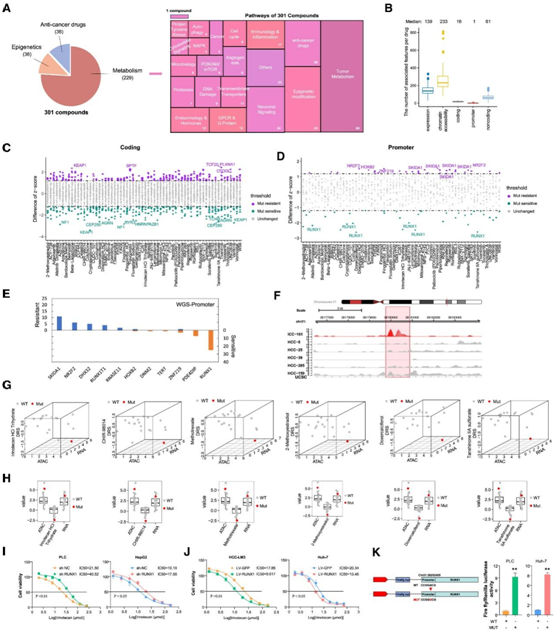
图5. 药物敏感性的药物基因组模型。
(A) 301 种筛选药物根据其治疗靶点、主要效应途径和细胞功能进行分类。(B) 箱线图显示每种药物的相关特征数量。(C-D) 散点图代表独特遗传特征-药物对之间具有统计显著性的相互作用。(E) 条形图显示肝胆肿瘤类器官中启动子突变的药物基因组相互作用。(F) RUNX1 启动子基因座处 6 个类器官样本的标准化 ATAC-seq 测序轨迹。(G-H) 本研究中描述的肝胆肿瘤类器官的归一化 ATAC-seq、RNA-seq 和药物反应。(I-J) 用增加浓度的伊立替康处理所示细胞系 72 小时,并计算它们的细胞存活曲线。(K) WT 或突变体(chr21_36262405 A-to-G 突变)RUNX1 启动子在用相应质粒转染的 PLC/Huh-7 细胞中的相对荧光素酶活性。
05
综合分析表征人类肝胆肿瘤的治疗特征
在各个特征-药物关联中,他们发现了许多重要的药物基因组学关系。瑞戈非尼(regorafenib)相关的遗传特征引起了人们的极大兴趣,因为瑞戈非尼已批准用于治疗索拉非尼病情进展后的不可切除的肝癌。CCND1 和 FGF19 是瑞戈非尼敏感性最重要的预测基因组特征(图 6 A)。FGF19/FGFR4 自分泌信号传导是多激酶抑制剂 (MKI) 的主要靶点之一。他们还鉴定了 PABPC1L 表达,它与瑞戈非尼耐药性高度相关(图 6 A)。
铁死亡作为一种有前景的杀死治疗耐药性癌症的方法最近引起了人们的广泛兴趣。RNA-seq 和 ATAC-seq 数据的综合分析发现了一组与脂质代谢相关的基因,例如 SLC30A2、CD74、LCN2 和 AGXT,它们介导非编码 DNA 调节元件的功能可及性,并且可能是产生敏感性的预测因子为铁死亡激活剂 RSL3(图 6 B)。
许多基因组改变同时发生或以相互排斥的方式发生,表明具有生物学功能。通路特异性药物基因组学分析确定了 64 种药物的预测模型(图 6 C)。与药物反应密切相关的信号通路包括磷脂酰肌醇3激酶(PI3K)/Akt、Myc、Nrf2、SWI/SNF和TGF-β信号传导。他们进一步研究了优先改变药物脆弱性的信号通路中的关键突变(图6D)。例如,细胞周期途径中肿瘤抑制基因 RB1 的截短突变预计与脂质过氧化抑制剂氟伐他汀钠的敏感性相关(图 6D)。
为了研究不同分子数据组合预测药物反应的能力,他们建立了 WGS、RNA-seq 和 ATAC-seq 药物敏感性的随机组合模型。对于每种药物,他们确定了表现最佳的数据类型组合和相应模型(称为“参考模型”),用于根据潜在治疗方法对患者进行分类(图 6 E)。例如,极光激酶抑制剂 tozasertib 显示与 KMT2A 错义突变和 KMT2C 表达特征(存在于 65% 的 PDHO 中)的染色质重塑信号相关(图 6 E)。此外,利用组合模型,他们研究了基于 RNA 分类的肝胆肿瘤的亚组特异性药理学特征和个体化治疗。亚组 1 显示Hippo 通路在基因组、转录组和表观遗传水平上存在显著异常。亚组 2 倾向于表现出对 RSL3 的抵抗力,并在 RTK/RAS 信号异常中具有专门的活性(图 6 F)。本研究中生成的“参考模型”可能是短期内对患者进行不同癌症治疗分层的有效手段。
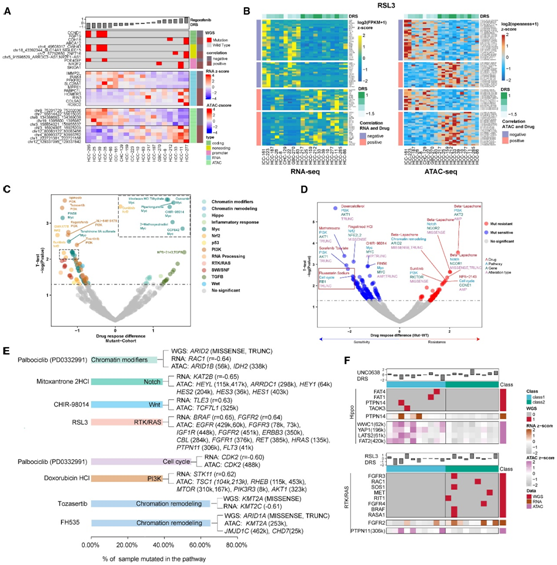
图6. 综合药物基因组分析重新定义药物反应。
(A) 热图显示与瑞戈非尼反应相关的顶级预测突变、表达和 ATAC-seq 可及性特征。(B) RNA-seq 和 ATAC-seq 之间预测 RSL3 响应的集成分析的热图表示。(C) 组合基因组相互作用的火山图。(D) 火山图代表信号通路-药物相互作用中关键突变的大小(药物反应差异)和显著性。(E) 信号水平基因组特征满足给定药物敏感性组合模型的肝胆肿瘤类器官的百分比。(F) 热图显示基于 RNA 分类的肝胆肿瘤的亚组特异性药理学特征和个体治疗方法。
+ + + + + + + + + + +
结 论
本项研究观察到对 265 种代谢和表观遗传学相关化学物质以及 36 种抗癌药物的不同反应。64 种临床相关药物的全基因组、转录组、染色质可及性特征和药物敏感性结果的整合定义了超过 32000 种基因组-药物相互作用。RUNX1启动子突变与染色质可及性的增加以及随之而来的基因表达的增加有关,促进一系列药物在肝胆肿瘤中敏感。这些结果不仅提供了带注释的人类肝癌PDHO生物库,而且还提出了一种系统方法来全面了解肝癌的基因调控网络,推进潜在个性化医疗的应用。
+ + + + +



 English
English


