文献解读|Immunity(32.4):潜伏和活跃 HIV-1 储存库的单细胞表观遗传、转录和蛋白质分析揭示IKZF3促进 HIV-1 持久性
✦ +
+
论文ID
原名:Single-cell epigenetic, transcriptional, and protein profiling of latent and active HIV-1 reservoir revealed that IKZF3 promotes HIV-1 persistence
译名:潜伏和活跃 HIV-1 储存库的单细胞表观遗传、转录和蛋白质分析揭示IKZF3促进 HIV-1 持久性
期刊:Immunity
影响因子:32.4
发表时间:2023.11.14
DOI号:10.1016/j.immuni.2023.10.002
背 景
尽管采用抑制性抗逆转录病毒治疗(ART),HIV-1 感染的细胞仍会终生持续存在。了解 HIV-1 感染细胞如何增殖和持续存在是根除 HIV-1 的关键,但 HIV-1 感染细胞的异质性和稀有性阻碍了机制研究。
实验设计
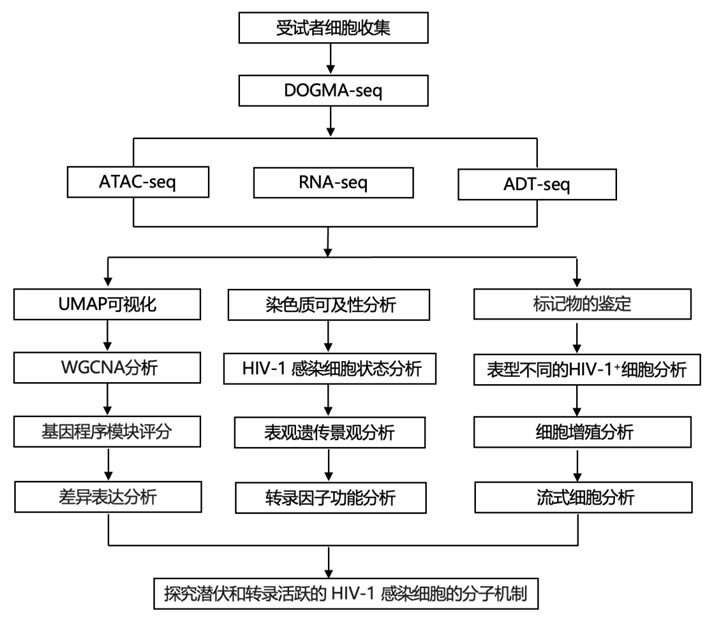
结 果
01
单细胞 DOGMA-seq 捕获了 HIV-1 感染期间记忆 CD4 + T 细胞的表观遗传、转录和表面蛋白状态
在本研究中,研究者团队研究了6名HIV-1感染者在病毒血症期间和抑制性ART后,转录非活性(HIV-1 DNA+ RNA-)和转录活性(HIV-1 RNA+) HIV-1感染的记忆CD4+ T细胞的表观遗传、转录和表面蛋白表达。使用寡核苷酸标记的多重扩增与深度测序相结合的数字化基因表达谱分析高通量测序技术(DOGMA-seq),通过转座酶可及染色质测序分析 (ATAC-seq) 确定了染色质可及转录因子结合区,通过RNA测序 (RNA-seq) 确定了转录程序,并通过抗体衍生标签测序 (ADT-seq) 确定了同一单个细胞内156种表面蛋白的细胞表面蛋白表达(图1A)。ATAC-seq、RNA-seq和ADT-seq的加权组合允许在统一加权最近邻(WNN)统一流形近似和投影(UMAP)上对同一单个细胞内的表观遗传调控因子、转录程序和表面蛋白表达进行综合分析(图1A)。他们确定了15个记忆CD4+ T细胞亚群(聚类)(图1B),在每个细胞中确定了中位785个基因、1553个RNA唯一分子标识符(UMI)、62个表面蛋白、700个ADT UMI、3143个独特的ATAC片段和4198个ATAC UMI。
为了识别在病毒血症期间诱导的不同基因程序,他们进行了加权基因相关网络分析(WGCNA),以识别在病毒血症细胞中高度共表达的基因。WGCNA在病毒血症中鉴定出5个有统计学意义的基因模块,其中增殖基因程序显示增殖基因MKI67、细胞存活基因BIRC5 (Survivin)和T细胞活化基因ICOS共表达。为了对比在增殖基因程序中鉴定的基因之间RNA表达的相关程度,将基因对RNA表达的Pearson相关系数可视化并在热图中进行比较(图1C-D)。事实上,与病毒抑制和未感染条件相比,在病毒血症条件下,增殖程序中的基因有更高的RNA共表达。为了验证在病毒血症期间增殖基因程序在增殖聚类中最富集,他们检测了增殖基因程序的标准化每个细胞平均RNA表达(模块评分)作为基因程序富集的评分。在病毒血症中,增殖基因程序得分总体上高于病毒抑制和增殖聚类中未感染条件下的得分(图1E)。
接下来,他们将重点放在增殖聚类上,以确定HIV-1感染与未感染条件下增殖T细胞的细胞状态(图1F-H)。他们首先鉴定了具有富集的结合基序的转录因子,这些结合基序无法结合染色质。在病毒血症期间,I型IFN应答的转录因子(IRF和STAT1/STAT2)、T细胞活化(AP-1 [JUN、FOS和BATF]、NFAT)、效应因子分化(TBX21和Eomes)和记忆发育(FOXO1)的可及性均发生富集(图1G)。
他们确定了在病毒血症的增殖聚类中,染色质可及性显著增加(ATAC峰),并且与DNA元件百科全书 (ENCODE) 候选顺式调节元件 (cCRE) 重叠和RNA表达增加的基因区域(图1G-J),以及在这些可及性增加的区域中存在的相应转录因子结合基序(图1I)。例如T细胞分化基因(IKZF3、CCL5和IFNG)、I型IFN应答基因(IFI16)和T细胞增殖基因(IL2RB)。例如,在病毒血症中, IKZF3(编码Aiolos,一种调节淋巴细胞增殖和分化的Ikaros锌指家族转录因子)的可及性增加(图1I)。在这个高度可及的足迹中发现的基序与IRF7、JUN:FOS、NFAT和T-box转录因子结合,这些转录因子在病毒血症的增殖聚类中都具有丰富的结合基序可及性(图1F)。与染色质可及性增加一致,IKZF3在增殖聚类中增加了RNA表达(图1J)。为了更直接地展示突出的cCRE处的染色质可及性和RNA表达之间的关系,他们发现在增殖聚类中,IKZF3 RNA+细胞的IKZF3基因处的染色质可及性高于IKZF3 RNA-细胞(图1I-J)。他们确定了Aiolos (IKZF3)在CCL5位点的结合和CCL5在增殖细胞中的表达之间的关系,发现CCL5 cCRE中的Aiolos结合基序在病毒血症中增加了染色质可及性(图1I)。其次,与Aiolos结合基序可及性较低的增殖细胞相比,相同的CCL5 cCRE在Aiolos结合基序可及性富集的增殖细胞中具有更高的染色质可及性。与Aiolos结合基序可及性较低的增殖细胞相比,Aiolos结合基序可及性较高的增殖细胞CCL5表达上调。最后,对CCL5增殖细胞和CCL5−增殖细胞的差异转录因子结合基序可及性(chromVAR)分析显示,IKZF3显著提高了CCL5细胞的整体可及性。除了Aiolos外,AP-1、IRF、STAT1:STAT2和T-box家族转录因子也可能结合在CCL5 cCRE上(图1I)。
为了确定增殖细胞在病毒血症期间诱导的免疫通路,他们在增殖细胞群中发现了病毒血症中显著上调的基因(图1G)。
最后,差异表达蛋白分析表明,在增殖的CD4+ T细胞中,病毒血症期间,活化标志物、Th1极化(CXCR3、CCR5和CD119 [IFNγ受体])和T细胞迁移(整合素α4和整合素β2)显著上调(图1H)。值得注意的是,与病毒抑制和未感染情况相比,在病毒血症期间,IL2RB在增殖细胞中始终具有较高的染色质可及性(图1I)、较高的RNA表达(图1G-J)和较高的表面蛋白表达(图1H)。
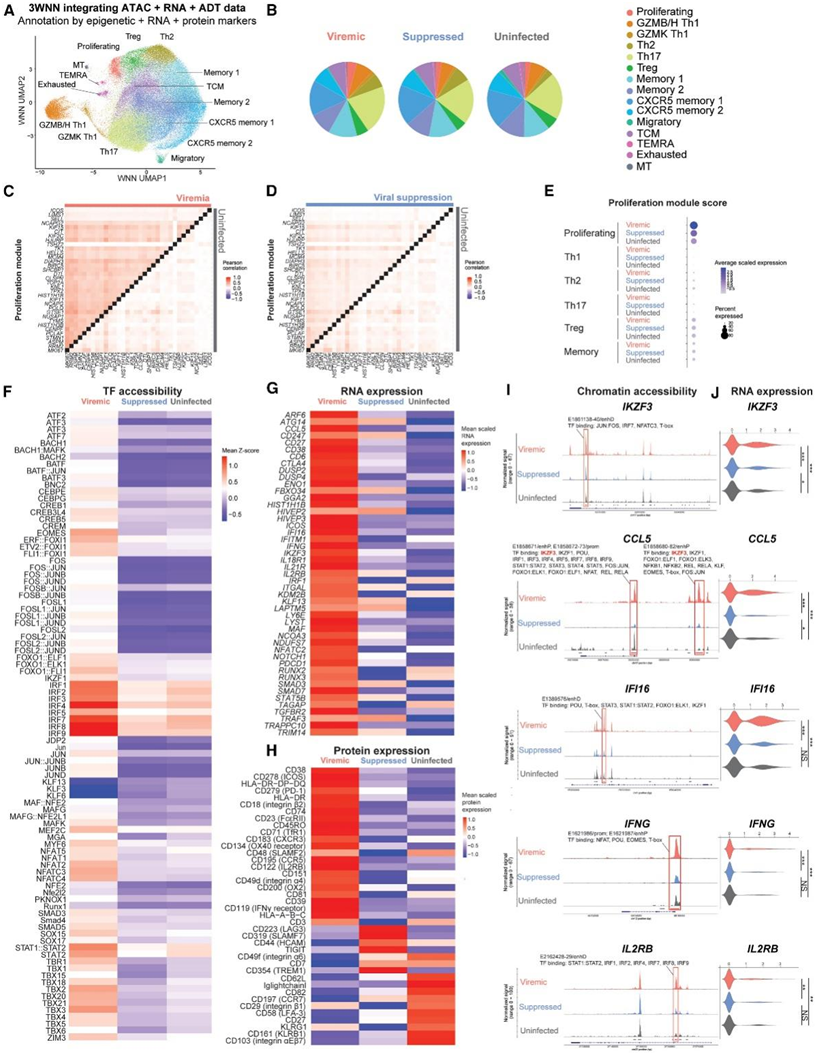
图1. 单细胞 DOGMA-seq 捕获 HIV-1 感染期间记忆 CD4+ T 细胞的异质表观遗传、转录和表面蛋白状态。
(a)UMAP可视化。(B) 饼图显示病毒血症、病毒抑制和未感染条件下的细胞亚群分布。(C-D) 对于 WGCNA 鉴定的增殖基因程序,成对基因表达 Pearson 相关系数的热图比较。(E) 每个细胞子集的增殖基因程序模块评分和感染条件。(F) 按条件分组的增殖聚类细胞中转录因子结合基序的染色质可及性。(G) 在增殖聚类细胞中差异表达基因。(H) 增殖聚类细胞中差异表达的表面蛋白。(I-J) 病毒血症中增殖细胞中染色质可及性和IKZF3、CCL5、IFI16、IFNG和IL2RB基因表达的增加。
02
单细胞DOGMA-seq鉴定了转录不活跃和转录活跃的HIV-1感染细胞的细胞状态
为了检测HIV-1感染细胞与未感染细胞之间的表观遗传、转录和蛋白质表达差异,他们将ATAC-seq 读数和RNA-seq 读数分别定位到HXB2 HIV-1参考基因组和自体病毒(图2),以鉴定HIV-1 DNA细胞和HIV-1 RNA细胞。值得注意的是,HIV-1 DNA细胞可以转录沉默或具有低于检测限的低HIV-1 RNA表达。在来自HIV-1感染、稳定整合的Jurkat细胞系的14780个细胞(每个细胞都有已知的HIV-1整合位点)中,他们在每个细胞阈值1读取量时识别出5220个HIV-1 DNA+细胞(灵敏度为35.32%),在每个细胞阈值2读取量时识别出4197个HIV-1 DNA+细胞(灵敏度为28.40%)。尽管这些HIV-1感染的Jurkat细胞具有活跃的HIV-1表达和高的HIV-1前病毒染色质可及性,但HIV-1 DNA的检出率仍然很低(28.4%)。
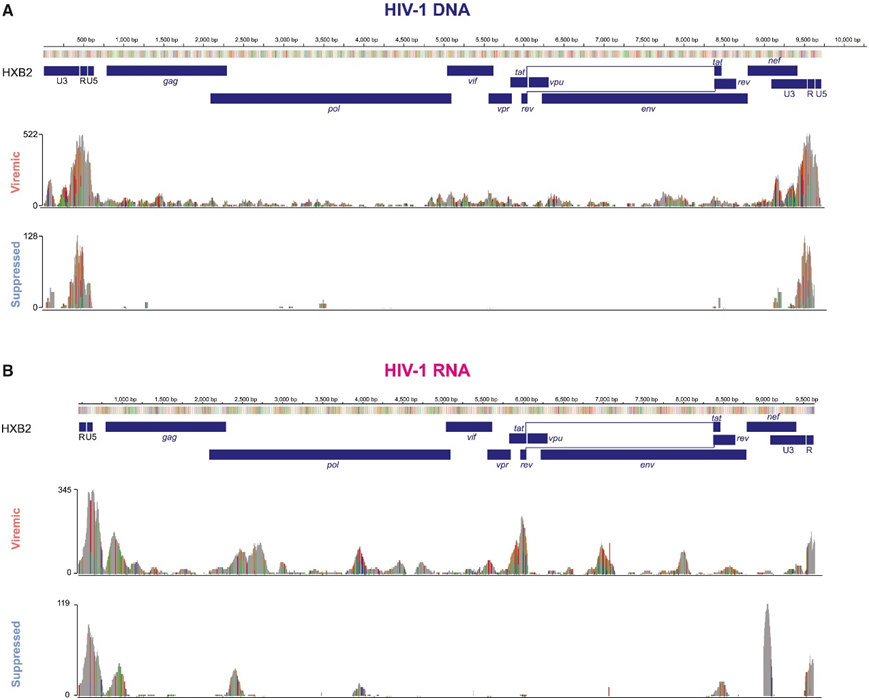
图2. DOGMA-seq 捕获的HIV-1 原病毒和病毒基因组图谱。
总共有233个HIV-1 DNA+ RNA -细胞和256个HIV-1 RNA+细胞出现病毒血症(图3A),19个HIV-1 DNA+细胞和14个HIV-1 RNA+细胞出现病毒抑制(图3B)。混合HIV-1 DNA细胞和HIV-1 RNA细胞分布在不同的记忆CD4+ T细胞亚群中,与HIV-1−细胞相比,在细胞类型组成上没有明显的偏差(图3C)。大多数HIV-1 DNA读取是在HIV-1长末端重复序列(LTR)启动子内捕获的(图2),这反映了HIV-1启动子的染色质可及性比蛋白质编码区高。
他们接下来检测了HIV -1感染细胞的表观遗传景观(图3D-F)。在病毒血症期间,类视黄酸相关(RAR)孤儿受体的结合基序(RORB和RORC,编码RORβ和RORγt)显著增加了HIV-1 DNA细胞的染色质可及性(图3D-F)。值得注意的是,由RORC编码的RORγt是驱动Th17分化的主转录因子。然后,他们比较了HIV-1 RNA+ 细胞与未感染细胞的转录因子结合潜能,发现AP-1(JUN, FOS和BATF)、CNC家族(NFE2L2, BACH1和BACH2)、 Maf和IRF转录因子的结合基序显著富集在HIV-1 RNA+细胞的染色质中(图3E-F)。同样,HIV-1 DNA+细胞也具有丰富的AP-1、CNC和Maf转录因子结合基序可及性,但程度低于HIV-1 RNA+细胞(图3F)。总的来说,AP-1、CNC、Maf和IRF家族转录因子的结合基序在HIV-1感染细胞中具有丰富的可及性,而RORβ和RORγt转录因子结合基序在HIV-1 DNA细胞中也具有丰富的可及性。
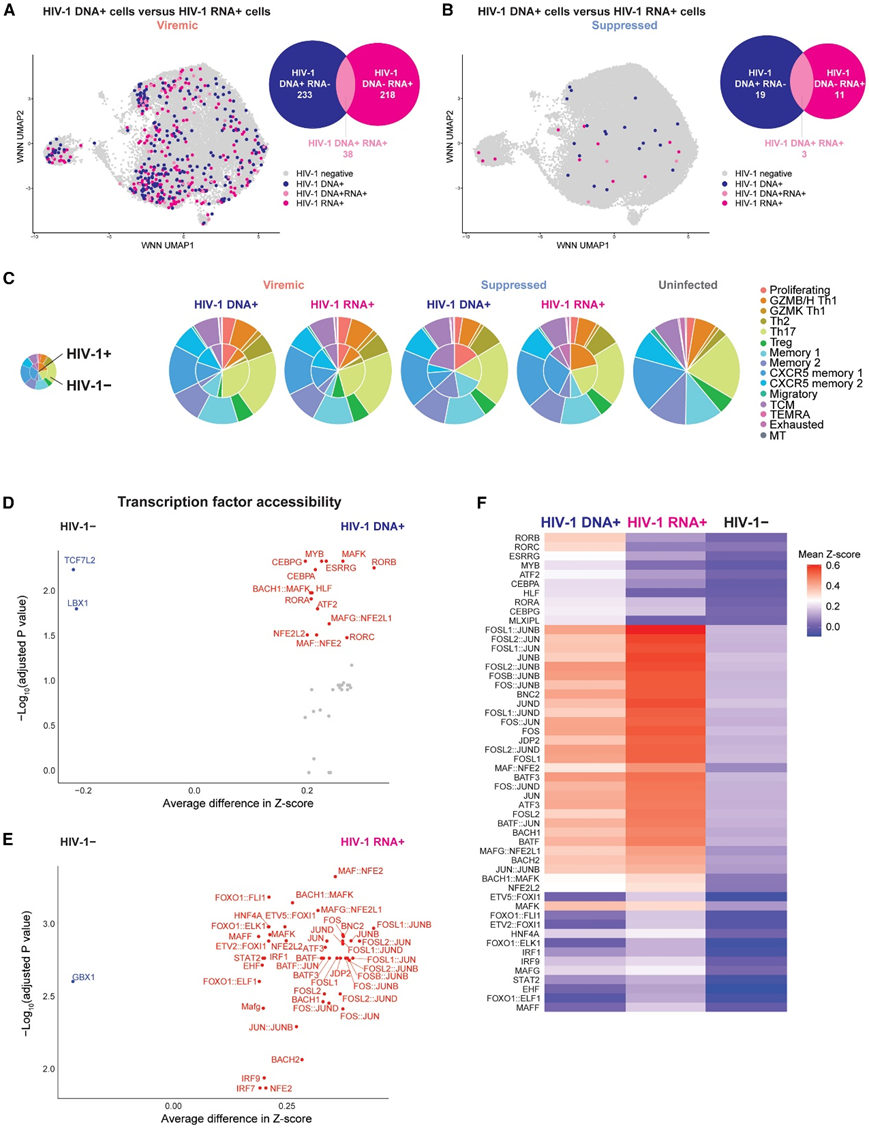
图3. 单细胞 DOGMA-seq 捕获转录不活跃的 HIV-1 感染细胞 (HIV-1 DNA + HIV-1 RNA−) 和转录活跃的 HIV-1 感染细胞 (HIV-1 RNA+) 及其各自的表观遗传景观。
(A-B) HIV-1 DNA+细胞和HIV-1 RNA+细胞分别通过ATAC-seq读数和RNA-seq读数定位到自体HIV-1和HXB2参考序列。(C)表示细胞子集分布的饼状图。(D-E)火山图表明转录因子结合基序增加了整体染色质可及性。(F)病毒血症中,转录因子结合基元在HIV-1 DNA+细胞和HIV-1 RNA+细胞中相对于彼此和HIV-1 -细胞的整体染色质可及性,在热图中表示为chromVAR偏倚校正偏差的组均值比较。
03
转录因子IKZF3与转录活性HIV-1感染细胞的存活和增殖有关
为了确定HIV-1感染细胞的转录谱,他们发现在病毒血症期间,与HIV-1-细胞相比,这些细胞中的基因显著上调(图4A-C),在HIV-1 DNA+ 细胞和HIV-1 RNA+细胞中发现了19个显著上调的基因。其中8个基因与T细胞分化密切相关:1个淋巴细胞分化基因(IKZF3,编码转录因子Aiolos)、3个I型IFN应答基因(STAT1、IFI44和IFI44L)、 2个细胞毒性T细胞效应基因(GZMA、SYTL2[介导裂解颗粒分泌])、1个T细胞活化基因(PDE4B介导IL-2产生)、1个T细胞迁移基因(ITGA4)(图4A-B)。值得注意的是,IKZF3在增殖细胞中也上调。
在HIV-1 DNA+细胞中,上调的基因参与了细胞毒性T细胞分化(GZMA、GZMK、KLRB1、CCL5和LTB)和T细胞迁移(ITGA4、T细胞归巢整合素α4)(图4A),这与之前发现的细胞毒性CD4+ T细胞和迁移细胞中HIV-1感染细胞富集一致。在HIV-1 RNA+细胞中,上调的基因包括细胞毒性T细胞分化(GZMA和CTSC[编码组织蛋白酶C])、T细胞迁移(ITGA4和ITGB7[整合素β7])、I型IFN应答(STAT1、STAT2和MX1)、T细胞分化转录因子(FOSL2、REL、MAF和STAT5B)和细胞生存基因(CFLAR,编码可阻止T细胞死亡的c-FLIP)(图4B)。在HIV-1 DNA+细胞中,细胞毒性标记物GZMA、GZMK、KLRB1和LTB在病毒血症和病毒抑制后均上调(图4D)。在HIV-1 RNA+细胞中,细胞毒性T细胞标记物(CTSC和SYTL2)、I型IFN应答基因(IFI44、STAT1和STAT2)、存活基因CFLAR和T细胞分化转录因子IKZF3在病毒血症和病毒抑制后均上调(图4E)。为了区分HIV-1感染细胞中的转录程序,他们使用WGCNA重新鉴定了共表达基因,发现了一个增殖程序,可以区分HIV-1 DNA+细胞、HIV-1 RNA+细胞和HIV-1-细胞之间的共表达基因(图4F-G)。这些共表达基因(如IKZF3和IL21[一种关键的Tfh细胞因子],BIRC5 [Survivin]和MKI67)参与细胞分裂和分化(图4H)。值得注意的是,BIRC5和IL-2是已知的促进HIV-1持久性的细胞因子。
他们接下来确定了不同的免疫通路,这些通路在病毒血症期间塑造了HIV-1感染的转录格局。使用基因集富集分析(GSEA),发现整合素通路在HIV-1 DNA+细胞中显著富集(图4I),而IL-6/JAK/STAT3信号通路在HIV-1 RNA+细胞中显著富集(图4J)。
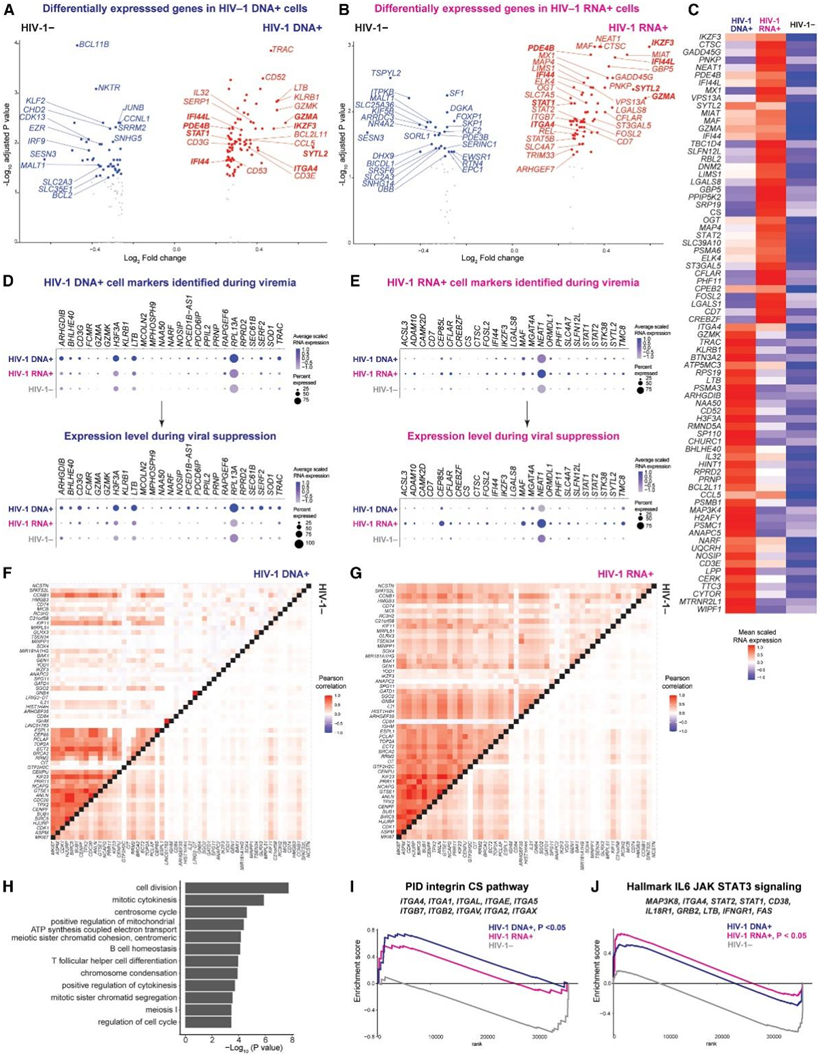
图4. 转录失活的 HIV-1 感染细胞上调 IKZF3、I 型 IFN、细胞毒性 T 细胞反应和归巢 RNA 表达。
(A-B) 火山图显示 HIV-1 DNA +细胞或 HIV-1 RNA +细胞与 HIV-1-细胞之间病毒血症中差异表达的基因。(C) 在 HIV-1 RNA +细胞和 HIV-1 DNA +细胞的病毒血症中高度表达的基因。(D-E) 在病毒血症中的 HIV-1 DNA +细胞中鉴定出显著上调的基因的点图。(F-G) HIV-1 RNA-1 +细胞与 HIV-1 -细胞之间的比较。(H)GO分析。(I-J) 基因表达排序,并使用 GSEA 鉴定了显著富集的通路。
04
转录失活的HIV-1感染细胞表达Th1/Th17、T细胞活化和迁移标志物
据报道,在HIV-1感染的细胞中,细胞标志物表达增加,如Th1极化(CXCR3和CCR5)、活化标志物(HLA-DR、CD2、ICOS和SLAM)、细胞毒性T细胞标志物(KLRB1和Granzyme B)、衰竭标志物(PD-1、CTLA-4和TIGIT)、Tfh极化(CXCR5和PD-1)和归巢标志物VLA-4(整合素α4β1)。此外,靶向HIV-1前病毒基因组测序鉴定了CD4 T细胞中含有完整HIV-1前病毒的蛋白特征,如PD-1、TIGIT、KLRG1、HVEM、CD49d(整合素α4)和CD95(Fas受体)。使用DOGMA-seq,他们确定了7种在HIV-1 DNA+细胞和HIV-1 RNA+细胞中均显著上调的表面蛋白,包括Th1极化标志物(CCR5、CXCR3和KLRB1)、T细胞迁移标志物(ITGAL)和T细胞活化标志物(CD38、LFA-2和SLAM)(图5A-C),这与之前的研究一致。在HIV-1 RNA+细胞中,共有18种蛋白发生上调,包括Th1和Th1(Th1/Th17)的标记物极化(CCR5、CCR6、CXCR3、KLRB1和IFNγ受体CD119)、迁移(整合素α4、整合素αL和HCAM)和T细胞活化(图5B)。值得注意的是,CD81降解SAM结构域和HD结构域1 (SAMHD1)并增强HIV-1的逆转录。病毒血症的HIV-1 RNA+细胞中显著上调的7个蛋白标记在病毒抑制的HIV-1 RNA+细胞中也发生上调(图5D-E)。在病毒血症和病毒抑制中,HIV-1 DNA+ 细胞和HIV-1 RNA+ 细胞中SLAM和CCR5蛋白表达上调。总体而言,他们发现在HIV-1 DNA+细胞中,Th1极化和T细胞激活的表面蛋白标记物与HIV-1- 细胞相比上调,而在HIV-1 RNA+细胞中,Th1和Th1/Th17极化、迁移和T细胞激活的表面蛋白标记物与HIV-1- 细胞相比上调(图5F-H)。
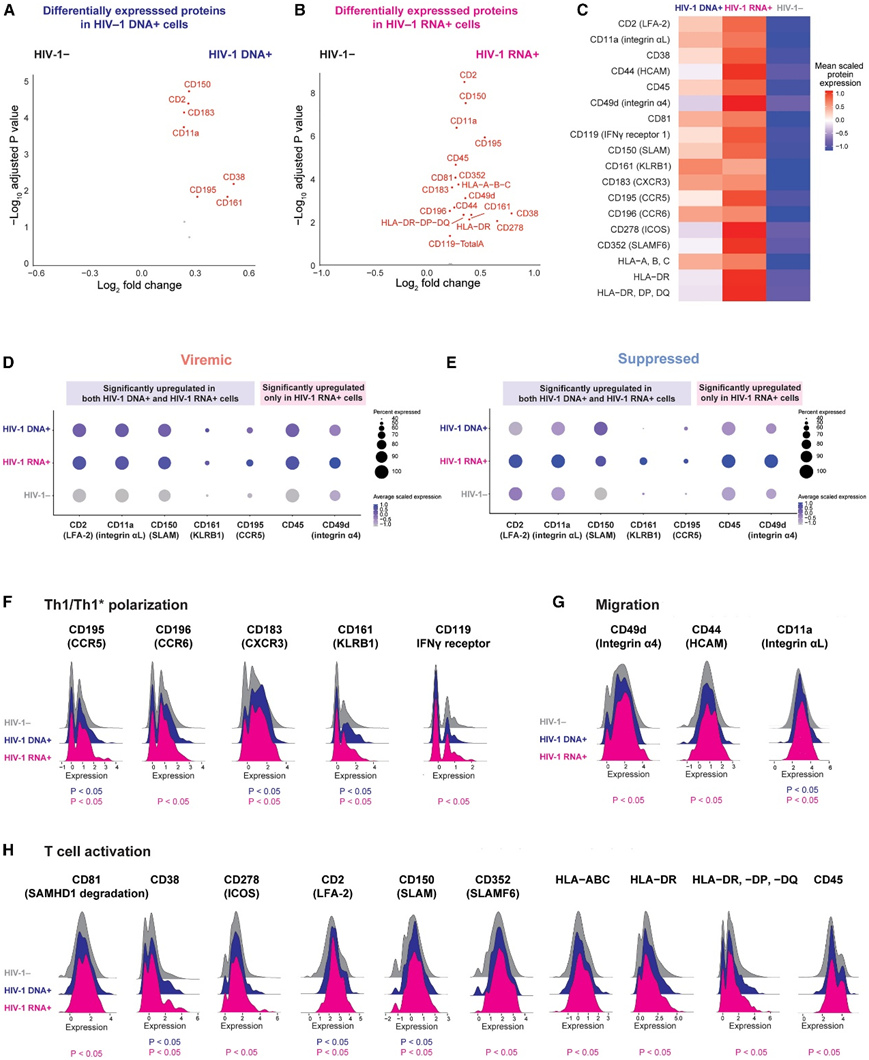
图5. 转录失活的 HIV-1 感染细胞上调 Th1/Th17、T 细胞激活和归巢蛋白表达。
(A-B) 火山图显示 HIV-1 DNA+ 细胞或 HIV-1 RNA+ 细胞与 HIV-1- 细胞之间病毒血症表面蛋白的差异表达。(C) HIV-1 DNA+ 细胞与 HIV-1 RNA+细胞与 HIV-1- 细胞中病毒血症中高度表达的表面蛋白。(D-E) 在病毒血症中的 HIV-1 DNA +细胞和 HIV-1 RNA + 细胞中鉴定出显著上调的蛋白质在病毒抑制中的 HIV-1 感染细胞中也上调。(F-H) 岭图显示(A) 和 (B) 中确定的显著高表达的表面蛋白的 DSB。
05
异质 HIV-1 感染细胞包含四种不同的细胞状态:IRF、细胞毒性、AP-1 和细胞死亡
他们鉴定了HIV-1+ 记忆CD4+ T细胞的表观遗传、转录和蛋白质特征。虽然参与者之间存在差异,但这些项目并不是由特定的参与者驱动的。他们收集了所有鉴定出的HIV-1感染细胞,通过它们的转录谱来确定不同的细胞群(图6A)。HIV-1细胞的聚集不是由感染条件、HIV-1 RNA表达的批量效应造成的(图6B),也不是由峰值病毒载量不同的研究参与者造成的(图6C)。每个聚类具有不同的转录谱(图6D),明显富集的转录因子可及性(图6E)和不同的表面蛋白表达谱(图6E)。他们在6名HIV-1研究参与者中确定了4个表型不同的HIV-1细胞群。
他们根据 HIV-1 感染细胞的不同细胞特征(转录因子可及性、转录程序和表面蛋白表达)对每个 HIV-1 感染细胞聚类进行了注释:(1) IRF 聚类增加了 IRF 和 STAT1/STAT2 转录因子可及性(图 6E)、“激活/增殖”基因共表达程序(ICOS、BIRC5、IL21和MKI67的共表达)(图6H)和激活表面蛋白(HLA-DR、ICOS和CD38)(图6F) ; (2) 细胞毒性聚类增加了 Eomes 转录因子的可及性(图 6E),细胞毒性效应 T 细胞基因标记上调(GZMA、GZMH、NKG7、CCL5、IFNG和ZEB2)(图 6D)、“细胞毒性”基因共表达程序(GZMB、GZMH、NKG7、IFNG和ZEB2)(图6I)和细胞毒性表面蛋白(GPR56和KLRG1)(图6F);(3)AP-1聚类增加了AP-1转录因子的可及性(图6E),增加了JUN和FOS RNA表达(图6D),“TNF”基因共表达程序(JUN 、FOS、TNFAIP3和NFKBIA)(图6J)和迁移表面蛋白(HCAM、PSGL-1 和整合素 α6 和 β1)(图6F);(4) MT(线粒体基因)聚类的线粒体基因表达上调,可能反映了细胞死亡(图 6D)。
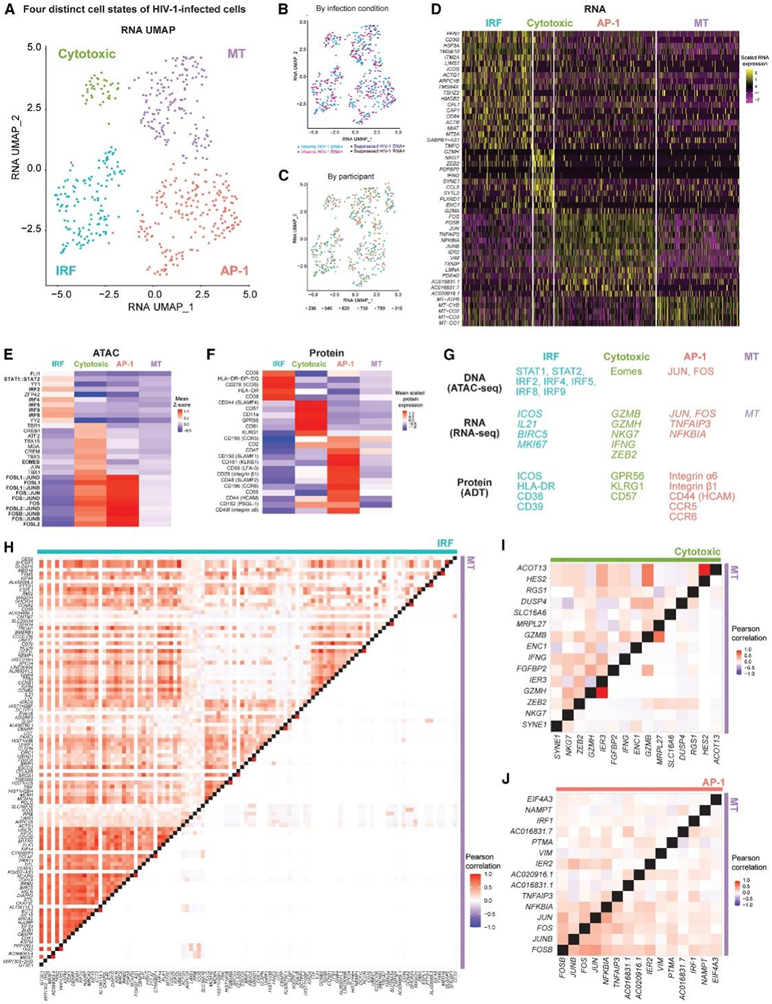
图6. 异质 HIV-1 感染的 T 细胞库包含四种不同的细胞状态:IRF、细胞毒性、AP-1 和细胞死亡。
(A–C) UMAP可视化。(D)每个细胞聚类中大多数高度差异表达基因。(E) 转录因子结合基序的差异可及性。(F) 每个细胞组高表达的蛋白质。(G) 在四个表型不同的 HIV-1+细胞聚类中,转录因子具有丰富的结合基序可及性并上调基因和蛋白质。(H–J) WGCNA 确定的基因程序的成对基因表达 Pearson 相关系数的热图比较。
06
转录不活跃和活跃的HIV-1感染细胞都上调IKZF3的表达
IKZF3促进了HIV-1感染细胞的增殖和存活,尤其是在急性HIV-1感染期间。AP-1、IRF7、NFATC3和Tbox转录因子可能与IKZF3中的可及CRE结合(图1I),从而调节其表达(图1J)。此外,IKZF3在HIV-1 RNA细胞中与细胞增殖和存活基因如IL21、MKI67和BIRC5共表达(图4G)。接下来,他们比较了IKZF3基因在感染条件下的可及性和基因表达(图7A),以及在记忆性CD4+ T细胞亚群中HIV-1+和HIV-1-细胞之间的表达(图7B)。增殖细胞在病毒血症期间,IKZF3基因可及性和RNA表达量增加(图7A)。与HIV-1+和HIV-1- 增殖细胞相比,IKZF3基因在HIV-1 DNA+ 细胞中的可及性明显更高(图7B-7D), IKZF3基因在HIV-1 DNA+ 细胞和HIV-1 RNA+ 细胞中的表达均显著更高(图7B-E)。总的来说,IKZF3在病毒血症下的增殖细胞,特别是HIV-1感染的细胞中增加了基因的可及性和基因表达。
为了验证Aiolos(由IKZF3编码)蛋白表达是否在急性HIV-1感染过程中增加,他们用先前报道的具有复制能力的NL4-3参考株和3株R5型临床分离株感染来自未感染个体的活化原代CD4+ T细胞(包括总CD4+ T细胞和记忆CD4+ T细胞),并使用流式细胞分析检测Aiolos蛋白表达。通过检测HIV-1 p24表达和CD4+下调来检测HIV-1感染。他们发现,通过平均荧光强度(MFI)测量,HIV-1感染的细胞在总CD4+ T细胞和记忆CD4+ T细胞中都具有更高的Aiolos蛋白表达(图7F-G)。而且Aiolos细胞具有更高的Ki-67(由MKI67编码)蛋白表达(图7I-J),表明Aiolos的表达可能促进HIV-1感染细胞的增殖。总体而言,单细胞DOGMA-seq和体外验证结果均显示,IKZF3在HIV-1感染细胞中的表达增加,并与增殖标志物Ki-67的表达相关。

图7. 转录不活跃和活跃的 HIV-1 感染细胞均上调IKZF3表达。
(A-B) 点图显示六个主要细胞亚群中IKZF3的染色质可及性(左)和 RNA 表达(右)。(C) IKZF3处的染色质可及性。(D-E) 小提琴图显示IKZF3染色质可及性和 RNA 表达的差异。(F) 代表性流式细胞图,显示 HIV-1 感染细胞与未感染细胞中 Aiolos 蛋白的表达。(G-H) HIV-1 感染和未感染细胞中 Aiolos 蛋白的表达。(I-J) Ki-67在Aiolos+ HIV-1+细胞与Aiolos−HIV-1+细胞中总CD4+ T细胞和记忆CD4+ T细胞中的表达比例。
+ + + + + + + + + + +
结 论
本项研究使用单细胞DOGMA-seq同时捕获来自6名HIV-1患者在病毒血症期间和抑制性抗逆转录病毒治疗后的记忆CD4+ T细胞中的转录因子可及性、转录组、表面蛋白、HIV-1 DNA和HIV-1 RNA。发现潜伏 HIV-1 感染细胞 (RORC) 和转录活性 HIV-1 感染细胞的转录因子可及性增加。增殖程序(IKZF3、 IL21、 BIRC5和MKI67共表达)促进转录活性 HIV-1 感染细胞的存活。潜伏和转录活跃的 HIV-1 感染细胞的IKZF3 (Aiolos) 表达均增加。不同的表观遗传程序驱动了 HIV-1 感染细胞的异质细胞状态,IRF:激活、Eomes:细胞毒性效应器分化、AP-1:迁移和细胞死亡。本项研究研究揭示了潜伏和转录活跃的 HIV-1 感染细胞的单细胞表观遗传、转录和蛋白质状态以及促进 HIV-1 持久性的细胞程序。
+ + + + +



 English
English


