文献解读|Adv Sci(15.1):大规模蛋白质组分析鉴定与疑似神经梅毒诊断相关的生物标志物
✦ +
+
论文ID
原名:Large-Scale Proteome Profiling Identifies Biomarkers Associated with Suspected Neurosyphilis Diagnosis
译名:大规模蛋白质组分析鉴定与疑似神经梅毒诊断相关的生物标志物
期刊:Advance Science
影响因子:15.1
发表时间:2024.02.21
DOI号:10.1002/advs.202307744
背 景
神经梅毒(NS)是由梅毒螺旋体(T. pallidum)引起的中枢神经系统(CNS)感染。 NS 可发生在梅毒的任何阶段,并表现为广泛的临床症状。 NS 通常可称为“伟大的模仿者”,由于缺乏标准的诊断测试,很容易忽视或误诊,可能导致严重且不可逆转的器官功能障碍。
实验设计
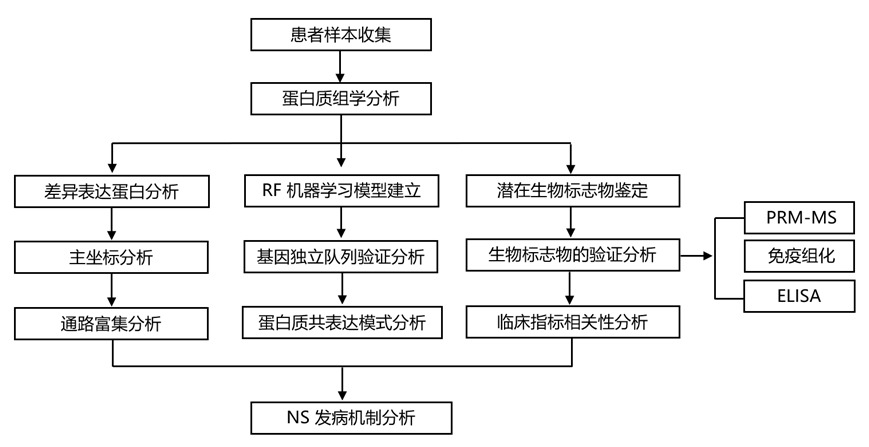
结 果
01
NS 患者脑脊液的蛋白质组特征
研究者团队使用数据独立采集(DIA)策略收集了来自 15 名 NS 患者、7 名 治疗后 NS (PTNS)患者、9 名无梅毒的非传染性脑部疾病(NIBD)_患者、7 名与梅毒无关的传染性脑部疾病(IBD)患者和 25 名非 NS (NNS)患者的 63 份脑脊液(CSF)样本,用于蛋白质组鉴定(图 1A)。总共鉴定了 1800 个蛋白质,其中 1749 个来自 NS,1638 个来自 PTNS,1638 个来自 NIBD,1549 个来自 IBD,1719 个来自 NNS 患者(图S1A)。结果显示属于 NS 和 NNS 组(队列 1)的样品中蛋白质丰度具有良好的相关性(图 S1B)。然而,NS 组和 NNS 组之间的相关性较差。NS组和NNS组之间相关性较差。采用主坐标分析(PCoA)量化NS组和NNS组的差异(图S1C)。CSF-RPR(RPRhigh;RPR滴度> 1:4),csf -白细胞(WBCs;WBS high;WBC > 8×106细胞L−1),csf蛋白(PRO;PROhigh;NS患者脑脊液中PRO > 0.45 g L−1)水平高于NS患者脑脊液(图S1D)。
层次聚类分析 (HCA) 和火山图分析揭示了 NS 和 NNS 组之间差异表达的 358 个蛋白质(图 1B)。与 NNS 组相比,NS 组中的 109 种上调蛋白主要富集于免疫反应,包括先天免疫和体液免疫,以及各种炎症反应(图 1C )。他们还发现 NS 组中与细菌防御反应、凝血和吞噬作用相关的蛋白质表达上调,这表明脑脊液中各种伤口愈合相关蛋白质的潜在贡献。NS患者中表达下调的蛋白质主要与神经系统发育相关,包括轴突发生、突触组织和神经元发育(图 1C)。
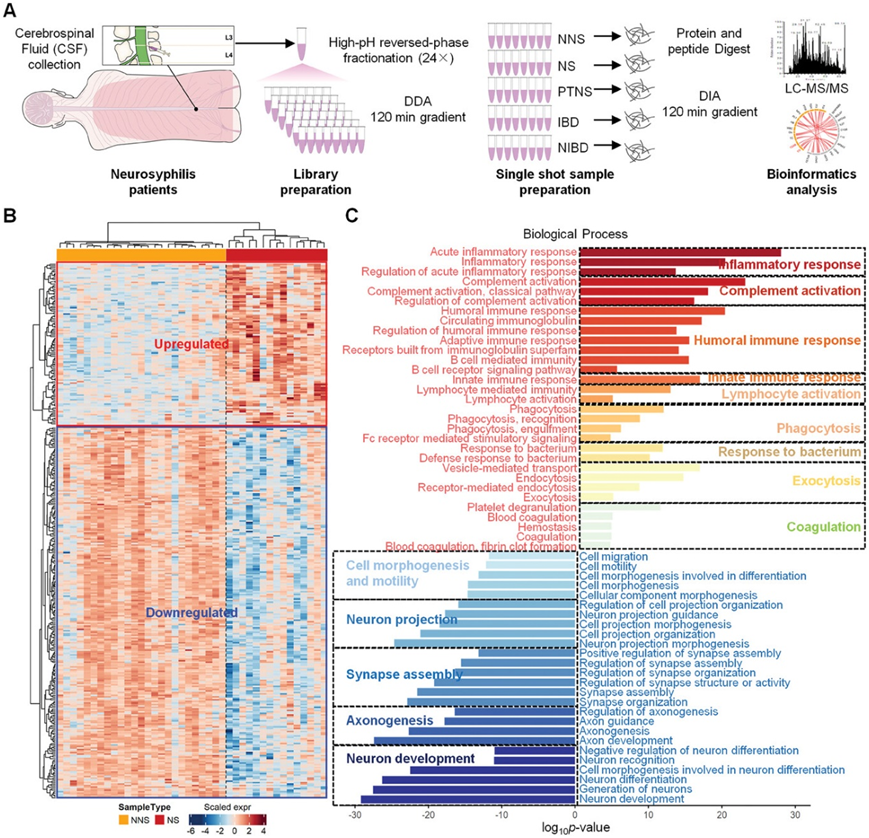
图1. NS 患者脑脊液的蛋白质组学特征。
(a) CSF 样本的定量蛋白质组和生物信息学分析的实验工作流程示意图。(b) 热图显示 NNS和 NS样本(队列 1)之间脑脊液中差异表达的蛋白质。(c) NS 与 NNS 患者脑脊液中差异表达蛋白的生物过程分析。
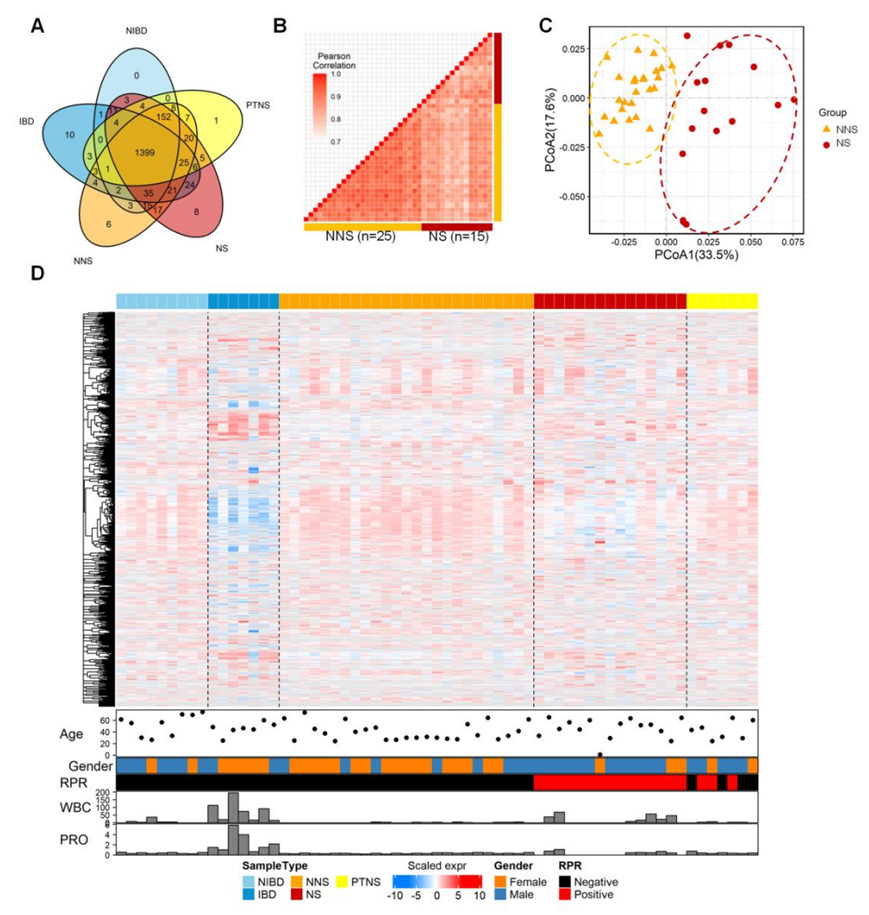
图S1. 基于DIA技术的定量蛋白质组学分析NNS、NS、PTNS、IBD和NIBD患者脑脊液样本的蛋白质组学特征。
(a) NNS、NS、PTNS、IBD和NIBD样本脑脊液中蛋白的维恩图。(b) 整体关联性的热图。(c) NS和NNS样品蛋白质组谱的PCoA分析。(d) 蛋白质组学一致聚类分析。
02
区分 NS 和 NNS 患者的潜在生物标志物
一般来说,NS 组和 NNS 组之间差异表达的蛋白质可以作为独特的标记。为了确定这些特征蛋白在识别 NS 中的重要性,基于 25 个 NS 和 15 个 NNS 样本的差异蛋白质表达数据构建了随机森林 (RF) 机器学习模型(队列 1)(图 2A)。他们还从平行反应监测质谱 (PRM-MS) 数据集中重新生成了这些基于 RF 的分类器,以验证候选生物标志物并对 NS 患者进行分类(队列 2)(图 2A)。该模型的准确率达到了 98.11%(图 2B)。在训练队列中观察到曲线下面积 (ROC) 为 0.936(图 2C)。然后,他们在 29 名疑似NS (SNS) 患者组成的独立队列中进一步测试了该模型(队列 3)(图 2A)。
此外,他们收集了 7 名 PTNS 患者样本并鉴定了 1638 种蛋白质(图 2D)。结果显示,232种蛋白质在NS组和NNS组之间存在差异表达,并且在治疗后PTNS组的CSF中呈负表达(图 2E);治疗后,NS组上调蛋白和下调蛋白分别减少和增加(图 2F)。这些发现表明,这232个差异表达蛋白是决定NS患者治疗效果和治疗后恢复程度的潜在生物标志物。
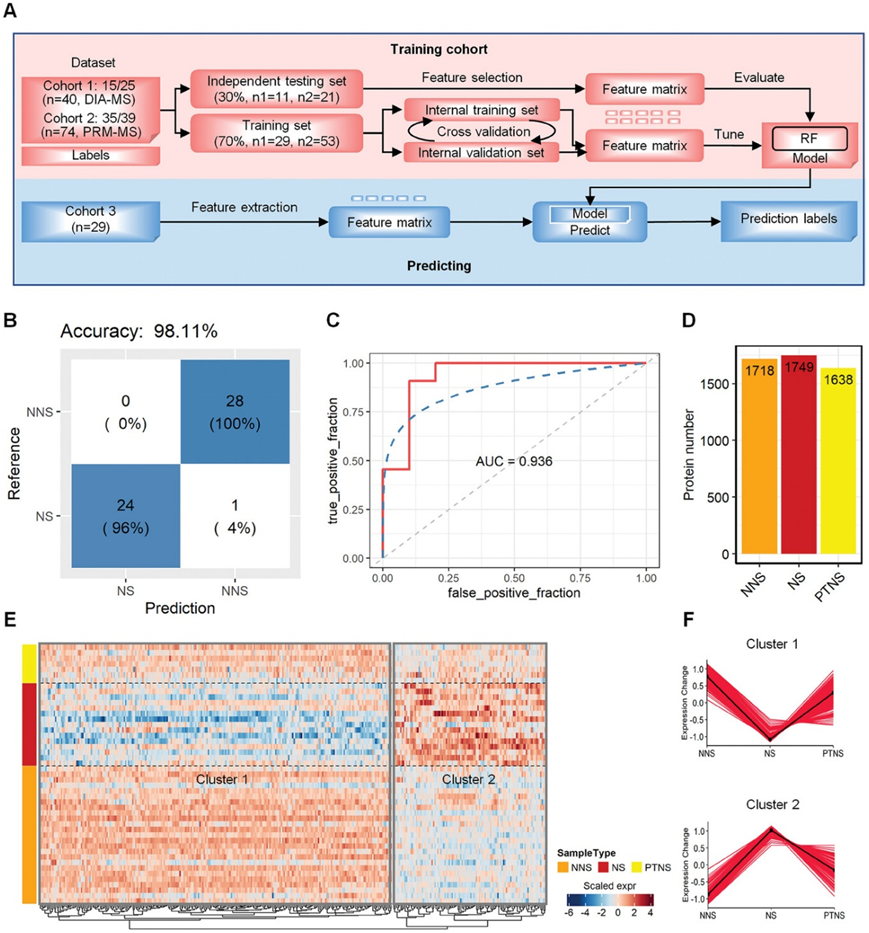
图2. 使用机器学习方法从 NNS 患者中识别出用于诊断 NS 患者的潜在生物标志物。
(a) 基于随机森林 (RF) 的机器学习策略示意图。(b) 混淆矩阵显示模型对 NS 个体进行分类的性能。(c) 用于对 NS 个体进行分类的基于 RF 模型的接收者操作特性曲线。(d) 从 NNS、NS 和 PTNS 样本的 CSF 中鉴定出的蛋白质的分布。(e) 热图显示 PTNS 样本中 NNS 和 NS 样本中差异表达蛋白质。(f) 显示了 Cluster 1 和 Cluster 2 模块中蛋白质的共表达模式。
03
区分 NS 和其他脑部疾病的潜在生物标志物
为了进一步研究 NS 组和 NNS 组之间差异表达的 358 个蛋白质是否能够区分 NS 和其他脑部疾病,他们收集了 7 名 IBD 患者(队列 5)和 9 名 NIBD 患者(队列 6)的 CSF 样本进行定量蛋白质组学分析(图3A,图S3A)。PCoA 分析显示,大多数 NS 样本与 IBD 和 NIBD 组的样本有区别;然而,小的重叠表明 NS 患者的蛋白质特征与 IBD 或 NIBD 患者的蛋白质特征部分相似(图 S3B)。 HCA还显示NS和IBD患者的蛋白表达模式更加相似(图 3B)。急性炎症相关蛋白,如 CA2、IGHM、CRP 和 CD27 共表达(图 3C),表明 NS 或其他感染性脑疾病患者的 CSF 中存在强急性反应蛋白,这与临床发现相符。相反,NS 患者与 NIBD 患者的蛋白质共表达相似性低于与 IBD 患者的相似性(图 3C-D)。通过对 NS 和 NNS 组之间差异表达的 358 个蛋白质进行综合分析,他们鉴定了 127 个将 NS 与 IBD 区分开的蛋白质(图 S3C)和 138 个将 NS 与 NIBD 区分开的蛋白质(图 S3D)。
最后,他们鉴定了 45 种可以区分 NS 与 IBD 和 NIBD 的蛋白质(图 3E)。例如,检测到与轴突连接形成和神经元轴突引导有关的蛋白质。这些包括免疫球蛋白超家族的成员(CNTN1、CNTN2 和 NCAM2)、L1 家族免疫球蛋白细胞粘附分子 (NFASC)、信号蛋白家族成员(SEMA4A 和 SEMA7A)、丝氨酸蛋白酶抑制剂超家族成员 (SERPINI1) 和细胞粘附蛋白 ( SPON1)。分析还揭示了与髓鞘形成相关的蛋白质的存在,包括髓鞘糖蛋白(MAG、MOG 和 OMG)、少突胶质细胞功能相关蛋白、前列腺素 D2 合酶 (PTGDS)、Z1 型蛋白酪氨酸磷酸酶受体 (PTPRZ1) 和网状蛋白 4 受体(PTN4R),在发育中的神经系统中(图3E-F)。这些生物标志物可能有助于区分 NS 和与大脑功能各个方面有关的其他脑部疾病。
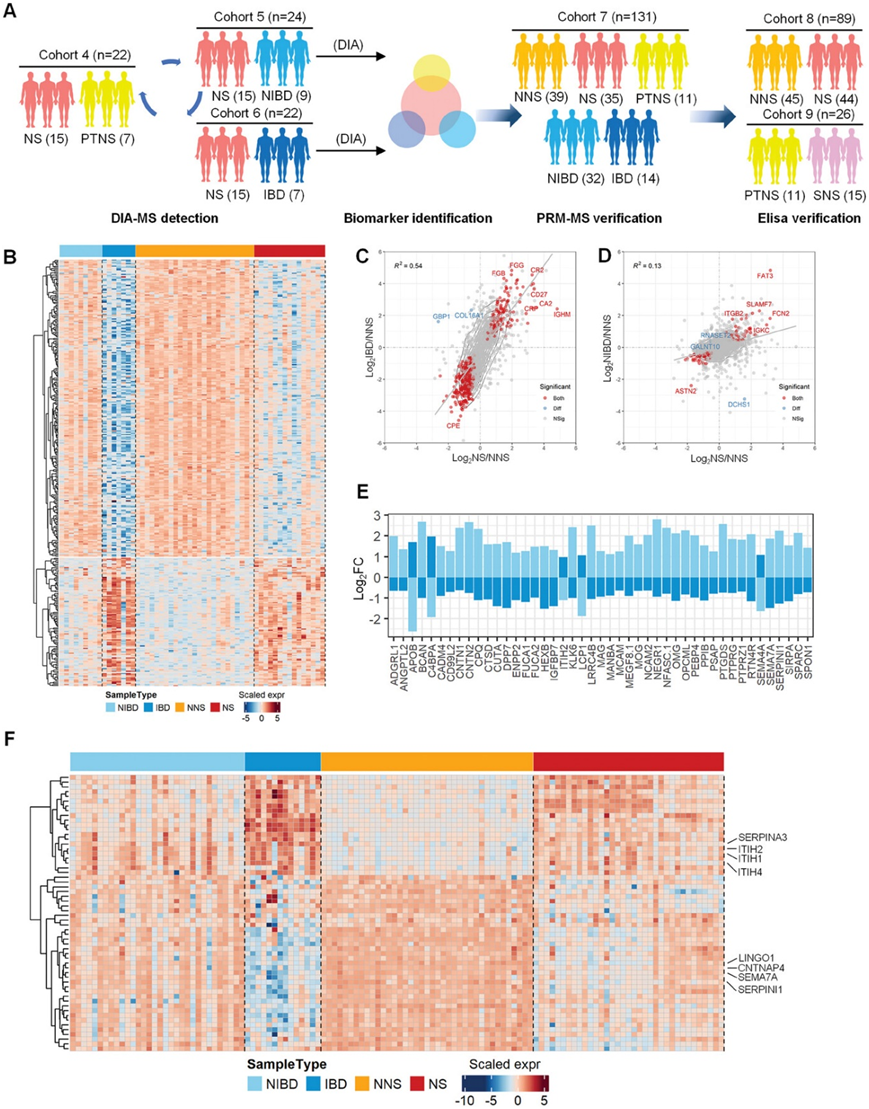
图3. 探索和鉴定用于诊断其他脑部疾病的 NS 的潜在生物标志物。
(a) 通过 DIA-MS、PRM-MS 和 ELISA 测量 NS、IBD、NIBD 和 PTNS 患者的 CSF 样本的工作流程。 (b) 热图显示 IBD 和 NIBD 样本中 CSF 中 NNS 和 NS 之间差异表达蛋白。(c) NS 与 NNS(x轴)和 IBD 与 NNS(y轴)的差异倍数散点图。(d) NS 与 NNS(x轴)和 NIBD 与 NS(y轴)的差异倍数散点图。 (e) 直方图显示NS 与 IBD(浅蓝色)组和 NS 与 NIBD(深蓝色)组中差异表达的蛋白质。(f) 热图显示NNS、NS、IBD和 NIBD用于 PRM-MS 验证的样品。
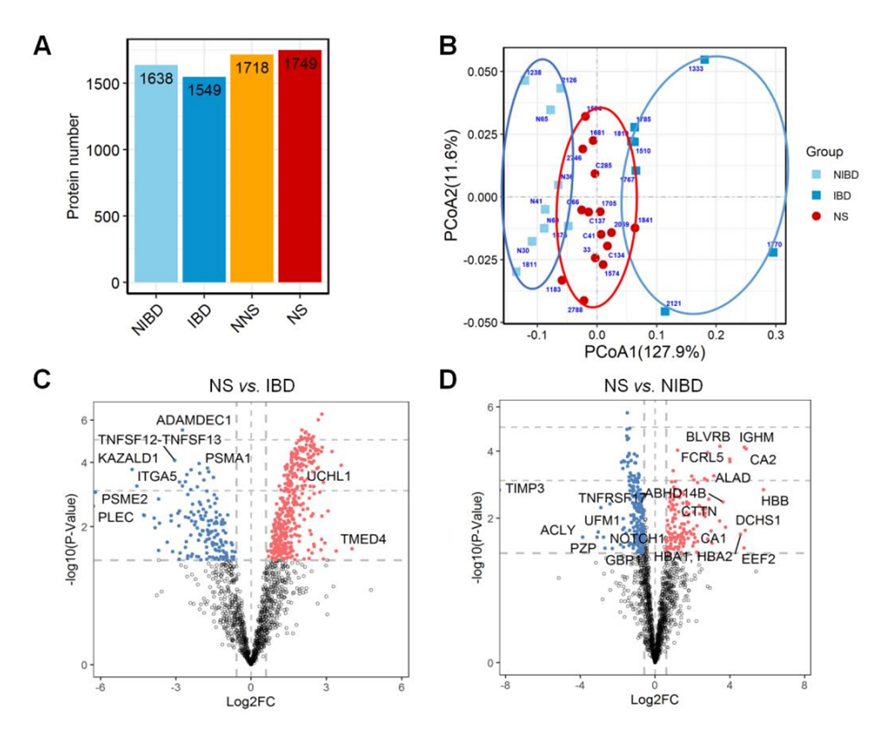
图S3. NNS、NS、IBD和NIBD患者脑脊液样本的蛋白质组学特征。
(a) NNS, NS, IBD和NIBD样本脑脊液中鉴定的蛋白质数量分布。(b) 蛋白质组的PCoA分析。(c-d) NS组与IBD 组或NIBD组脑脊液蛋白丰度比较。
04
通过PRM-MS和ELISA验证生物标志物
为了进一步识别可靠的 NS 生物标志物,他们根据机器学习模型得分、差异倍数以及NS 组和其他组之间的 统计p值构建了一个评分表(图S4A)。根据该评分表,对鉴定出的前 80 种蛋白质进行了 PRM-MS 验证。接下来,他们收集了131个CSF样本,包括35个NS、14个IBD、32个NIBD、11个PTNS样本和39个对照组样本(NNS),作为一个独立队列(队列7)(图 3A),PRM 将 80 种蛋白质中的 58 种鉴定为潜在的生物标志物(图 S4B),其表达模式与队列 1、5 和 6 的先前发现一致(图 1B、图 3B)。大多数上调的NS蛋白与诊断NS的临床指标呈正相关(图 4A),例如间α-胰蛋白酶抑制剂家族成员(ITIH1、ITIH2和ITIH4),它们充当透明质酸的载体;丝氨酸蛋白酶抑制剂家族A成员3(SERPINA3),可抑制中性粒细胞组织蛋白酶G。相反,NS队列中大多数下调的蛋白质与临床指标呈负相关(图 4B)。该组包括少突胶质细胞分化和轴突髓鞘形成的重要负调节因子 (LINGO1)、调节轴突生长的丝氨酸蛋白酶抑制剂 (SERPINI1)、参与多巴胺能突触传递的突触前蛋白 (CNTNAP4) 和 SEMA7A。
此外,PRM结果和DIA结果显示,PTNS组中有21种上调蛋白和37种下调蛋白,例如SEMA7A、SERPINA3、ITIH4、LINGO1、SERPINI1、CNTNAP4、ITIH1、ITIH2等(图 4C-D,图S5B-C)。病理学显示,与正常对照(NC)组相比,NS脑组织中LINGO1、SERPINI1、CNTNAP4、SEMA7A表达水平降低,ITIH1、ITIH4、SERPINA3表达水平升高(图4E),这表明这些基因在区分NS组和NNS组方面具有潜在的作用。值得注意的是,SEMA7A是一种膜锚定蛋白,已知可促进神经轴突生长并调节神经系统中突触的形成和消除。该蛋白广泛表达于多个脑区,包括海马、下丘脑-垂体系统、中间脑多巴胺系统和脊髓。因此,NS患者脑脊液或脑组织中SEMA7A不足可能提示神经元损伤。ITIH4是α间抑制蛋白(IAIP)家族的一员,是神经炎症的指征,有助于评估脊髓损伤的程度。该蛋白受IL-6调节,在炎症反应的急性期高度表达,不依赖于促炎因子TNF-α。SERPINA3 已确定为导致血脑屏障 (BBB) 功能障碍的候选星形胶质细胞因子。在源自人类诱导性多能干细胞的 BBB 共培养模型中,发现 SERPINA3 表达在 TNF 激活后增加,导致星形胶质细胞转变为炎症反应状态,从而导致 BBB 功能障碍。ITIH4 和 SERPINA3 的表达水平与中枢神经系统炎症相关,并可预测 NS 患者脑炎的严重程度。
为了验证这些潜在生物标志物的可靠性,他们扩大了人群并收集了额外的 89 个 CSF 样本(44 个 NS 和 45 个 NNS)用于 ELISA 验证(队列 8)(图 3A)。与 NNS 组相比,NS 组中 ITIH4 和 SERPINA3 的表达显著上调,SEMA7A 的表达显著下调(图 4F)。他们还检测了 PTNS 患者的 CSF,发现 ITIH4 和 SERPINA3 的表达水平下降(队列 9)(图 3A,图 S5A)。这些结果为这三种蛋白作为 NS 诊断标记物的潜力提供了额外的支持。
接下来,通过分析来自不符合CDC诊断标准的SNS患者的29份CSF样本来评估本项研究的评分模型在NS临床诊断中的表现(队列3)(图2A)。评分系统将19名患者划分为疑似NS患者,10名患者划分为疑似NS患者。预测NS评分高的患者表现出记忆力下降(S-24)、意识障碍(S-4、S-8)和双眼视力下降(S-8、S-10)等症状,这一现象证实了这种分类(图4G)。此外,他们选择了其中一个生物标志物ITIH4进行验证(队列9)(图3A),发现模型预测结果与ELISA结果具有很强的一致性(图4H)。S-8和S-10样品的脑脊液中ITIH4表达上调。该评分模型和确定的生物标志物表明,NS评分高的患者应该接受密切的临床监测。
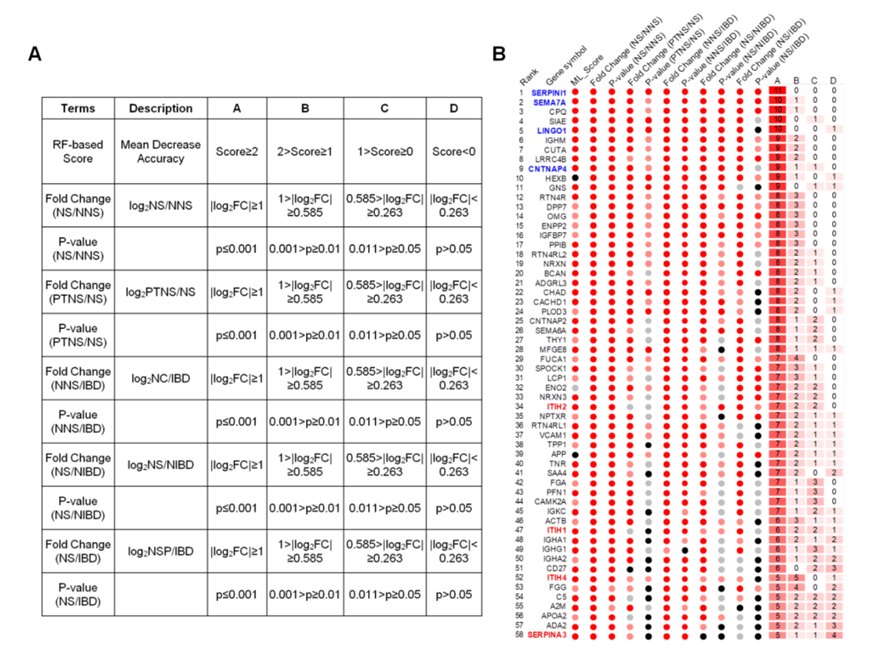
图S4. 基于蛋白质组学特征和机器学习模型评分的潜在生物标志物评分表的构建。
(A) PRM-MS验证蛋白选择评分表,包括11个详细评分项。(B) 经PRM-MS验证的58个蛋白的可视化。
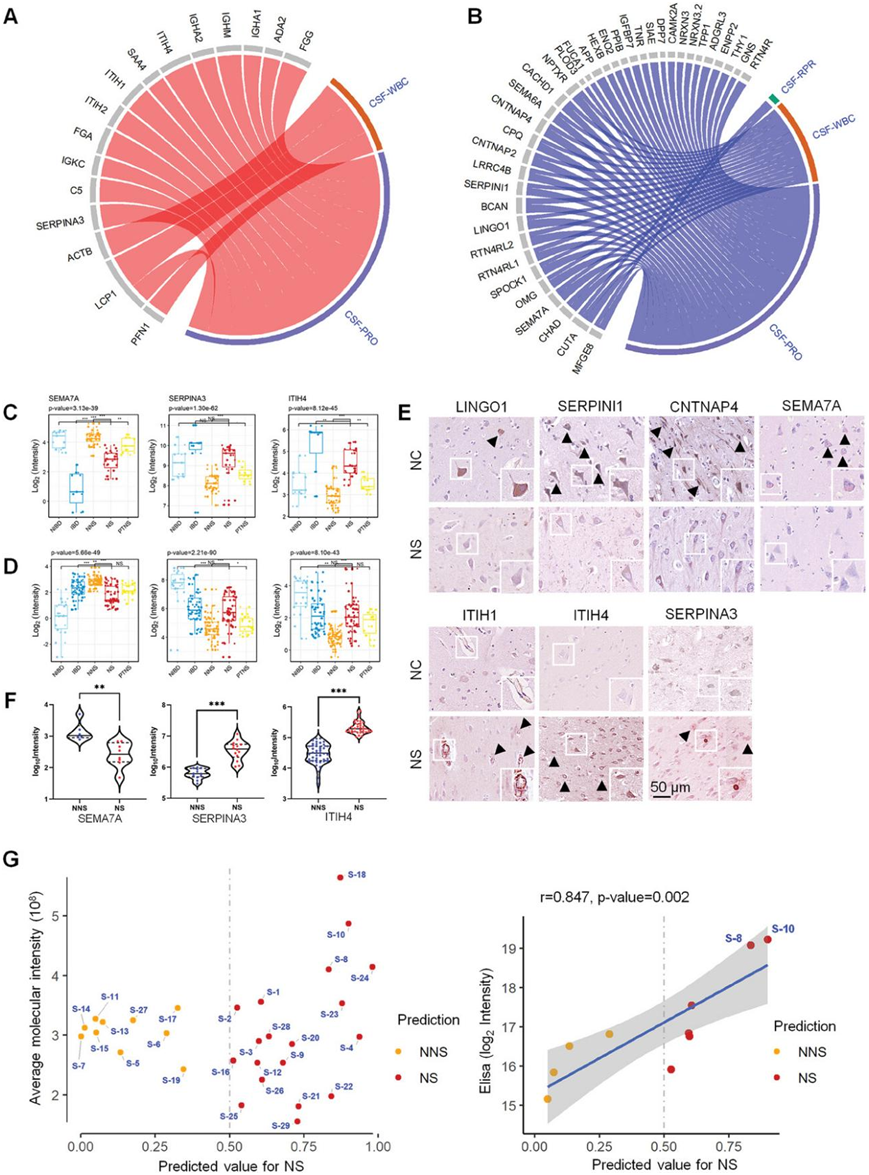
图4. 验证用于诊断 NS 的潜在生物标志物。
(a-b) 上调和下调蛋白质与临床指标(CSF-PRP、CSF-WBC 和 CSF-PRP)的相关性。(c-d) 通过 DIA 鉴定和 PRM-MS 验证检测 NNS、NS、PTNS、IBD 和 NIBD 组中 SEMA7A、SERPINA3 和 ITIH4 的蛋白表达水平。(e) NNS 组和正常对照 (NC) 组脑组织中 LINGO1、SERPINI1、CNTNAP4、SEMA7A、ITIH1、ITIH4 和 SERPINA3 的免疫组织化学染色。(f) 对 NNS和 NS组CSF 中的 SEMA7A、SERPINA3 和 ITIH4 表达进行 ELISA 分析。(g) RF 模型在 29 名疑似 NS 患者的测试队列(队列 3)中的表现。
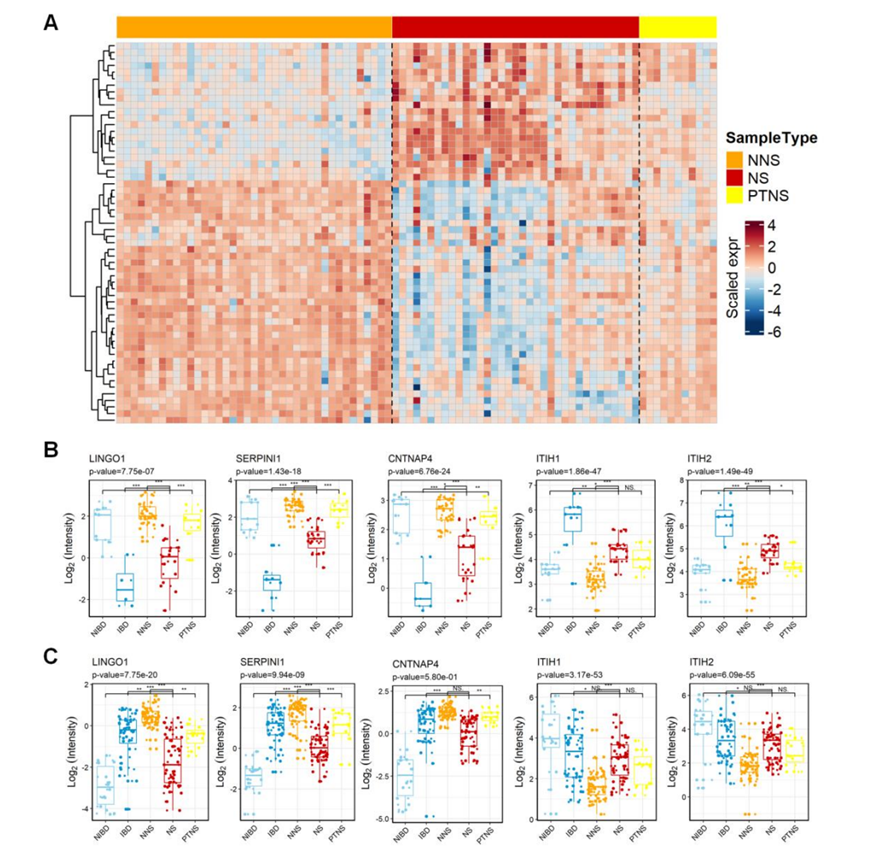
图S5. 鉴别NS与PTNS的潜在生物标志物。
(a) 热图显示PRM-MS验证的差异表达蛋白 (b-c) 采用DIA鉴定和PRM-MS验证检测NS、PTNS、IBD、NIBD和NNS样品CSF中LINGO1、SERPIN1、CNTNAP4、ITIH1和ITIH2的蛋白表达水平。
+ + + + + + + + + + +
结 论
在这项研究中,蛋白质组学和机器学习模型技术用于表征 223 个脑脊液 (CSF) 样本,以识别 NS 的诊断标记物,并深入了解相关炎症反应的潜在机制。通过对另外 115 个脑脊液样本的多中心验证, SEMA7A、SERPINA3 和 ITIH4可作为 NS 诊断的标志物。预计所鉴定的生物标志物将成为辅助诊断 NS 的有效工具。本项研究对脑组织中 NS 发病机制的见解可能会为 NS 患者的治疗策略和药物发现提供新的理论依据。
+ + + + +



 English
English


