文献解读|Cell Rep(8.8):恶性和正常葡萄膜黑色素细胞的多组学比较揭示了葡萄膜黑色素瘤的分子特征
✦ +
+
论文ID
原名:Multi-omics comparison of malignant and normal uveal melanocytes reveals molecular features of uveal melanoma
译名:恶性和正常葡萄膜黑色素细胞的多组学比较揭示了葡萄膜黑色素瘤的分子特征
期刊:Cell Reports
影响因子:8.8
发表时间:2023.09.13
DOI号:10.1016/j.celrep.2023.113132
背 景
葡萄膜黑色素瘤 (UM) 是一种罕见的癌症(每年每百万人中有 5-7 例),主要影响成年人,占所有黑色素瘤的 5%。UM 是眼睛葡萄膜(包括虹膜、睫状体和脉络膜)黑素细胞恶性转化的结果。UM原发肿瘤可通过手术和/或放疗得到良好控制;然而,超过30%的患者出现转移,主要发生在肝脏,预后极差。提高对侵袭性UM的理解对于确定有效的新治疗方法至关重要。
实验设计
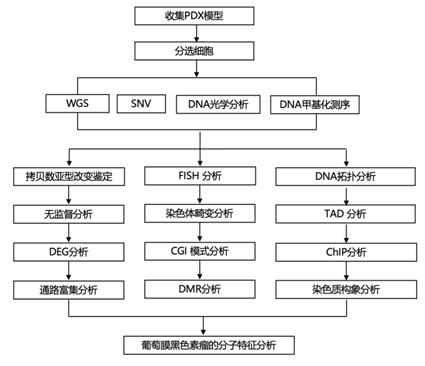
结 果
01
研究样本和模型建立
研究者团队从两种侵袭的 PDX 模型 MP41 和 MP46 中分选了 UM 细胞。MP41模型是通过对一名50岁女性患者进行UM去核而生成的,该患者在初次诊断后31个月内发生转移,并在诊断出多发转移(包括骨、肺和皮下病变)后43个月死亡。MP46模型是通过摘除一名69岁男性患者的肿瘤而建立的。该患者在原发肿瘤诊断后 6 个月出现肝转移,并在初次诊断后 7 个月死亡(图 1 A)。
这两种侵袭性模型在GNAQ/11中包含典型的激活突变,并共享 8q 和 6p 染色体增益。MP46 显示 3 号染色体等二体性,并且通过免疫组织化学 (IHC) 检测发现 BAP1 缺陷(图 1 B)。并且通过 Sanger 测序未发现BAP1、SF3B1 或 EIF1AX中存在突变。
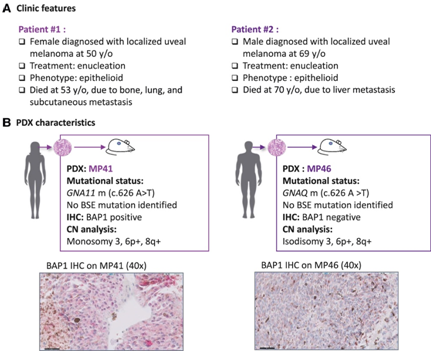
图1. MP41和MP46 pdx的主要特征建立于侵袭性葡萄膜黑色素瘤。
(A) UM 病例的临床特征。(B) 先前建立和表征的相应患者来源异种移植模型的主要分子特征。
02
全基因组测序和拷贝数分析证实MP41和MP46是高危UM
首先,他们对 MP41 和 MP46 进行全基因组测序 (WGS),以进行单核苷酸变异 (SNV) 和拷贝数分析。TCGA UM 研究区分了具有不同非整倍体事件的四种拷贝数亚型,并根据体细胞拷贝数改变将二体性 3 (D3)-UM 和 M3-UM 分为两个亚组。从 MP41 和 MP46 模型的全基因组测序中鉴定出的体细胞拷贝数改变为丢失 (L)、增加 (G) 和单体 (M),特别包括 MP41 的:M3、G6p、L6q、L8p、G8q,以及 MP46 的:isodisomy 3、 G6p、L8p、G8q(图2A-B)。根据UM的TCGA拷贝数亚型将MP41和MP46分别分为第2组和第4组。MP46的分类与第4组bap1缺失肿瘤的富集一致。虽然TCGA组2在SF3B1突变的UM中富集,但在MP41中没有观察到SF3B1或SRSF2突变,也没有观察到SF3B1剪接模式。总的来说,对MP41和MP46模型的WGS分析证实了在Gαq通路中存在一个独特的致癌驱动突变,即M3和G8q的存在,并揭示了TCGA拷贝数与MP41组2和MP46第4组的关联。
接下来,他们进行了基因表达分析,以比较 UM 模型和正常脉络膜黑素细胞 (NM)的转录组。使用无监督主成分分析和所有差异表达基因的层次聚类,他们发现高度再现性以及 UM 模型和 NM 之间的清晰分离(图2C-D)。
为了识别侵袭性 UM 中一致的差异表达基因 (DEG),他们将每个 PDX 与 NM 进行比较,然后比较生成的基因列表(图 2 E)。在MP46中鉴定的9368个基因中,4337个基因过表达,5031个基因过表达。MP41和MP46之间重叠基因包括3066个下调基因和2334个上调基因。
接下来,他们使用Reactome分析来鉴定DEG中富集的通路(图2G)。这些通路包括增殖相关通路(细胞周期、有丝分裂、检查点)以及染色质维持和DNA修复通路。
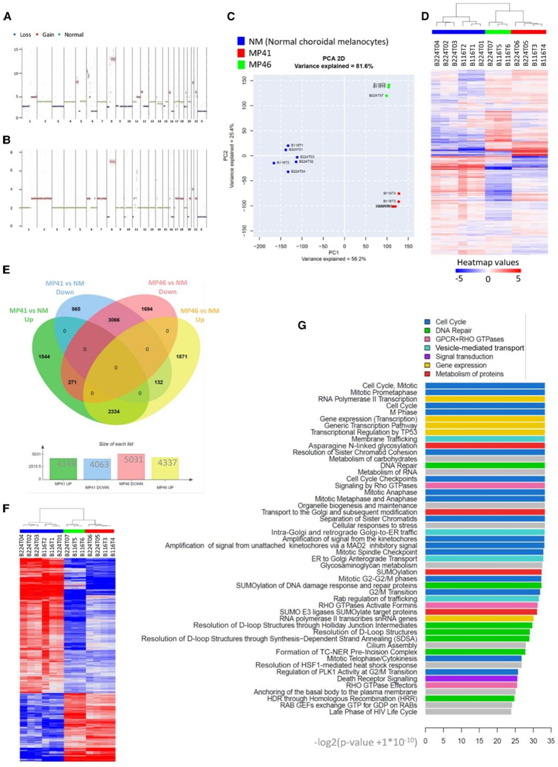
图2. 基因组和基因表达全局概览。
(A-B)通过全基因组测序建立的 MP41 和 MP46 拷贝数谱。(C) 6个正常脉络膜黑素细胞样本(蓝色)和4个MP41 UM细胞制剂(红色)的RNA-seq主成分分析。三种MP46 UM细胞制剂(绿色)。(D)相同剖面的层次聚类。(E) MP41 与 NM 和 MP46 与 NM 的差异基因表达分析。(F) MP41 与 NM 和 MP46 与 NM 中常见调控基因的热图。(G) 对常见调控基因进行反应组分析。
03
光学分析和FISH分析揭示了主要的染色体畸变
为了进一步研究与UM相关的基因组畸变,他们使用Bionano平台进行了光学定位,并在MP41和MP46上进行了端粒和着丝粒染色和M-FISH (TC + M-FISH34)。
光学图谱达到了500 bp的分辨率,揭示了两种模型中的远程DNA改变,包括易位、插入、重复和小缺失(图3A-C)。
在MP41中,光学定位显示染色体内和染色体间易位((t(19;19) t (1;12)、t(6);8))。在MP46中,chr19染色体内易位和染色体间易位(t(1;22))。还鉴定出了结构变异(SV),包括缺失、插入和重复(图3C)。TC M-FISH显示MP41的超三倍体基因组具有双中心染色体(dic(14;16), i(8q),dic(1;11;8))(图3D)。染色体末端-末端融合常与双着丝粒染色体和染色体结构异常有关。MP46也显示了复杂的核型(图3E):具有多条双着丝粒染色体的超二倍体基因组:dic(1;17)、dic(6;10)、dic(8;21)、dic(13;22)、dic(16;20)和dic(20;22)。在MP46中发现了两个易位:t(1;22)和der(15)t(11;15)。
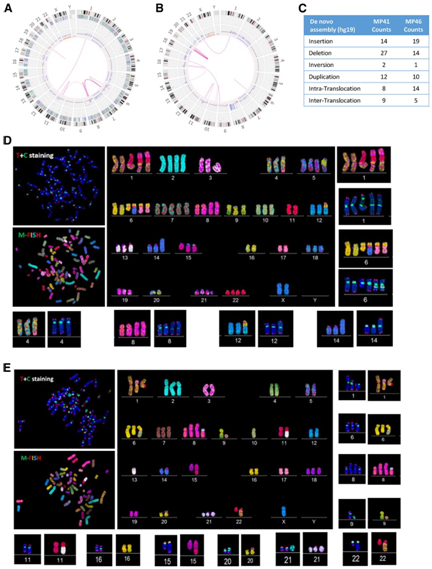
图3. MP41和MP46的DNA光学图谱和FISH分析。
(A-B) MP41 和 MP46 中的 Circos 像差图。(C) Bionano光学图谱详细描述了MP41和MP46的插入、缺失、反转、重复以及内部和内部易位的数量。(D) MP41和(E) MP46的端粒和M-FISH分析来自两种不同的FISH分析。
04
DNA甲基化分析揭示了CpG岛(CGI)模式的差异,并确定了BAP1启动子缺失
为了表征 DNA 甲基化模式,他们对 UM 和 NM 进行了 OxBS 测序。首先,随机森林分析将MP41和MP46分别置于TCGA甲基化第2组中。接下来,他们根据以下基因组定位对甲基化水平进行分类:CGI启动子、非CGI启动子、外显子、内含子、基因间区域和重复元件(使用RepeatMasker注释识别)(图4A)。虽然CGI启动子在UM样本和NM样本中通常是相同的去甲基化,但与所有基因组定位类别的正常样本相比,肿瘤样本中非CGI启动子的甲基化程度总体较低。
接下来,他们确定了每个PDX中的差异甲基化区域(DMR)(图4B)。大多数DMR在UM中低甲基化(H-),与整体甲基化模式一致(图4A)。高甲基化(H+) DMR 的基因组定位在 MP41 和 MP46 中存在差异,最大差异出现在重复元件和 CpG 岛中。692 个共享 H+ 的定位主要位于重复元件中,一个子集位于 CGI 和非 CGI 启动子中(图 4 B)。
MP46 在启动子中显示出特定的超甲基化模式(CpG129,UCSC Genome Browser [hg19;chr3:52443678–52445104]),与全基因组OxBS数据中鉴定的809-bp缺失的边界共定位(图 4 C-D)。
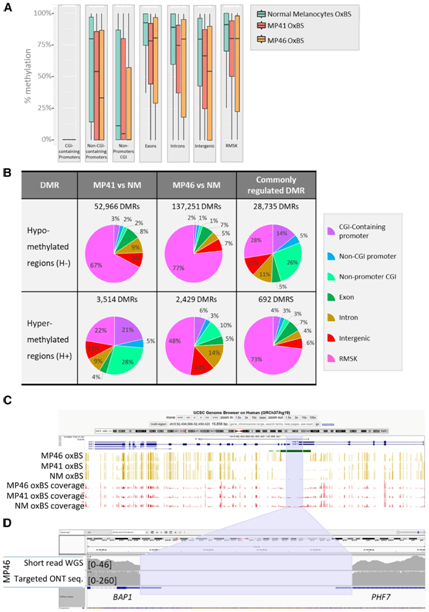
图4. UM模型和正常黑素细胞的全基因组DNA甲基化分析。
(A) DNA甲基化水平。(B) MP41 与 NM 以及 MP46 与 NM 中 300 kb 窗口中的差异甲基化区域 (DMR) 为低 DMR 和高 DMR(H– 和 H+)。(C) CpG甲基化百分比。(D) MP46短读测序(第一行)和靶向ONT测序(第二行)的IGV视图显示了BAP1和PHF7基因中启动子/5' UTR缺失的边界。
05
DNA拓扑分析揭示了包含大多数差异表达基因的稳定区室和拓扑相关域
黑素细胞基因组的空间组织,特别是它们的物理相互作用,可能有助于转化过程中基因表达的调节。他们进行了染色体构象捕获 (Hi-C),以阐明基因表达变化是否与 MP41 和 MP46 中的染色质组织和 DNA 折叠相关(图 5 A)。基因组折叠在多个长度尺度上存在,染色体区域包含物理分离的正染色质和异染色质区域,称为A区和B区,以及由环挤压产生的拓扑相关结构域(TAD)。
首先,他们以 250 kb 分辨率进行了区室分析,在 NM、MP41 和 MP46 中(并分别用三字母代码来指代每个窗口,以了解其在 NM、MP41 和 MP46 中的隔室状态)。大多数区室 (∼72%) 在三个模型之间具有相同的状态:A 区室对应于常色区域和活性区域(标记为“AAA”),B 区室对应于异色区域和非活性区域(标记为“BBB”)(图5B)。
他们进一步分析了区室状态的变化是否与基因含量和表达重叠(图5C-D)。大多数DEG位于组成活性的A区室(AAA),当校正了基因总数和每个区室的基因数时,这种富集并不显著。在UM模型中,ABB和BAA区分别对应特定的非活性区和活性区,含有159个DEG。
其次,其次,使用 40 kb bin 中的绝缘评分分析在两个 UM 模型中对 TAD 进行了分析。UM 和 NM 之间未观察到 TAD 的数量或大小存在显著差异(图5E-F)。
总之,未发现 TAD 结构存在差异,并且大多数 DEG发现于未从 NM 变为 UM 状态的隔室中(图4 C)。然而,159 个 DEG与区室状态的变化相关:63 个 DEG 属于 MP41 和 MP46 中的活性区室,96 个 DEG 属于非活性区室,表明拓扑结构的差异可能是基因表达差异的基础。
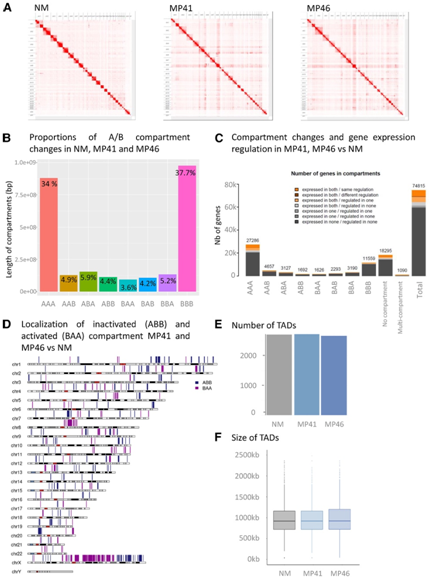
图5. NM 和 UM 中的区室和 TAD。
(A) NM、MP41和MP46的全基因组水平原位Hi-C接触图谱。(B) NM、MP41和MP46的区室变化直方图。(C)从全基因组的角度来看,MP41和MP46中失活(ABB)和活化(BAA)的区室与NM的定位。(D) NM、MP41和MP46间细胞室变化和基因表达的整合。(E和F) NM、MP41和MP46中TAD的数量和大小。
06
染色质拓扑和组蛋白标记变化与PRAME上调相关
为了进一步丰富对NM、MP41和MP46的染色质组织和基因表达调控的理解,他们对活性表观遗传标记H3K4me3和抑制标记H2AUb和H3K27me3进行了ChIP-seq分析。如图5D所示,Hi-C在PDX样品(BAA)中特异性鉴定出371个活性区室,MP41和MP46与NM相比,含有63个一致的DEG(37个较高,26个较低)。活化区室中37个上调基因中,PRAME和ZNF280A在H3K4me3中富集。33个基因(包括PRAME、ZNF280 A/B、EZH2)显示H3K27Ac峰,2个基因(PITX2和COL4A5)丢失H3K27me3标志,4个基因在其启动子区发生去甲基化。在26个下调的基因中,只有一个基因(ZC4H2)丢失了H3K4me3, 20个基因包含H3K27Ac标记,没有基因显示DNA高甲基化启动子。
与活化区室相关的上调基因包括EZH2、EPHA4和PRAME(图6A)。PRAME基因位于PRAME下游串联锌指蛋白ZNF280A和ZNF280B之间的22q11.22 (hg19 chr22:22890123-22900022)和PRAME上游推定的膜糖蛋白(POM121L1P)基因之间(图6D)。在这个位点上,与NM相比,只有ZNF280A、ZNF280B和PRAME在激活的隔室中上调(图6A)。他们还观察到NMs中PRAME启动子的超甲基化和MP41和MP46肿瘤模型中的低甲基化(图6B),这与UM中PRAME表达的激活相关(图6A-D)。
与UM MP41和MP46模型相比,PRAME位点的染色质构象分析显示NM有不同的模式。在NM中,接触在整个基因座中密集连接(图6C),而在两种肿瘤模型中,总体接触密度降低,“反对角”模式明显,与染色质开放一致,并由一组受限的相互作用锚定(图6C)。
他们对MP41和MP46 PRAME位点的组蛋白标记进行了分析,发现存在活性组蛋白标记(H3K4me3和H3K27Ac),而不存在抑制组蛋白标记(如H3K27me3)。在NM中,PRAME启动子上未观察到H3K4me3和H3K27me3峰(图6D)。
重要的是,H3K27Ac图谱显示MP41和MP46在PRAME启动子的近端以及ZNF280B下游的远端都有一致的峰。为了研究这些H3K27Ac区域是否存在物理相互作用,他们以高分辨率分析了Hi-C相互作用。将分析锚定在远端峰“增强子1”或PRAME启动子近端峰“增强子2”,结果显示在两种肿瘤模型中,相互作用明显富集,而在NM中则没有(图6E),证实了与这一远端调节位点存在三维环状相互作用。
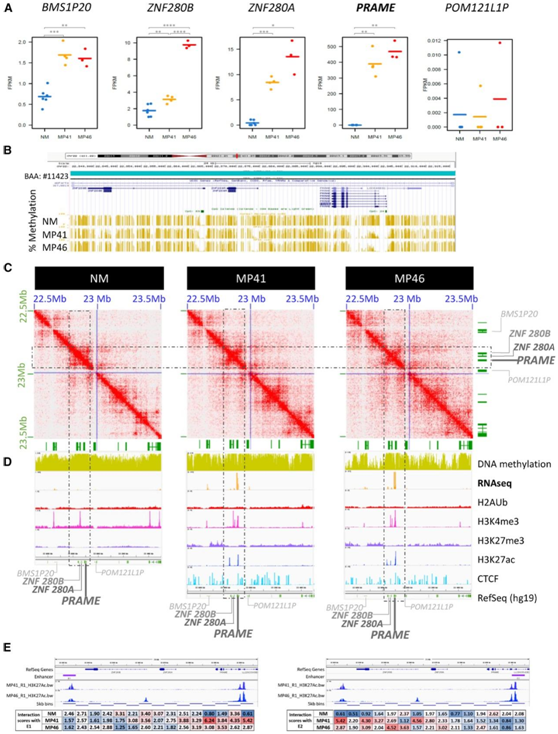
图6. PRAME位点的多组学分析。
(A) PRAME及其邻近基因作为BMS1P20、ZNF280B和ZNF280A上游基因以及 POM121LP 下游基因的基因表达。(B) UCSC基因组 (hg19) DNA甲基化百分比。(C)在PRAME TAD中NM、MP41和MP46在5kb分辨率下的DNA接触图谱。(D) UCSC的PRAME基因座视图,显示DNA甲基化、RNA-seq (log2)、H2AUb、H3K4me3、H3K27me3、H3K27Ac、CTCF和RefSeq基因。(E) PRAME启动子与潜在远端增强子E1的H3K27Ac标记和HiC相互作用。
+ + + + + + + + + + +
结 论
本项研究对两种侵袭性UM患者来源的异种移植模型与正常脉络膜黑素细胞进行了广泛的多组学特征分析(包括全基因组分析、DNA甲基化测序、DNA光学图谱、特异性组蛋白修饰和使用Hi-C进行的DNA拓扑分析)。本项研究的基因表达和细胞遗传学分析表明,基因组不稳定性是UM的一个标志,还发现BAP1启动子的反复缺失导致表达缺失,并与UM患者的高转移风险相关。Hi-C显示染色质拓扑结构改变与PRAME上调相关,PRAME是UM的独立预后生物标志物,也是潜在的治疗靶点。本项研究结果说明了多组学方法能提高对肿瘤发生的理解,并揭示了UM中基因表达失调的两种不同机制。
+ + + + +



 English
English


