文献解读|Cell(64.5):分子制图揭示散发性结直肠肿瘤的进化和微环境动态
✦ +
+
论文ID
原名:Molecular cartography uncovers evolutionary and microenvironmental dynamics in sporadic colorectal tumors
译名:分子制图揭示散发性结直肠肿瘤的进化和微环境动态
期刊:Cell
影响因子:64.5
发表时间:2023.12.07
DOI号:10.1016/j.cell.2023.11.006
背 景
结直肠癌 (CRC) 进展的遗传模型定义了导致人类结肠上皮发育不良和恶性肿瘤的突变事件的累积序列。这种传统的腺瘤-癌轨迹涉及驱动基因APC、KRAS和TP53的改变,导致染色体不稳定 (CIN)。由所谓的锯齿状途径产生的散发性 CRC 的子集更有可能是BRAF驱动的和微卫星不稳定 (MSI-H),这主要是由于MLH1的双等位基因超甲基化导致超突变。
实验设计
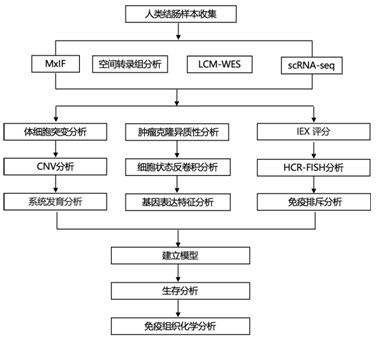
结 果
01
空间图谱探索散发性结直肠肿瘤的分子异质性层
为了模拟结合空间异质性的 CRC 进展,研究者团队选择了 31 个人类结肠标本,其区域形态代表肿瘤进展阶段之间的转变,他们鉴定了同时存在恶变前、恶性和侵袭区域的样本(图1A-B)并进行了多重免疫荧光 (MxIF) 分析(图1C)。肿瘤分期、分级和位置的多样性代表了微卫星稳定 (MSS)和MSI-H crc以及癌前肿瘤亚型。他们采用空间转录组学 (ST)和全外显子组测序 (LCM-WES) 来进行基因分析单个肿瘤的空间不同区域中的感兴趣区域(ROI)(图1C-D)。传统的 TA/TVA 息肉和 MSS CRC 富含干性、内在共有分子亚型 2 (iCMS2) 上皮特征和 CD4 + T 淋巴细胞主导的免疫反应,而 SSL/HP 和 MSI-H CRC 由化生和iCMS3 特征伴随细胞毒性(CD8 + T 细胞)免疫反应(图1E)。此外,将每个患者的多区域外显子组测序汇总到批量分析中,发现TA/TVA/MSS肿瘤中APC、KRAS和TP53突变,与传统的腺瘤-癌序列一致,并且在SSL/HP/MSI-H样本中存在BRAF变异(图1F)。MSI-H crc表现出在其SSL/HP对应体中不存在的超突变,这表明这种转变发生在锯齿状的恶性前病变之后此外,一小部分传统途径肿瘤(1个TVA - HTA11_01938和1个MSS- PAT15211)鉴定为超突变(HM)(图1F)。
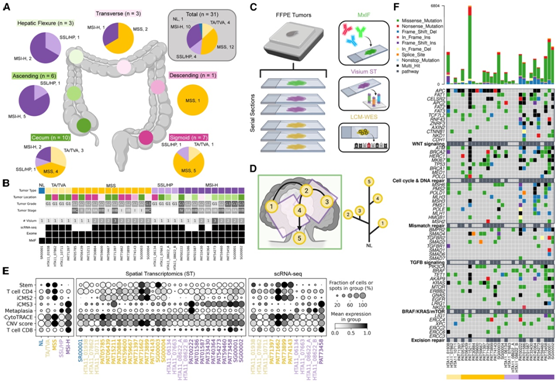
图1. 空间图谱分析散发性结直肠肿瘤的分子异质性层。
(A-B) 详细说明为图集实验选择的结直肠标本的图表。(C) 实验设计,包括对 FFPE 组织块的连续切片进行分层空间分子测定。(D) 根据 LCM-WES 数据绘制的系统发育地理学图,其中包含 ST 和 MxIF 图像。(E) 按肿瘤类型(左)、ST 患者(中)和匹配的 scRNA-seq 患者(右)划分的基因特征评分摘要。(F) LCM-WES 样本中检测到的体细胞突变。
02
拷贝数变异(CNV)推断建立了空间分辨的肿瘤克隆及其系统发育关系
引起 MSS CRC 的传统腺瘤-癌序列的一个标志是染色体不稳定(CIN),它会导致可遗传给细胞后代的大段 DNA 的体细胞获得、丢失和重排。从上皮ST微孔中推断的CNV谱的降维和嵌入产生肿瘤特异性聚类,表明大的连续拷贝数变化(图2A)。
为了通过基因组改变的直接测量进一步验证这些推断的CNV结果,他们对来自该图谱的肿瘤子集以及来自更大队列的其他恶性前病变和crc进行了全基因组和/或全外显子组测序(分别为WGS和WES),证明具有APC突变的传统腺瘤 (TA/TVA) 表现出与锯齿状息肉 (SSL/HP) 和基线正常上皮相当的低 CIN(图2B-C)。他们对选定的肿瘤子集进行了WGS和WES,并根据这些数据类似地计算了总CNV评分,这证实了从基因表达推断的TA/ tva中缺乏CIN(图2H)。在CIN+肿瘤亚群(主要是MSS)中,肿瘤克隆区可以通过不同的推断CNV谱来定义,这些CNV谱在空间上与组织学注释的组织结构域以及增生异常上皮中富集的基因特征一致(图2A-D)。具体来说,ST CNV图谱基于相似性聚在主要肿瘤克隆中,而正常粘膜和间质区域聚集在一个背景聚类中(“S”)(图2D-H)。
此外,CNV 推断还识别出 ST 微孔中上皮和周围基质混合产生的肿瘤边缘区域 (“E”),该区域抑制了 CNV 信号。这些区域可用于表征肿瘤边界处发生的上皮活动,例如上皮间质转化 (EMT) 和肿瘤细胞侵袭,以及与肿瘤微环境 (TME) 的相互作用,包括抗原呈递、淋巴细胞耗竭、细胞毒性和中性粒细胞募集(图2D-G)。在CNV谱不同的CIN+肿瘤中,主要克隆区可以根据其在每个肿瘤中的推断进展阶段进行排序和标记,由共有的CNV谱和总体CNV负担确定(PAT71397克隆区“1”、“2”、“3”)(图2E-H)。在单个肿瘤基础上对CNV克隆进行排序,可以通过与恶性和进展趋势相关的组织特征得到验证,例如在PAT71397中,肿瘤突变负担(TMB)、CD4+ T细胞和抑制性T细胞浸润的增加,以及包括iCMS2、干性和CytoTRACE在内的基因标记评分(图2G)。
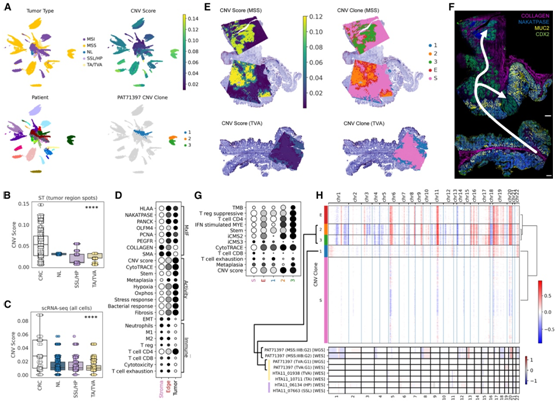
图2. CNV 推断建立空间分辨的肿瘤克隆及其系统发育关系。
(A)UMAP可视化。(B) 图谱主要 CNV 克隆区域中所有 ST 微孔的 CNV 评分箱线图。(C)上皮细胞 CNV 评分箱线图。 (D) 通过 CNV 推断确定的主要组织域的 MxIF 强度、细胞活性和免疫基因特征总结。(E) PAT71397 的 CNV 评分(左)和肿瘤克隆区域(右)。(F) MxIF 以及 PAT71397 的推断进展轨迹。(G) PAT71397 的 CNV 克隆区域的 TMB、CNV 评分和基因特征。(H) PAT71397 ST 推断的 CNV 热图。
03
多区域体细胞突变谱提供系统发育地理学拓扑
系统发育重建可以及时了解肿瘤的进展,特别是从肿瘤切除和固定的那一刻起,他们的分析揭示了肿瘤内空间克隆异质性(图3A-C)。ROI 之间常见和独特的遗传改变允许重建肿瘤区域之间的进化关系,这可以遵循几种提出的进化模型之一(图3D)。当按系统发育结构对图谱中的癌症进行分类时,他们观察到 22 个肿瘤中有 8 个表现出线性进化,其特征是许多共有突变和低区域异质性。在其余 14 种癌中,他们将 6 种归类为中性癌(实际上与线性癌相反,共有突变很少且区域异质性较高),另外 8 种归类为分支癌。有趣的是,他们将 15 个 CIN+ 肿瘤中只有两个的进化归类为线性。相反,线性进化在 HM 队列中占主导地位(图3E)。事实上,CIN+ 特征(如 iCMS2、干细胞、CD4 T 细胞和高 CNV 评分)在具有中性和分支进化的肿瘤中丰富,而 HM 标记(包括 iCMS3、化生和 T 细胞耗竭)在经历线性进化的肿瘤中高度表达(图3F)。
接下来,他们建立了排序并计算了定义为 LCM ROI 和 CNV 克隆组合的肿瘤区域的CIN 指数(量化 CIN 和超突变的比较程度)(图3E-G)。重要的是,他们注意到,在具有高 CIN 指数的肿瘤区域中,TP53突变比APC突变更丰富,证明CIN 在 CRC 发展过程中出现较晚(图3G)。
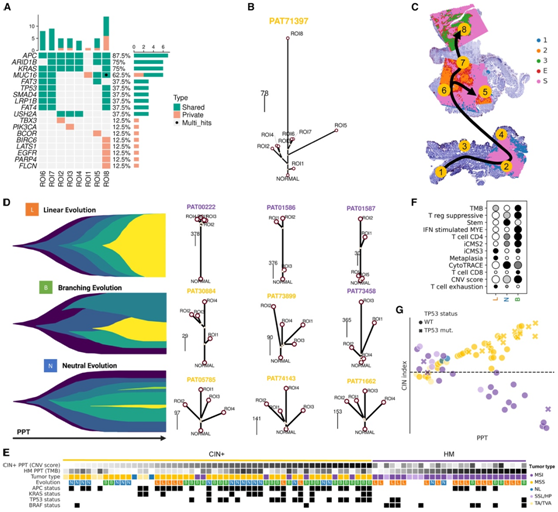
图3. 多区域体细胞突变谱的系统发育分析。
(A) PAT71397 的空间采样 LCM ROI 中检测到的驱动突变的 Oncoplot。(B) PAT71397 的系统发育树。(C) PAT71397 块中的 LCM ROI 图表,覆盖在 ST 中识别的 CNV 克隆区域上。(D)肿瘤进化模式图。(E) 肿瘤区域及其临床和突变元数据按类别划分,并按相应的 PPT 从左到右排序。(F) 所有肿瘤区域的基因特征总结。(G) CIN 指数与来自 (E) 的肿瘤区域的 PPT。
04
细胞状态反卷积分析
他们发现了30多种不同的细胞状态,这些细胞状态包括正常粘膜细胞,如聚类状细胞 (TUF)、肠内分泌细胞 (EE1-2) 和杯状细胞 (GOB);源自锯齿状病变 (SSC) 和癌 (CRC1-4) 的肿瘤特异性状态;以及浸润性免疫群体,包括辅助性 T 细胞 (TL1)、细胞毒性 T 细胞 (TL2) 和中性粒细胞 (MYE4) 等。反卷积的细胞状态分数与连续组织切片中选定的 MxIF 细胞类型标记表达以及基于文献的细胞类型和细胞活性基因特征的表达相关(图4A-C)。例如,GOB、ABS(吸收性结肠细胞)和 CT(隐窝顶部结肠细胞)富集与正常上皮中的高 MUC2 IF 染色一致,MYE1(M1 巨噬细胞)与 CD11B 染色一致,以及 CRC2/STM 富含区域(CIN+ 肿瘤)细胞和干细胞样细胞)在 PAT30884 中表达 PCNA 和 OLFM4(图 4 A)。SSL, HTA11_08622_A,进一步验证了refNMF状态,如ABS、成纤维细胞(FIB2)和锯齿状特异性细胞(ssc)在粘膜和粘膜下层的空间分布,以及与MUC5AC和AQP5 IF染色的对应关系(图4B-C)。
由于空间分辨率低,微孔间距离大,将细胞-细胞相互作用群落重建算法应用于ST数据的结果不准确。专注于基于像素的社区检测,他们采用refNMF算法作为MILWRM(区域形态学多重图像标记)模型的预测因子,根据细胞状态组成将组织划分为一致域(图4D)。MILWRM模型产生8个结构域(D0−D8),对应于CIN+上皮(D4;CRC2和STM高表达),正常黏膜(D5;ABS和CT富集),无柄锯齿上皮(D0;高SSC和GOB),除其他外,所有这些在空间上都与区域组织学一致(图4A-E)。
当总结跨MILWRM结构域和肿瘤类别的refNMF丰度时,他们可以识别出由独特肿瘤群体富集的上皮细胞状态(CRC1 = HM;CRC2 = CIN+),对每一类肿瘤的微环境组成有了粗略的了解(图4F)。最后,他们注意到MILWRM结构域D1是HM的特征,而D4是CIN+肿瘤的特征(图4E)。
通过对CIN+和HM肿瘤的CNV评分和TMB分别进行进展伪时间(PPT)排序,他们追踪了肿瘤进展的全局指标refNMF状态的变化,并建立了广义加性模型(GAM)(图4G)。
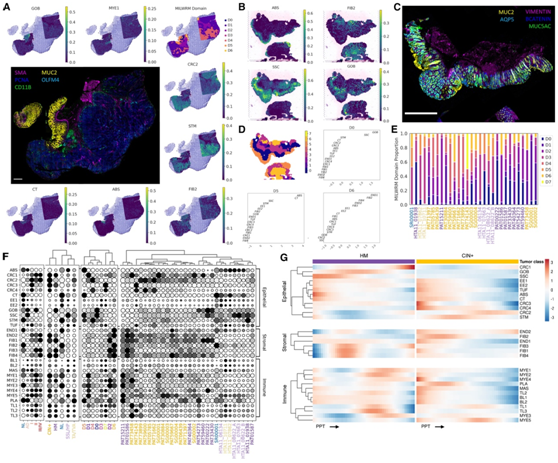
图4. 细胞状态反卷积揭示伪颞组织动力学。
(A) 示例PPT MxIF 周围环绕着七种细胞状态的 refNMF 用法以及投影到 PAT30884 组织学上的 MILWRM 组织域。(B) HTA11_08622_A 的正常 ABS、FIB2、SSC 和 GOB 状态的 refNMF 用法。(C) HTA11_08622_A 的 MxIF 图像。(D) HTA11_08622_A 的 MILWRM 组织域分析。(E) 在每位患者的 ST 中检测到的 MILWRM 结构域的比例。(F) refNMF 状态按区室分组,并对所有 ST 样本的肿瘤分期、肿瘤类别、MILWRM 域和患者进行总结。(G) GAM 热图适合 HM(左)和 CIN+(右)肿瘤按 PPT 排序的所有 ST 肿瘤区域中的 refNMF 状态。
05
CIN+ CRC 的基因表达特征预测免疫排斥
为了进一步研究 CIN+ 肿瘤进展与免疫排斥之间的联系,他们将 GAM 分析扩展到 ST 图谱数据中的非靶向基因表达,鉴定了 HM 和 CIN+ CRC 的轨迹内和之间具有统计显著的基因,并按生物途径或功能对它们进行分组(图 5 A)。具体来说,他们了在晚期 CIN+ PPT 中显著的基因,这些基因形成了细胞外基质 (ECM) 信号传导和组织的功能模块(DDR1、TGFBI和PAK4)。此外,他们观察到 CIN+的DPEP1表达显著,该基因在 CRC2 上皮状态中也高度富集(图 5A)。
他们将DPEP1加入到DDR1、TGFBI和PAK4的特征中,分析该图谱中的免疫排斥,因为它与中性粒细胞募集有关,这可能导致TME中的淋巴细胞排斥。DPEP1是腺瘤和CRC中编码膜或分泌蛋白的基因子集中表达上调最多的基因之一,也是CRC细胞经典外泌体中发现的最丰富的蛋白质,其在CRC患者血浆中的水平高于正常对照,突出了其作为循环CRC生物标志物的潜力。这四个基因在肿瘤上皮中高度共表达,它们共同组合成一个免疫排斥特征(IEX),与iCMS2和CNV评分表现出空间相关性,并且在CIN+特异性MILWRM结构域D4中富集(图5B-C)。通过研究其他基因特征,他们发现了与IEX相一致的其他程序,如pEMT和氧化代谢,它们是晚期癌症的标志,并随着肿瘤进展而上调,证实了IEX在晚期PPT中的出现(图5C)。此外,在患者水平上,纤维化和缺氧特征与IEX相关,因为这些特征与微环境免疫抑制有关(图5C)。
免疫排除的结直肠癌图谱中的一个例子是PAT71662,它包含一个主要的CNV克隆区域,该区域描绘了ST中的上皮隔(图5D)。他们注意到,该 MSS 肿瘤的大部分区域属于 MILWRM D4,并且 CRC2、干细胞、纤维化和氧化代谢相应较高(图 5C-D)。空间共现分析显示,该肿瘤在肿瘤核心中表现出上皮固有 IEX 的富集,同时排除了 CNV 克隆 1 区域中的 T 淋巴细胞(图 5 E)。他们通过空间配准的 MxIF 数据确认了 CD8 +细胞的排除,展示了免疫浸润如何隔离到富含胶原蛋白的基质中(图 5F-G)。相反,免疫浸润的肿瘤如 PAT73458 表现出低得多的 IEX 上皮表达,而细胞毒性 (TL2) 和细胞毒性 (TL2) 的明显浸润(TL3) T 细胞在 ST 和 MxIF 数据中可见(图5H-5K)。
接下来,他们使用 MxIF 数据来验证 ST 结果,从而扩展了免疫排除分析,通过每个细胞中量化的各种标记蛋白染色的存在来识别免疫细胞亚群。免疫排斥的 PAT71662 其基质(“S”)中的免疫细胞比其肿瘤上皮(“E”和“1”)更多,而浸润性肿瘤 PAT73458 不仅表现出更高水平的细胞毒性(CD3+/CD8+)和辅助(CD3+/CD4+)T 细胞,但在肿瘤边缘和肿瘤核心区域的所有浸润免疫细胞中也占较大比例(图 5F-L)。
将相同的 MxIF 分析扩展到整个空间图谱,他们观察到具有高 IEX 分数的肿瘤区域仅具有少量 CD8+ T 细胞浸润,而 IEX 低区域是唯一具有大量 CD8+ T 细胞的区域(图5 M)。对66个人类scRNA-seq样本进行的类似普查显示,来自癌特异性上皮细胞(CSC)的强烈IEX信号,以及与上皮IEX评分相匹配的CD8+ T细胞谱(图5N)。
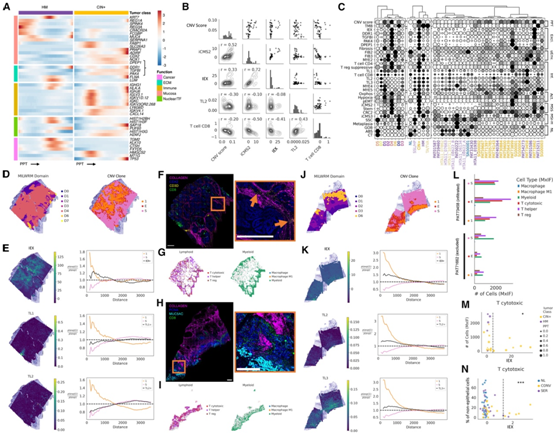
图5. CIN+ CRC 的基因表达特征预测免疫排斥。
(A) GAM 热图适合所有 ST 肿瘤区域总结的顶级基因。(B) 所有 CIN+ 肿瘤区域中进展指标(CNV 评分和 iCMS2)、IEX、细胞毒性 T 细胞 refNMF 状态 (TL2) 和 CD8 T 细胞基因特征之间的配对 Pearson 相关性。(C) 基因、基因特征和 refNMF 状态分为伪时间指标 (“PPT”)、免疫排除标记 (“Excl.”)、微环境细胞 (“uEnv.”)、浸润免疫细胞 (“Inf.”)、所有 ST 样本的肿瘤活性(“Act.”)以及 MSS、MSI-H 和正常粘膜的上皮特异性标记物,按 MILWRM 结构域、肿瘤类别和患者进行总结。(D) PAT71662 ST,带有来自 MILWRM(左)和 CNV 克隆区域(右)的注释组织结构域。(E) PAT71662 中 IEX、辅助 T 细胞 (TL1) 和细胞毒性 T 细胞 (TL2) 的表达叠加和空间共现分析。(F) PAT71662 MxIF 显示胶原蛋白、CDX2(标记 MSS 上皮)和淋巴细胞(CD3 和 CD8)。(G) 在整个载玻片空间中绘制的 PAT71662 MxIF 分段单细胞的质心,分为淋巴和骨髓区室。(H) 与 PAT73458 的 (F) 相同。(J-K) 与 PAT73458 的 (D) 和 (E) 相同。(L) 通过 CNV 克隆区域总结的 (G) 和 (I) 中 PAT71662(下)和 PAT73458(上)中浸润免疫细胞的分析。(M) MxIF 中检测到的浸润 CD8+ T 细胞数量与所有肿瘤区域的 IEX 评分作图。(N) CD8+ T 细胞占 66 个 scRNA-seq 样本中检测到的非上皮细胞总数的百分比。
06
IEX 随肿瘤进展趋势并预测患者预后不良
从人类样本生成的配对 scRNA-seq 数据与 ST 数据再次概括了他们的发现(图6A)。即,IEX主要由肿瘤细胞表达(图6B-C),并且IEX可以通过CD8+ T细胞浸润程度和T细胞激活状态来分离肿瘤样本(图6D)。杂交链反应荧光原位杂交(HCR-FISH)也证实了IEX基因在IEX+肿瘤中的恶性细胞中表达(图6E)。为了进一步探讨肿瘤进展和免疫排斥之间的联系,他们对图谱上所有肿瘤区域的 CIN+ 上皮固有 IEX 评分以及浸润免疫细胞状态和针对 PPT 的癌症进展特征进行了建模,证实了 IEX 富含 CIN+肿瘤和趋势与 CNV 信息的 PPT、iCMS2 和 CRC2 富集成正比(图 6F)。
接下来,他们通过强调其在预测具有较大样本量的外部队列中的患者结果方面的转化效用,着手验证该表达特征作为免疫排斥的预测因子。与 MSI-H (HM) TCGA COAD 和 READ 样本相比,MSS (CIN+) 的 IEX 评分显著升高(图6G)。与整个 TCGA 队列中低评分肿瘤患者相比,高 IEX 表达导致高评分肿瘤患者的无进展生存期 (PFS) 显著下降(图6H)。
他们从由 163 个结直肠腺癌样本组成的肿瘤微阵列 (TMA) 的免疫组织化学 (IHC)染色中,与 MSI-H 肿瘤相比,MSS 中总体 DDR1 和 TGFBI 蛋白表达更高(图6I-K)。对这个 TMA IHC 队列的生存分析显示,DDR1 和 TGFBI 具有高 IHC 染色的肿瘤的 PFS和总生存率(OS)较低(图 6 L)。
最后,他们在 CRC 同基因模型中通过改变 IEX 蛋白 DDR1 的表达,从功能上验证了 IEX 在抑制肿瘤进展中的作用。他们使用 CRISPR-Cas9 干扰CT26 小鼠结直肠细胞中的Ddr1表达,然后通过流式分选分离出表面 DDR1 水平 (DDR1r) 低于野生型对照的细胞。将 DDR1r 和对照细胞注射到 BALB/c 小鼠的侧腹以形成肿瘤。在为期 40 天的肿瘤生长检查中,将肿瘤分为不同的生长速率组——快、中、慢。有趣的是,只有 DDR1r 肿瘤归类为生长缓慢类别,没有一个对照肿瘤表现出类似的特征(图 6 M)。此外,观察到与对照组相比,DDR1r 条件下快速生长的肿瘤数量有所减少,在31 天时的肿瘤体积显著减小(图 6 N)。这些结果表明DDR1作为IEX的一部分,在抑制肿瘤生长和进展方面发挥着功能性作用。
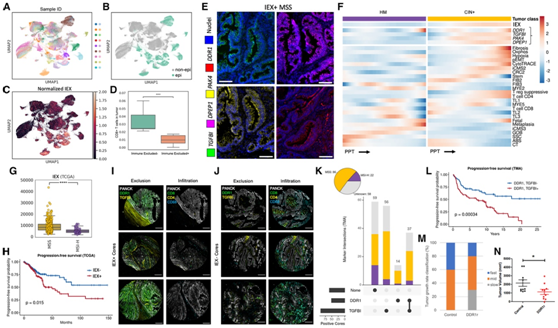
图6. IEX 随肿瘤进展趋势并预测患者预后不良。
(A–C) 为 CRC 样本生成的配对 scRNA-seq 数据与研究中的 ST 数据的 UMAP 。(D)基于配对 scRNA-seq 得出的 IEX 评分。(E) IEX+ MSS CRC 中TGFBI、DPEP1、PAK4、DDR1转录本的 HCR-FISH。 (F) GAM 热图适合所有 ST 肿瘤区域总结的基因、基因特征和 refNMF 细胞状态。(G) TCGA COAD 和 READ 样本中 IEX 分数的箱线图。(H) 来自 (B) 的 TCGA COAD 和 READ 样本的 Kaplan-Meier PFS 曲线。(I-J) MxIF 图像显示来自 CRC TMA 的代表性 (I) IEX+ 和 (J) IEX-核心的上皮标记(左)和免疫标记(右)。(K)饼状图显示了CRC TMA中MMR状态的细分(上),以及比较DDR1和/或TGFBI IHC染色评分高的CRC TMA核与两种标记评分低的核的IHC(下)。(L)生存分析。(M)对照和ddr1减少(DDR1r)同基因肿瘤中每种生长速率分类的百分比。(N)从(M)开始,对照组和ddr1减少组第31天肿瘤体积图。
+ + + + + + + + + + +
结 论
本项研究对 31 个人类结直肠标本的空间多组学数据进行分析,绘制出肿瘤进化的系统图谱,揭示个体化的进展轨迹以及伴随的微环境和克隆变化,根据其进化动力学对肿瘤进行分类,并将克隆区域沿着包含CIN+和HM途径的整体伪时间进展轨迹分析。整合的单细胞和空间转录组数据揭示了沿着伪时间进展的重复上皮程序和浸润免疫状态。他们发现了由细胞外基质调节因子DDR1、TGFBI、PAK4和DPEP1组成的免疫排除特征 (IEX) ,它与CIN+ 肿瘤进展相关,与细胞毒性细胞浸润减少相关,并在独立队列中显示出预后价值。该空间多组学图谱提供了对结直肠肿瘤-微环境共同进化的见解,可作为分层和靶向治疗的资源。
+ + + + +



 English
English


