文献解读|Cancer Cell(50.3):使用大型肝癌类器官生物库对肿瘤内异质性进行药物基因组分析
✦ +
+
论文ID
原名:Pharmacogenomic profiling of intra-tumor heterogeneity using a large organoid biobank of liver cancer
译名:使用大型肝癌类器官生物库对肿瘤内异质性进行药物基因组分析
期刊:Cancer Cell
影响因子:50.3
发表时间:2024.04.08
DOI号:10.1016/j.ccell.2024.03.004
背 景
原发性肝癌(PLC)是全球癌症相关死亡的第三大原因,其中包括肝细胞癌(HCC)、肝内胆管癌(ICC)和一种混合型肝细胞胆管癌(CHC)。肿瘤间和肿瘤内异质性(ITH)是癌症治疗的主要障碍。患者来源的类器官(PDO)培养已证明是重现肿瘤异质性和研究不同癌症类型的药物敏感性的有力工具,包括 PLC 中的疾病建模和药物筛选。因此,开发具有多区域采样的大规模 PLC 类器官活体生物库将有助于对肿瘤异质性进行广泛的表征,开发用于患者分层的预测生物标志物,并揭示耐药性的潜在机制以识别新的治疗机会。
实验设计
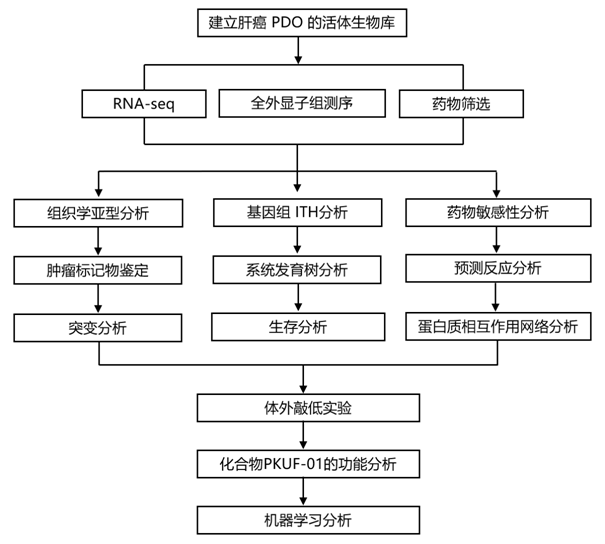
结 果
01
多区域类器官生物库概括了肝癌的组织学、基因组和转录组学特征
研究者团队利用多区域采样策略建立了肝癌 PDO 的活体生物库,该策略代表了 144 名肝癌患者切除标本中不同空间的区域(图 1 A)。简而言之,他们从每个组织样本中收集了 1-5 个区域(图 1 B),包括来自原发肿瘤的 522 个区域、来自肝转移的 6 个区域和来自邻近肝脏 (AL) 组织的 30 个区域。在142例原发性肿瘤患者(123例HCC, 18例ICC, 1例CHC)和2例肝转移患者中,共成功建立了399例肿瘤类器官和12例正常类器官。此外,通过多区域采样获得的肿瘤类器官与七种批准用于肝癌治疗干预的药物进行了筛选。基于H&E染色,观察到类器官与亲代组织之间的组织病理学相似(图1C),衍生类器官呈现出多样性的形态,从实体/致密结构(HCC和CHC)到更不规则形状的囊肿样结构(ICC)。病理标记分析表明,在HCC和ICC患者中,多区域类器官精确地显示HCC标记物(HepPar1/AFP)和ICC标记物(KRT19/EPCAM)作为亲本肿瘤组织,而CHC类器官在HCC标记物(AFP)和ICC标记物(EPCAM)中均显示阳性信号(图1D)。
为了评估开发的类器官生物库是否概括了PLC组织的肿瘤间和肿瘤内异质性,他们对来自36例患者的99对衍生类器官和亲本肿瘤组织进行了全外显子组测序和转录组分析 (RNA-seq)。通过 RNA-seq 对另外 156 个 PDO 进行了分析,总共 255 个 PDO 具有转录组和药物筛选特征(图 1 A),用于开发预测生物标志物。在类器官生物库中发现了反复突变的基因,包括TP53 (43%)、AXIN1 (11%) 和CTNNB1 (9%),其次,在肿瘤组织和类器官之间发现了87.5%的癌症相关突变的中位数一致性(图1E)。突变谱和显著拷贝数改变(CNA)峰(如12q12、17p13.3和22q11.23)在亲代肿瘤和衍生类器官之间高度相似(图1F)。大多数肝癌相关基因的克隆和亚克隆突变都保留在pdo中(图1G),如P13、P20和P23的克隆性TP53突变,以及P23的亚克隆性HIF1A突变,表明克隆结构可以保留在类器官中。进一步的表达分析在转录组水平上确定了成对肿瘤和类器官之间的高度相关性(图1H)。
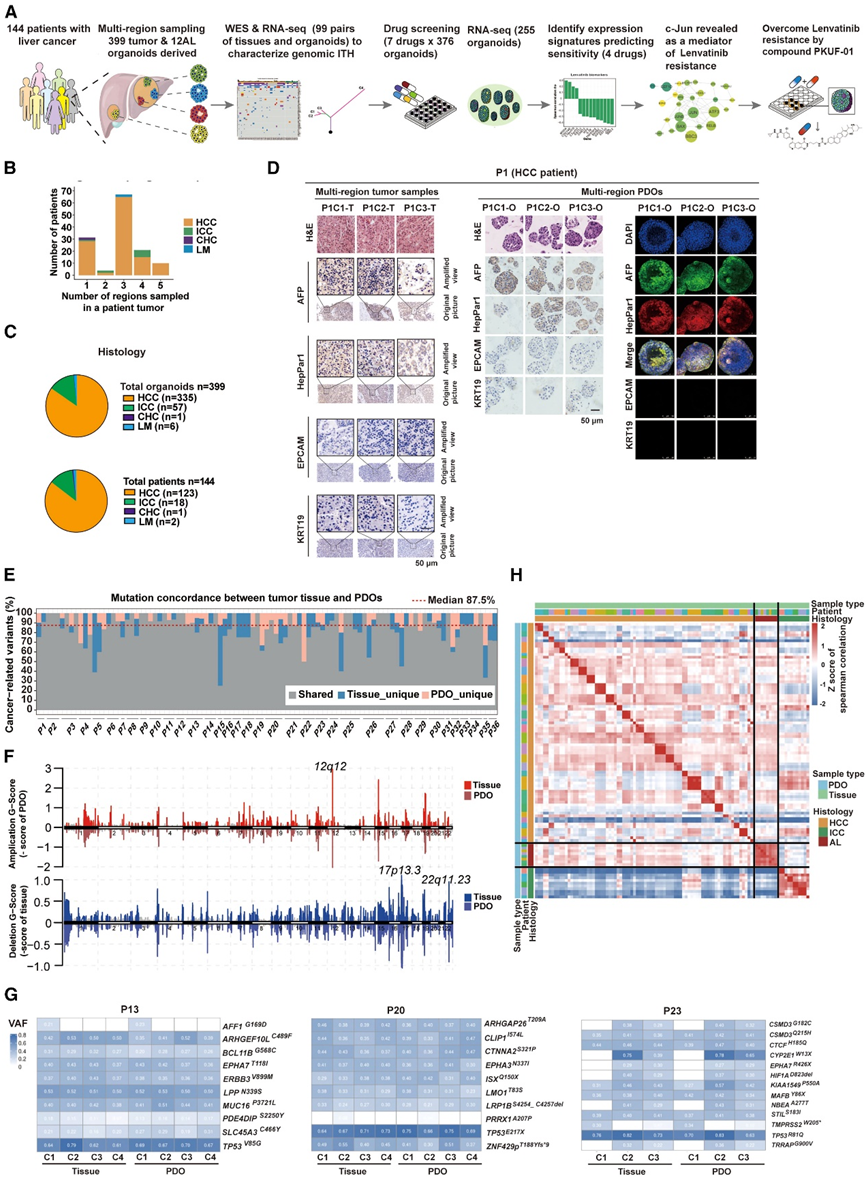
图1. 多区域类器官生物库概括了肝癌的组织学、基因组和转录组学特征。
(A) 研究的概要。从接受手术切除的肿瘤中获得多区域样本,并随后进行类器官培养和基因组分析。(B) 柱状图总结了从 144 名患者中采样的区域数量,颜色分别表示 142 名 PLC 患者和两名肝转移 (LM) 患者的组织学亚型。(C) 饼图说明所有 399 个类器官的组织学亚型。(D) HCC 患者 P1 的三个肿瘤区域的 H&E 和 IHC 染色,以及相应类器官的 H&E、IHC 和免疫荧光染色。(E) 肿瘤组织和类器官之间癌症相关体细胞基因组变异的一致性。(G) 热图比较三名代表性 HCC 患者肿瘤组织和 PDO 之间 COSMIC 癌症基因的突变VAF。(H) 热图显示肿瘤组织和配对类器官转录组之间的相关性。
02
肝癌基因组 ITH 的多区域类器官分析及其功能意义
他们研究了 32 名 PLC 患者的基因组 ITH,这些患者已建立并测序了多区域 PDO(图 2 A),并鉴定出大量的基因组 ITH。首先,包括 P1、P4、P6 和 P23 在内的一组患者在多区域样本中的肿瘤突变负荷 (TMB) 表现出明显的差异。接下来,发现多个 PLC 癌症相关基因在同一肿瘤采样的区域中具有异质体细胞突变和/或 CNA。例如,P1 的两个区域(P1C1 和 P1C2)含有致癌性TP53和AXIN1突变,而第三个区域(P1C3)则未检测到。同时,P1C3 具有独特的致癌APOB突变和额外的RB1缺失(图 2 A)。此外,另外 4 名患者(包括 P9、P19、P22 和 P23)仅在单个区域中发现了APOB突变。含有肿瘤抑制基因的染色体区域[包括 16p13.3 ( AXIN1 )、12q14.2 ( RB1 ) 和 10q23.2 ( PTEN )]的缺失也在一部分患者中存在异质性。
为了系统地表征基因组ITH,他们通过应用基于已识别的体细胞点突变的最大简约算法,为个体患者构建了系统发育树(图2B)。在PLC中发现了许多常见的主要事件,包括TP53、RB1、AXIN1和CCND1的突变。根据主干比,即普遍存在的突变与非普遍存在的突变的比率,12名患者的系统发育树是主干优势(主干比>1),而另外20名患者是分支优势。主干比例降低似乎与较高的 ITH 相关(图 2 C)。此外,突变-ITH 和 CNA-ITH 之间存在正相关性(图 2D),并且突变-ITH 和 CNA-ITH 水平均与队列中的患者预后显著相关(图 2E)。突变/CNA-ITH 水平较高的 PLC 患者的总生存期较差。
接下来,他们推测基因组和相关转录组异质性可能导致药物敏感性的异质性。使用来自 79 名患者的 255 个 PDO 研究了 PLC 一线药物索拉非尼和仑伐替尼的靶基因表达。一些患者,例如 P6 和 P32,在来自不同区域的类器官中表现出明显不同的靶基因表达(图 2F),表明药物反应可能存在肿瘤内异质性。为了确认这一点,他们用这两种药物处理了患者 P6 和 P32 的多个类器官培养物,发现靶基因表达水平降低的区域显示出耐药性增加(图 2F-G)。总体而言,这些结果揭示了 PLC 中广泛水平的基因组 ITH,这显示出预后相关性,并可能导致对药物治疗的异质反应。
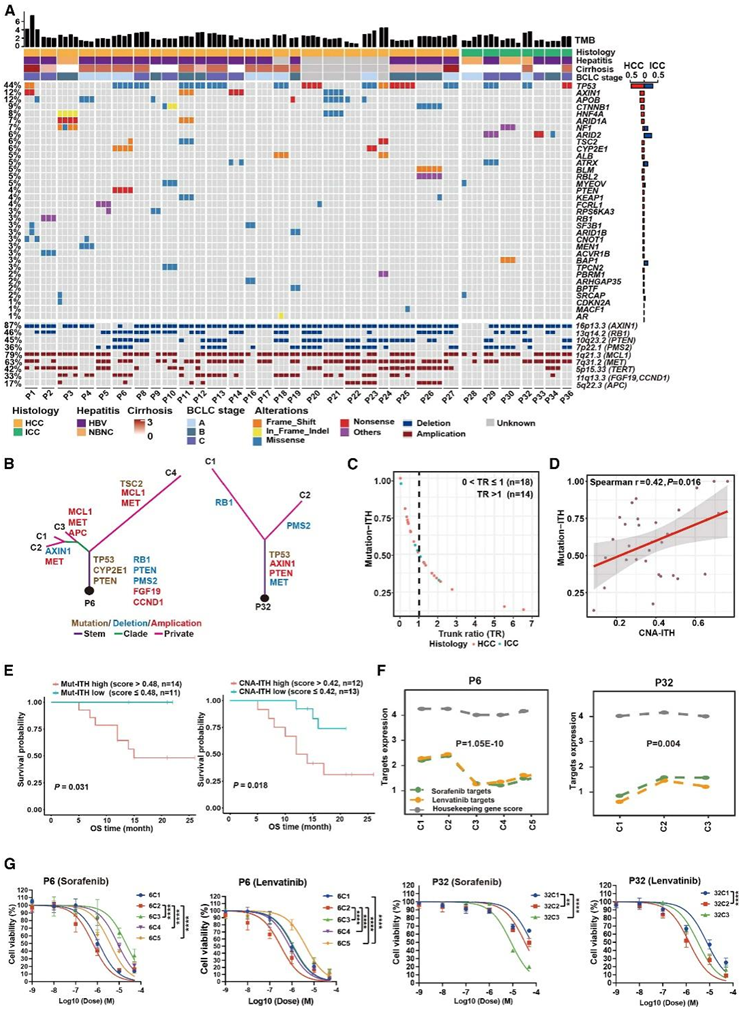
图2. 多区域类器官分析表征了基因组 ITH 和相关功能含义。
(A) 来自 32 名患者的多区域采样的 95 个 PLC 类器官的基因组图谱。(B) 表示具有潜在驱动事件的两名代表性患者的系统发育树。(C) 散点图描绘了 32 名患者中每人的突变主干比率 (TR),其中 12 名患者为主干优势 (TR > 1),20 名患者为分支优势 (TR ≤ 1)。(D) 散点图显示突变-ITH 和 CNA-ITH 水平之间呈正相关。(E) Kaplan-Meier 生存曲线分别说明了突变-ITH(左)和 CNA-ITH(右)与总生存的关联。(F) 源自患者 P6(5 个类器官)和 P32(3 个类器官)的类器官培养物中已知索拉非尼和仑伐替尼靶标的平均表达。(G) 索拉非尼和仑伐替尼对患者 P6 和 P32 的类器官培养物的剂量反应曲线。
03
筛选临床相关药物可预测患者反应并揭示肿瘤内药物敏感性的异质性
他们在来自 116 名患者的 376 个类器官中筛选了 7 种 PLC 相关药物(图 3A),其中包括一线药物仑伐替尼和索拉非尼,二线药物瑞戈非尼和阿帕替尼,抗 VEGFR 抗体贝伐珠单抗,以及针对具有可操作突变的 ICC 的药物,包括 pemigatinib(针对含有 FGFR2 融合/重排的胆管癌)和 ivosidenib(针对 IDH1 突变、化疗难治性胆管癌)。
他们计算了两个常见的汇总统计量,半最大抑制浓度(IC50)(图3B)和归一化曲线下面积(AUC)(图3C),观察到7种筛选药物的IC50和AUC值之间存在很强的相关性(图3D)。根据患者水平AUC对所有患者进行排序,并根据报告的临床总缓解率将每种药物分别分为敏感组和耐药组(图3E)。通过16个类器官早期和晚期的药物敏感性测试,他们发现大多数类器官(12/16)在早期和晚期对仑伐替尼治疗的敏感性几乎相同,而在P61C3、P75C2、P76C2和P133四个类器官中观察到差异。这四种 PDO 在早期传代时对仑伐替尼不敏感,在传代后期变得更加耐药。他们推测这四种类器官由非同质群体组成,在传代过程中耐药群体不断扩大。
接下来,基于对 14 名在研究期间复发并接受至少一种研究药物治疗的患者的调查,他们将基于类器官的药物敏感性结果与相应的临床反应进行比较,这些患者包括仑伐替尼、索拉非尼和阿帕替尼(图 3 F)。在接受仑伐替尼治疗的 8 名患者中,两名患者(P51 和 P112)出现完全缓解 (CR),来自所有三个区域的类器官均表现出敏感性,而另外 6 名患者则出现疾病进展,且来自至少一个区域的类器官该区域对仑伐替尼耐药。因此,临床反应似乎支持使用类器官的仑伐替尼敏感性结果(图 3 F)。与索拉非尼和阿帕替尼的类似比较也证实了类器官药物筛选的预测价值(图3F)。
此外,在研究期间,14名患者中有4名在根据mRECIST标准评估反应后经历了治疗方案的改变。例如,患者P51对索拉非尼和经导管动脉化疗栓塞治疗无反应,但后来接受仑伐替尼共5个月表现出CR(图3G)。类器官药物筛选的结果也使用类器官来源的异种移植物进行了验证(图 3 H)。来自患者 P15、P51 和 P74 的类器官移植到免疫缺陷小鼠体内,并分别接受仑伐替尼治疗。所有源自药物敏感类器官的异种移植物均显示出对仑伐替尼治疗的抑制作用。总而言之,这些结果证明了 PLC 类器官在预测患者治疗反应方面的临床应用潜力。
最后,他们利用药物筛选结果定量评估了目前临床实践中使用的七种靶向治疗药物对 PLC 患者的潜在益处。区域(类器官)水平和患者水平之间的个体敏感性和累积敏感性之间存在显著差异(图 3 I)。
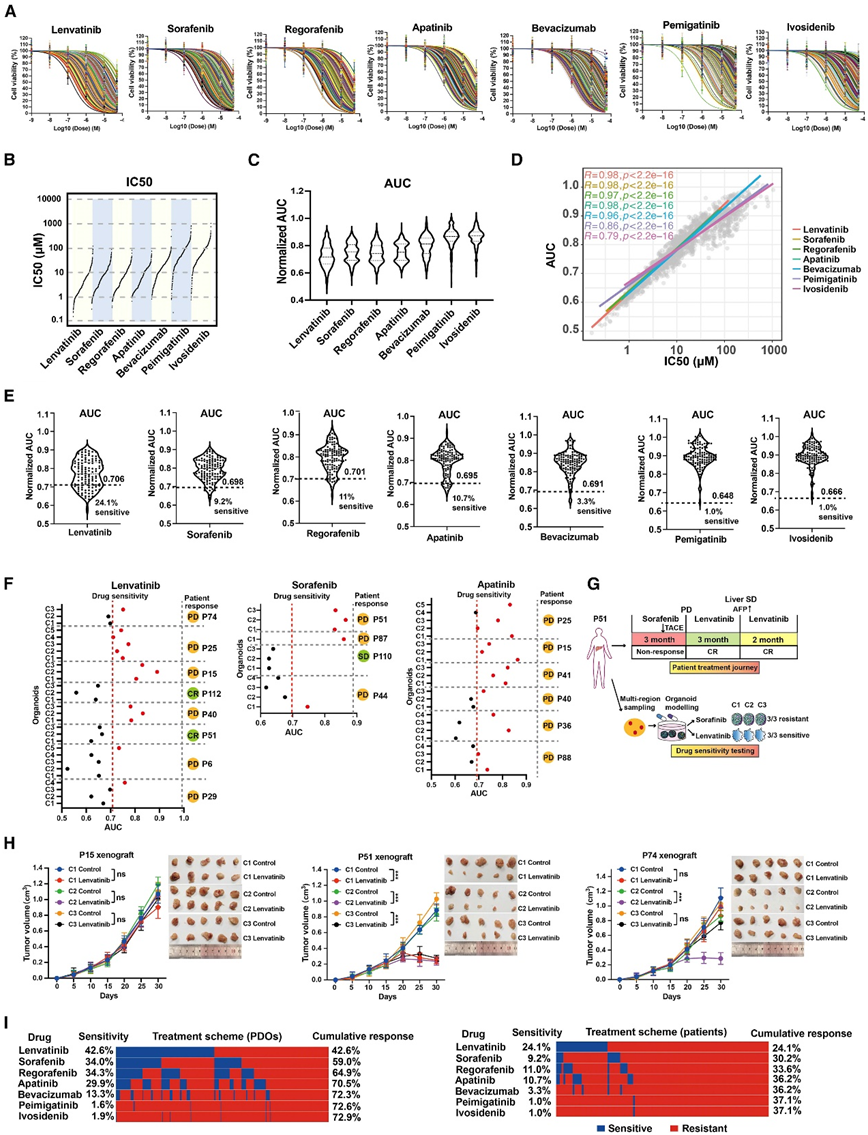
图3. 使用活体生物库进行大规模药物筛选。
(A) 七种靶向药物(索拉非尼、仑伐替尼、瑞格非尼、阿帕替尼、贝伐单抗、培米加替尼和 ivosidenib)对所有筛选的 376 个类器官(来自 116 名患者)的剂量反应曲线。(B) 散点图显示 7 种靶向药物在 376 个类器官中的 IC50 分布。(C) 小提琴图显示 376 个类器官中七种靶向药物的标准化 AUC。(D) 散点图表明七种靶向药物中每一种的 AUC 和 IC50 值之间存在显著正相关。(E) 小提琴图,显示 116 名患者中七种靶向药物的标准化 AUC。(F) 对于接受仑伐替尼/索拉非尼/阿帕替尼治疗的复发患者,个体散点图显示通过匹配类器官的药物筛选确定的 AUC 值。(G) 示意图,说明每次复发后接受指定治疗的 P51 患者过程(上),以及源自其原发肿瘤的多区域类器官的药物敏感性结果(下)。(H) 接受仑伐替尼治疗的三名患者(P15、P51 和 P74)的不同肿瘤区域的类器官来源异种移植物(左)的肿瘤生长曲线。处死小鼠后对肿瘤进行成像(右)。(I) 显示类器官生物库中七种靶向药物在 PDO 水平(左)和患者水平(右)的累积敏感性
04
确定预测仑伐替尼和其他三种药物敏感性的表达特征
他们使用多区域 PDO 研究了基因组异质性与 PLC 药物敏感性的关联。仑伐替尼耐药组患者的 ITH 和 CNA-ITH 突变水平均显著高于敏感组(图 4 A),表明 ITH 在仑伐替尼治疗耐药中发挥作用。利用 RNA-seq 和药物筛选资料前瞻性开发的两组类器官分别用于训练和验证。使用训练集,首先鉴定了254个与仑伐替尼敏感性显著相关的基因(图4B),其中三个基因(JUN 、IL1B和TNFRSF8)至关重要。他们对 TCGA-LIHC 项目中的 424 名 HCC 患者进行了聚类对四组患者进行分层。值得注意的是,一组患者普遍表现出耐药基因低表达(图4D),比例(24.86%)与仑伐替尼报道的临床疗效(24.1%)相似,表明该组可能富含对仑伐替尼治疗敏感的患者。为了确定生物标志物开发中与药物反应相关的关键基因,他们进一步应用了基于机器学习的方法,并确定了一组13个特征基因作为仑伐替尼反应的预测性生物标志物,包括JUN、HIST1H1E和WNT6A(图4E)。该多基因生物标志物在受试者操作特征(AUROC)曲线下的面积达到0.86(图4F),并且在类器官验证集(AUROC 0.81)中表现出类似的良好表现。此外,他们对其他三种PLC药物(索拉非尼、瑞戈非尼和阿帕替尼)应用了相同的分析程序,并开发了索拉非尼治疗反应的预测性生物标志物(训练集和验证集的AUROC >0.9)(图4G-K)。
最后,他们利用在研究期间接受所研究的PLC药物治疗的患者和多区域类器官分析来评估所开发的生物标志物的临床效用,其中7名接受仑伐替尼治疗的患者中有6名在特征预测和临床反应之间显示出一致的结果(图4L),而索拉非尼治疗的患者则完全一致(图4L)。总之,他们开发并验证了多基因表达特征,预测 PLC 临床实践中使用的四种抗肿瘤药物的反应,为未来的临床研究(包括生物标志物引导的试验)提供了保证。
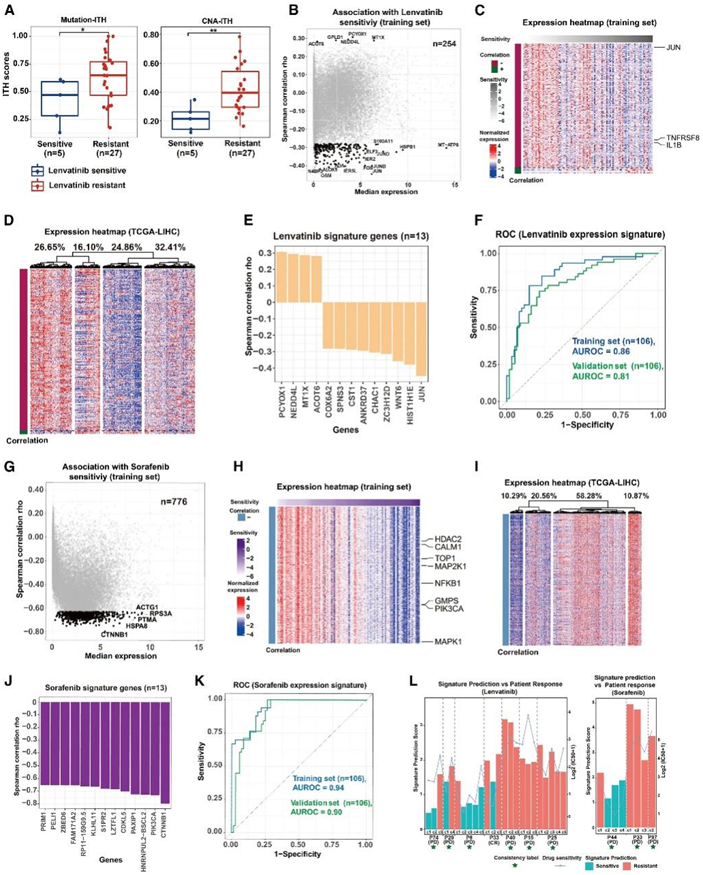
图4. 分子分析确定的表达特征可预测临床相关药物的敏感性。
(A) 箱线图比较仑伐替尼敏感组和耐药组之间的基因组 ITH 水平。(B) 蛋白质编码基因的散点图,横轴显示中位基因表达,纵轴显示表达与药物敏感性的相关性。(C) 热图显示了这 254 个基因的标准化表达,样本按药物敏感性排序,并标记了 FDA 批准药物的靶标。(D) 热图显示基于这 254 个基因表达的 TCGA-LIHC 样本的无监督聚类。(E) 条形图显示预测仑伐替尼反应的 13 个特征基因的表达与药物敏感性的相关性。(F) 训练组和验证组中 13 基因仑伐替尼预测特征的 ROC 曲线。(G–K) 是预测索拉非尼反应的表达特征 (B–F) 的对应物,其中 776 个基因与索拉非尼敏感性显著相关,13 个基因确定为索拉非尼特征基因。(L) 条形图显示了对复发患者的仑伐替尼特征(左)和索拉非尼特征(右)的预测。
05
c-Jun介导的仑伐替尼耐药
为了识别仑伐替尼耐药基因,他们使用与仑伐替尼敏感性显著相关的基因进行了蛋白质-蛋白质相互作用(PPI)网络分析(图5A)。值得注意的是,JUN是该 PPI 网络的枢纽蛋白,是仑伐替尼反应的 13 个特征基因之一,也是 FDA 批准的药物的靶点。JUN的基因表达与仑伐替尼治疗的耐药性呈正相关,如 IC50 值所示(图5B)。通过免疫组织化学染色评估,在仑伐替尼耐药类器官的 216 个区域中,c-Jun 的蛋白水平也显著高于敏感类器官的 160 个区域(图 5 C)。此外,在 100 名患者的肿瘤中发现 c-Jun 表达高于配对的癌旁组织(图 5D)。总之,高水平的 c-Jun 与 PLC 的肿瘤发生和仑伐替尼耐药有关。
他们进一步进行了功能分析,使用一名 HCC 患者 (P20) 和一名 ICC 患者 (P94),其来自不同区域的类器官对仑伐替尼治疗表现出不同的反应。与敏感类器官(P20C3、P20C4 和 P94C1)相比,仑伐替尼耐药类器官(P20C1、P20C2 和 P94C2)中 c-Jun 水平升高(图 5 C)。接下来,他们敲低了 P20C2 和 P94C2 类器官中的 c-Jun,发现 c-Jun 敲低可以使仑伐替尼耐药的类器官变得敏感(图 5 E)。相反,P20C3和P94C1中c-Jun的过度表达使它们对仑伐替尼治疗产生耐药性(图5F)。
为了系统地评估 c-Jun 在广泛的 PLC 患者中对仑伐替尼耐药的作用,他们进一步敲低了 216 个仑伐替尼耐药类器官中的 c-Jun,其中 63 个(29.2%)转化为敏感(图 5 G)。采用了三种c-Jun抑制剂(藜芦胺、SR11302和NY2267),发现先前耐药类器官对仑伐替尼治疗的敏感性增加,分别增加了17.1%、17.1%和13.4%(图5H) 。对整个类器官生物库的进一步分析表明,最多 37.1% 的患者可能对仑伐替尼和三种 c-Jun 抑制剂之一的联合治疗敏感(图 5 I)。总之,这些结果表明高水平的 c-Jun 可能导致 PLC 中的仑伐替尼耐药。
为了揭示 c-Jun 介导的仑伐替尼耐药性背后的信号传导,他们使用与仑伐替尼反应显著负相关的 245 个基因进行了功能富集分析,发现 Wnt 和 c-Jun N 末端激酶 (JNK) 信号通路显著失调(图 5 J)。β-catenin(一种关键的Wnt信号调节因子)和c-Jun的表达在仑伐替尼耐药类器官中呈正相关(图5K)。他们继续使用P20 (HCC)和P94 (ICC)类器官进行功能研究。c-Jun和β-catenin在仑伐替尼耐药类器官(P20C2和P94C2)中均呈高水平,而在敏感类器官(P20C3和P94C1)中呈低水平(图5K)。P20C2和P94C2类器官中β-catenin的敲低降低了c-Jun蛋白水平(图5L),增加了对仑伐替尼治疗的敏感性(图5M)。他们进一步筛选了所有仑伐替尼耐药类器官,发现18.5%的类器官对β-catenin敲低敏感(图5N)。相比之下,野生型和突变型β-catenin(S33A/S37A/T41A/S45A,这些磷酸化受体位点的突变稳定了β-catenin,使其能够积累、转运到细胞核,并激活WNT信号传导)的过度表达显著降低了两种敏感类器官对仑伐替尼治疗的敏感性(图5O)。这些结果共同表明,β-catenin是仑伐替尼耐药类器官中 c-Jun 的关键调节因子。
然而,β-catenin的敲低仅部分解释了c-Jun敲低后类器官对lenvatinib治疗的致敏(图5G-N)。他们继续研究另一种失调的信号:JNK。在仑伐替尼耐药区域,JNK和c-Jun的表达也呈正相关(图5P)。由于P20和P94类器官JNK表达水平较低,他们选择P103和P10作为研究对象,因为它们的多区域类器官JNK表达水平不同,并且β-catenin敲除没有任何益处。仑伐替尼耐药类器官P103C2和P10C1的JNK水平高于敏感类器官P103C3和P10C2(图5P),JNK敲低显著增加了这两种耐药类器官对仑伐替尼治疗的敏感性(图5Q)。此外,使用类器官生物库系统敲除JNK显著提高了所有216个仑伐替尼耐药类器官中的28个对仑伐替尼的敏感性(图5R)。相比之下,在P103C3和P10C2类器官中过表达野生型或组成活性形式的JNK (JNKK2-JNK)可显著降低仑伐替尼的敏感性(图5S)。总之,JNK 和 Wnt/β-catenin 可能作为 c-Jun 的上游调节因子介导仑伐替尼耐药。
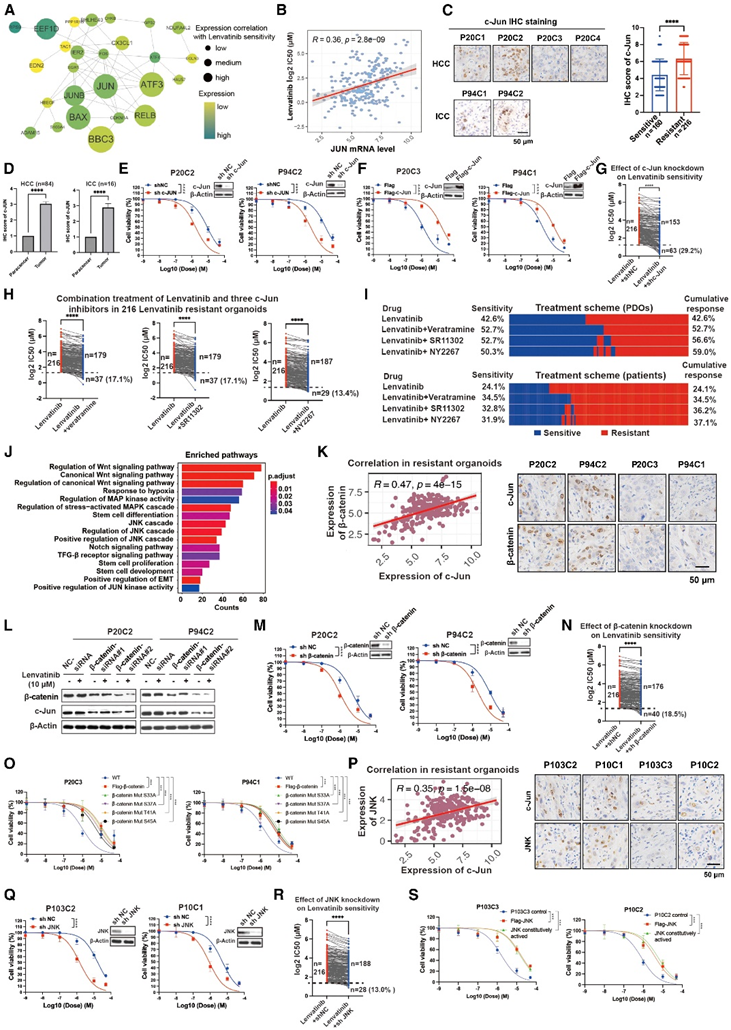
图5. c-Jun鉴定为仑伐替尼耐药的介质。
(A) 由仑伐替尼耐药蛋白组成的相互作用网络。(B) 散点图显示所有 255 个类器官中仑伐替尼耐药水平与JUN mRNA 表达呈正相关。(C) 两名代表性患者 P20 (HCC) 和 P94 (ICC) 中 c-Jun(左)的 IHC 染色。对肿瘤组织所有区域的 c-Jun 进行 IHC 定量(右)。(D) 直方图表示 HCC 和 ICC 组织中的 c-Jun 蛋白水平比配对的癌旁样本(100 对)显著增加。(E) 两种仑伐替尼耐药类器官(P20C2 和 P94C2)中 c-Jun 敲低(sh c-Jun)后仑伐替尼的剂量反应曲线。(F)在两种仑伐替尼敏感的类器官(P20C3 和 P94C1)中,仑伐替尼对 c-Jun 过表达(Flag- c -Jun)的剂量反应曲线。(G) 散点图显示 216 个仑伐替尼耐药类器官中 c-Jun 敲低后仑伐替尼的 IC50 变化。(H) 散点图显示,在 216 个仑伐替尼耐药类器官中,仑伐替尼分别与藜芦胺/SR11302/NY2267(1:1 比例)联合治疗时的 IC50 变化。(I)显示类器官生物库中仑伐替尼和藜芦胺/SR11302/NY2267联合治疗在 PDO 水平(上)和患者水平(下)的累积敏感性。(J) 富含与仑伐替尼敏感性显著负相关的基因的通路。(K) 散点图(左)显示代表区域(P20C2、P94C2、P20C3 和 P94C1)中 c-Jun 和 β-catenin 的 mRNA 水平与两种蛋白质(右)的 IHC 染色之间的正相关性。(L) 免疫印迹表明 siRNA 介导的 β-连环蛋白敲低后 c-Jun 蛋白表达减少。(M) 两种仑伐替尼耐药类器官(P20C2 和 P94C2)中 β-连环蛋白敲低(sh β-catenin)后仑伐替尼的剂量反应曲线。(N) 散点图显示 216 个仑伐替尼耐药类器官中 β-连环蛋白敲低后仑伐替尼的 IC50 变化。(O) 在两种仑伐替尼敏感的类器官(P20C3 和 P94C1)中过度表达 β-连环蛋白(Flag-β-catenin)和突变型 β-连环蛋白(S33A/S37A/T41A/S45A)时仑伐替尼的剂量反应曲线。(P) 散点图(左图)显示 c-Jun 和 JNK 的 mRNA 水平与代表性区域(P103C2、P10C1、P103C3 和 P10C2)中两种蛋白质的 IHC 染色(右图)之间呈正相关。(Q) 两种仑伐替尼耐药类器官(103C2 和 10C1)中 JNK 敲低(sh JNK)后仑伐替尼的剂量反应曲线。(R) 散点图显示 216 个仑伐替尼耐药类器官中 JNK 敲低后仑伐替尼的 IC50 变化。(S) 在两种仑伐替尼敏感的类器官(P103C3 和 P10C2)中过度表达 JNK (FLAG-JNK) 和 JNK 的组成型活性形式 (JNKK2-JNK) 时仑伐替尼的剂量反应曲线。
06
化合物 PKUF-01对仑伐替尼耐药的类器官表现出协同作用
由于仑伐替尼和 c-Jun 抑制剂所揭示的协同作用,他们设计并合成了六种连接仑伐替尼和藜芦胺的化合物(三种 c-Jun 抑制剂中疗效最好),这使得能够针对相同的癌症细胞同时进行,以获得更好的组合治疗效果。一种命为 PKUF-01的化合物(图 6A),可以有效阻断 c-Jun 和 FGFR(图 6B),并显示出良好的抑制功效(图 6C)。当用类器官生物库进一步评估 PKUF-01 时,与仑伐替尼治疗相比,敏感性显著提高(以 IC50 值下降表示)(图 6D)。特别是,对于来自90名患者的216个仑伐替尼耐药类器官,PKUF-01在20.0%的这些类器官中诱导了显著反应(图6E),与两种单独药物(藜芦胺和lenvatinib)联合治疗相比,PKUF-01在仑伐替尼耐药类器官中有2.9%的增加。PKUF-01在类器官移植异种移植模型中进一步检测其疗效。仑伐替尼或藜芦胺治疗未能抑制三种类器官来源的异种移植模型的肿瘤生长。相比之下,PKUF-01治疗可显著抑制肿瘤生长(图6F)。
此外,他们应用上述基于机器学习的方法并开发了用于预测 PKUF-01 反应的多基因特征。首先,发现 205 个基因的表达与 PKUF-01 敏感性显著相关,并且可以使用这 205 个基因将 TCGA-LIHC 数据集中的肿瘤分为四组。随后开发了由 17 个特征基因组成的生物标志物,其中也包括 c-Jun(图 6 G)。该特征在训练集中的 AUROC 为 0.836,在验证集中的 AUROC 为 0.731(图 6 H)。此外,基于转录组谱的 GSEA 分析,比较了对 PKUF-01 治疗具有不同敏感性的类器官组(图 6 I),他们发现胚胎器官发育相关基因集和与干细胞相关的基因集在耐药类器官中上调(图6J);因此,针对干性可能是克服 PKUF-01 治疗耐药性的潜在策略。
最后,通过分析整个 PLC 类器官生物库, PKUF-01 比单独使用仑伐替尼治疗 PLC 的疗效显著提高,并且可以使 PLC 患者受益增加(图 6K)。加上临床实践中使用的七种经过筛选的药物,靶向治疗总共可使 48.3% 的 PLC 患者受益(图 6 K)。综上所述,仑伐替尼可以靶向 FGFR 和其他受体,减弱下游信号传导,从而抑制肿瘤生长;然而,受 Wnt/β-catenin 和 JNK 通路调节的 c-Jun 会导致仑伐替尼耐药。PKUF-01 靶向 c-Jun 在仑伐替尼耐药类器官中显示出显著疗效,并可能使更多 PLC 患者受益(图 6 L)。
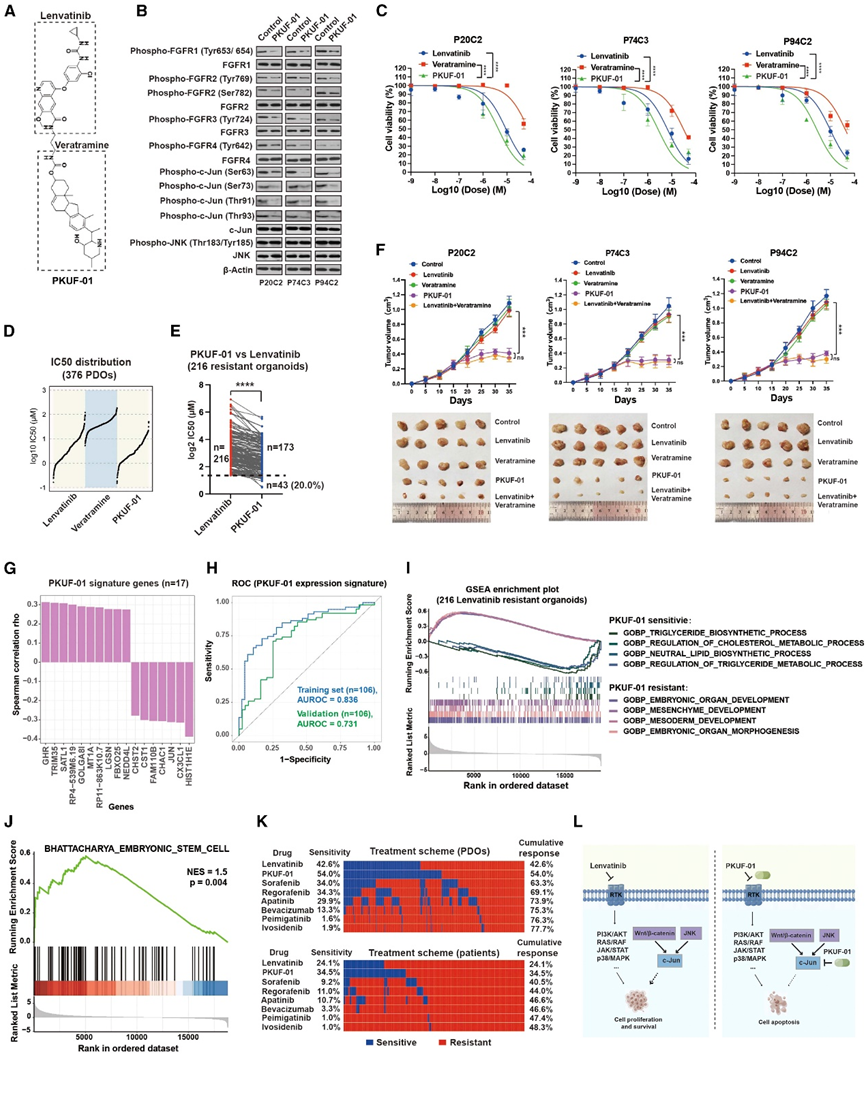
图6. 化合物PKUF-01增加仑伐替尼耐药类器官的敏感性。
(A) 连接仑伐替尼和藜芦胺的PKUF-01 的配方。(B) 免疫印迹分析。(C) 仑伐替尼、藜芦胺和 PKUF-01 对三种仑伐替尼耐药类器官(P20C2、P74C3 和 P94C2)的剂量反应曲线。(D) 散点图显示仑伐替尼和 PKUF-01 在所有筛选的 376 个类器官中的 IC50 分布。(E) 散点图显示 216 个仑伐替尼耐药类器官中仑伐替尼和 PKUF-01 的 IC50 值。(F) 三种类器官来源的异种移植物(P20C2、P74C3 和 P94C2)单独使用仑伐替尼/藜芦胺/PKUF-01 治疗或仑伐替尼和藜芦胺联合治疗的肿瘤生长曲线(上)。处死小鼠后对肿瘤进行成像(下)。(G) 条形图显示预测 PKUF-01 反应的 17 个特征基因的表达与药物敏感性的相关性。(H) 预测训练集和验证集中 PKUF-01 反应的 17 个基因特征的 ROC 曲线。(I) GSEA 富集图显示所选基因组在 PKUF-01 未能致敏的仑伐替尼耐药类器官中发生显著变化。(J) 与干细胞相关的基因集的 GSEA 富集图。(K) 定制图表显示类器官生物库中七种已批准的靶向药物和 PKUF-01 在 PDO 水平(上)和患者水平(下)的累积敏感性。(L) 提出的 c-Jun 介导的仑伐替尼耐药模型。
+ + + + + + + + + + +
结 论
本项研究建立了一个 PLC 生物库,由来自 144 名患者的 399 个肿瘤类器官组成,它概括了亲本肿瘤的组织病理学和基因组图谱,并且对于药物敏感性筛选是可靠的,正如体内模型和患者反应所证明的那样。综合分析剖析了 PLC 异质性,涉及基因组/转录组特征和对七种临床相关药物的敏感性以及临床关联。药物基因组学分析可识别并验证多基因表达特征,预测药物反应,从而更好地对患者进行分层。此外,发现 c-Jun 通过 JNK 和 β-catenin 信号传导是仑伐替尼耐药的主要介质。合成并筛选了包含仑伐替尼和藜芦胺(c-Jun抑制剂)部分的化合物(PKUF-01),表现出显著的协同作用。总之,这项研究描述了 PLC 异质性的特征,开发了预测性生物标志物组,并确定了联合治疗的仑伐替尼耐药机制。
+ + + + +



 English
English


