文献解读|Nat Cell Biol(21.3):联合表观基因组分析揭示了人类皮质类器官中细胞类型特异性基因调控程序
✦ +
+
论文ID
原名:Joint epigenome profiling reveals cell-type-specific gene regulatory programmes in human cortical organoids
译名:联合表观基因组分析揭示了人类皮质类器官中细胞类型特异性基因调控程序
期刊:Nature Cell Biology
影响因子:21.3
发表时间:2023.11.23
DOI号:10.1038/s41556-023-01296-5
背 景
基因表达受到多种表观遗传机制的调节,这些机制在发育和疾病中相互协调。然而,当前的多组学方法通常一次仅限于一种或两种模式,这使得获得全面的基因调控特征具有挑战性。本项研究开发了一种方法-3D 基因组、染色质可及性和甲基化测序 (3DRAM-seq) ,该方法可以在全基因组范围内以高分辨率同时分析空间基因组组织、染色质可及性和 DNA 甲基化。
实验设计
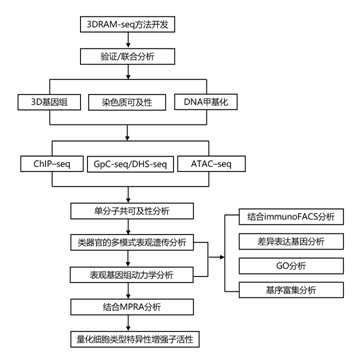
结 果
01
3DRAM-seq 的开发和验证
研究者推断,通过将大量固定的细胞核与GpC甲基转移酶M.CviPI结合酶处理,可以同时测量染色质可及性、DNA甲基化和3D基因组(图1a,图S1a)。此外,CTCF和转录起始位点(TSS)具有预期的可及性和DNA甲基化模式(图1b,图S1b-f)。重要的是,3DRAM-seq 的单核苷酸分辨率能够可视化 NRF1 等转录因子(TF) 的基序足迹,这与其作为同源二聚体的结合一致(图1c,图S1g)。为了能够分析转录组和表观基因组,他们还优化了从固定细胞中回收高质量RNA的方法。基因表达具有重复性高、覆盖均匀等特点(图S1h-i)。3D 基因组组织在重复中具有高度可重复性(图S1j–l),其特点是接触概率随距离而下降(图S1k),跨 TAD 边界的绝缘(图 1d)和与结合CTCF位点相关的染色质环(图1e)。在Sox2位点,他们观察到增强子和启动子的可及性增加和低DNA甲基化,以及连接这两个元件的染色质环的存在(图1f-g)。

图1. 3DRAM-seq 可对 3D 基因组组织、染色质可及性和 DNA 甲基化进行联合分析。
(a) 3DRAM-seq 的示意图。 (b) 以基序为中心和定向 CTCF ChIP-seq 峰(5 bp bin)的平均 CpG 甲基化和 GpC 可及性水平。(c) 与(b)相同,但针对以基序为中心的 NRF1 ChIP–seq 峰(2 bp bin)。(d) TAD 的平均接触富集、DNA 甲基化和 GpC 可及性水平。(e) ChIP-seq 峰内会聚 CTCF 基序之间的聚合接触富集。(f-g) 接触图和基因组轨迹显示了Sox2基因座的 DNA 甲基化、GpC 可及性、ATAC-seq、H3K27ac ChIP-seq 和 RNA-seq。
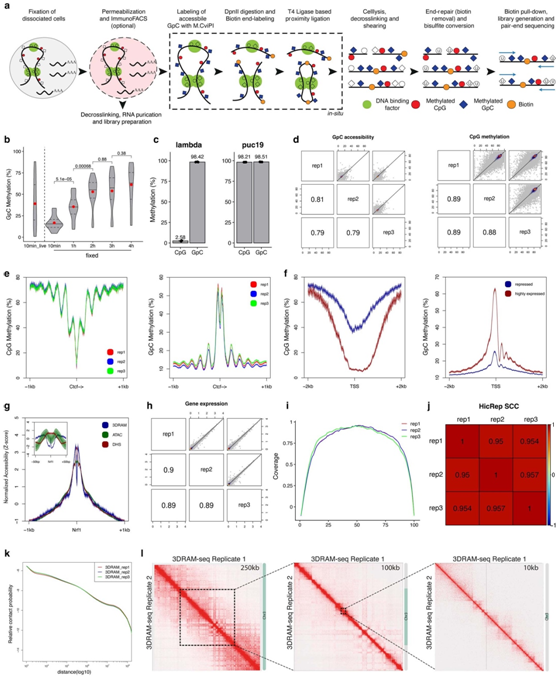
图S1. 3DRAM-seq 实验概述和其他指标分析。
(A) 3DRAM-seq 实验流程。(B) 显示GpC 甲基化水平的小提琴图。(C) 显示lambda 甲基化水平(仅 GpC 甲基化)和完全甲基化 puc19 DNA 标记对照的条形图。(D) 成对相关矩阵显示 Pearson 相关系数以及 1 kb bin 中 GpC 可及性和 DNA 甲基化的散点图。(E) 单个重复的以基序为中心的 Ctcf ChIP-seq 峰的平均 CpG 甲基化和 GpC 可及性水平。(F) 以 TSS 为中心的抑制基因或高表达基因的平均 CpG 甲基化和 GpC 可及性水平。(G) 以基序为中心的 Nrf1 ChIP-seq 峰(2 bp bin)的平均可及性水平。(H) 显示相关系数 (Spearman) 的成对相关矩阵,以及基因表达的散点图。 (I) 由 RSeQC 计算的管家基因中每次重复的 RNA-seq 读取覆盖率。 (J) 显示 3D 基因组相关系数的成对相关矩阵(层调整相关系数,10 kb bins,由 HiCRep 计算)。 (K) 对数箱中的接触概率。 (L)重复 1 和 2 的 3 号染色体接触图。
02
3DRAM-seq 生成高质量的表观基因组数据
接下来,他们重点关注染色质可及性,基于GpC甲基化鉴定了67177个可及区域(称为GpC峰),这些区域与转座酶可及染色质测序(ATAC-seq)和DNA酶I超敏位点测序(DHS-seq)鉴定的峰基本一致(图2a),其特征是可及性和核小体相位增加(图2b-c)。
3DRAM-seq的特点是具有高覆盖率和高分辨率的结合CTCF基序(图2d)。3DRAM-seq还具有高比例的唯一映射读取和总接触,并具有高顺反比(图2e)和远距离依赖性接触轮廓、区室和环的特征(图2f-g)。
这些结果表明,3DRAM-seq可以联合测量所有三种表观遗传模式和基因表达,具有较高的重现性、覆盖范围和数据质量。
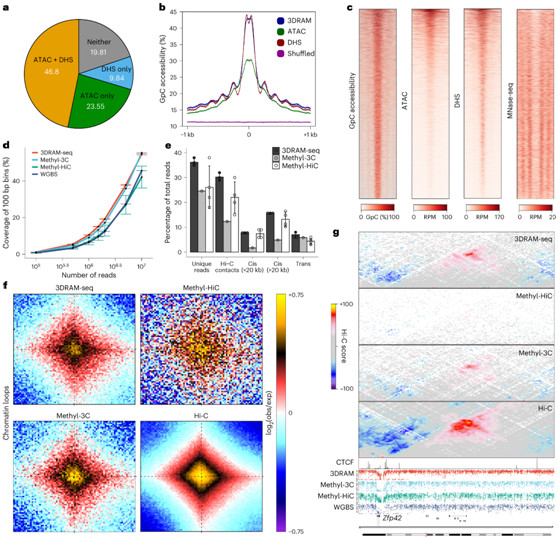
图2. 3DRAM-seq 与其他多组学方法的比较。
(a) 饼图描绘了 3DRAM-seq GpC 可及峰仅与 ATAC-seq 峰重叠、仅与 DHS 峰重叠、与 ATAC 和 DHS 重叠或两者都不重叠的百分比。(b) 3DRAM-seq GpC-seq、ATAC-seq、DHS-seq 或混合区域(5 bp bin)的平均 GpC 可及性水平。(c) 热图显示通过 GpC 甲基化、ATAC-seq、DHS以及核小体占用(MNase-seq)的可及性数据。(d) 不同方法的覆盖率比较。(e) 3DRAM-seq、Mmethyl-3C 和 Mmethyl-HiC 的测序统计。 (f) 使用 HICCUP 5 kb 分辨率在原位 Hi-C 数据。(g) 不同方法的接触图和 DNA 甲基化模式的比较,以及 CTCF ChIP-seq 轨迹。
03
3DRAM-seq 支持单分子共可及性分析
他们接下来制定了一种策略来量化单分子水平上的共可及性或共甲基化程度。传统的方法限制在几百个碱基对之间,或者使用长读长测序时限制在几千个碱基对之间。相比之下,3DRAM-seq可用于分析相隔很远的区域(图3a)。
他们首先关注与染色质免疫共沉淀测序(ChIP-seq)测定的CTCF峰重叠的CTCF结合基序,发现19%是可及的(图3b-c),而在随机对照中,5%是可及的,对于随机对照,只需一次读取即可重叠CTCF位点。然而,两个CTCF位点同时共同可及的概率并不高于偶然预期(图3b-c)。重要的是,基于平均可及性,所有区域都是开放的,并且受到CTCF和SMC1约束,这表明单个细胞中局部表观遗传景观的快速更新(图3d-e)。
最后,他们分析了顺式调控元件(CRE)-TSS对。与CTCF一样,无论是在单分子水平(图3f-g)还是在整体水平(图3h-i),他们均未观察到这些区域对染色质可及性的任何协同效应,这表明染色质可及性也受到局部调节。
总体而言,这些结果展示了 3DRAM-seq 量化基因组距离较大的区域对的单分子共可及性的能力,并表明,染色质可及性和 DNA 甲基化的变化主要是局部事件,通常不受其他基因组区域邻近程度的影响。

图3. 3DRAM-seq 能够以单分子分辨率进行配对共可及性测量。
(a) 单分子共可及性测定的示意图。 (b) 以至少 1 kb 间隔的会聚 CTCF 基序为中心的 100 bp 窗口中的聚类平均配对共可及性水平。(c) 与(b)相同,但显示每次读取中各个 GpC 二核苷酸的甲基化状态。(d) 对于包含 CTCF 正向基序的相同 read 1 区域,±500 bp 窗口中的平均 CTCF ChIP-seq 信号和 GpC 可及性水平。(e) 箱线图显示 CTCF 或 SMC1的ChIP-seq 信号。(f-g) 与b和c相同,但对于读取 1 上包含 CRE(定义为远端开放彩色区域)和读取 2 上包含 TSS 的读取对。(i) 箱线图在以 CRE 或 TSS 为中心的 ±250 bp 窗口中显示 ATAC-seq 和 H3K27ac ChIP-seq 信号。
04
人类皮质类器官的多模态表观遗传重组
为了分析人类大脑发育的调控动态,他们将 3DRAM-seq 与immunoFACS结合起来,从人皮质类器官中纯化胶质细胞 (RGC)和中间祖细胞(IPC)。他们首先关注的是基因表达,在IPC中,RGC特异性基因如SOX2、HES1和PAX6下调,参与神经元分化的基因如EOMES和NEUROG2上调(图4c),GO分析显示神经元分化和细胞形态等通路发生上调(图4d)。DNA甲基化和ctcf可及基序或ChIP-seq峰的可及性符合预期模式(图4e-f)。
他们发现细胞类型之间的整体染色质组织在远距离相互作用,拓扑关联域(TAD)和CTCF环的水平上是相似的(图4g-h)。相反,特定的调控相互作用,如在SOX2位点,是动态的,并且与其假定的增强子的可及性丧失相关(图4i)。
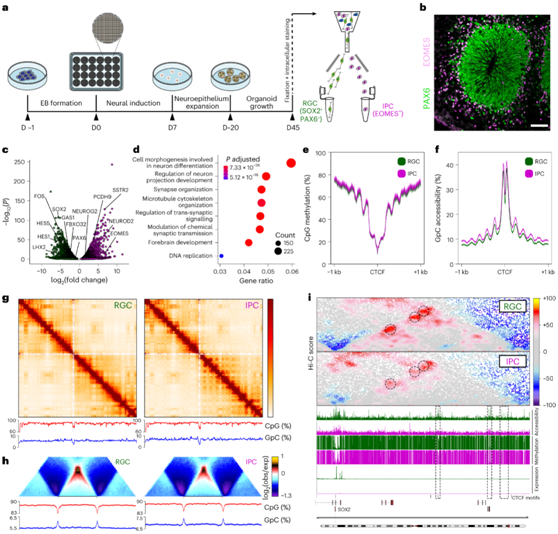
图4. 将3DRAM-seq与immunoFACS相结合,可以对人类皮质类器官中细胞类型特异性表观遗传景观进行多模式分析。
(a) 人类皮质类器官生成的实验概述。(b) 第 45 天皮质类器官内形成的神经玫瑰花结的代表性免疫荧光图像。(c) 散点图显示RGC-to-IPC分化中显著上调或下调的基因。 (d)GO分析。(e-f) CpG 甲基化和 GpC 可及性水平。(g) 3 号染色体(200 kb bin)的接触图(上)以及 CpG 甲基化和 GpC 可及性水平(下)。 (h) RGC 和 IPC 在 TAD 上的平均接触富集、CpG 甲基化和 GpC 可及性水平。 (i) SOX2基因座上 RGC 和 IPC 的接触图、GpC 可及性、DNA 甲基化和表达水平。
接下来,他们研究了rgc向ipc转变后表观遗传景观的动态。在rgc到ipc的转变过程中,确定了19316个差异可及性位点(图5a)。如预期的那样,IPC 的差异可及区域(DAR)在可及性升高后发生去甲基化(图5b),而RGC DAR的DNA甲基化没有变化。与DAR相互作用的基因在神经分化、细胞形态发生和迁移等方面表现出富集(图5c)。
为了确定这些表观基因组变化的机制,他们进行了基序富集分析,SOX2、LHX2和FOS-JUN(也称为AP-1)在RGC dar中富集(图5d),而EOMES、NFIA和神经源性bHLH tf如NEUROG2或NEUROD1在IPC dar中富集(图5f)。
接下来,他们研究TF结合是否与动态染色质环相关。LHX2(对前脑发育和RGC增殖很重要的TF)与RGC特异性调控环相关(图5e)。这些结果与含有neurog2基序的IPC dar相反,后者显示IPC中连通性增加和DNA甲基化水平降低(图5g)。
GAS1基因座在RGC中高度表达,并且有几个含有lhx2的远端dar,在IPC中可及性的丧失伴随着与GAS1启动子的弱相互作用(图5h)。在含有多个NEUROG2基序并在IPC中上调的NFIA位点,他们观察到相反的模式:染色质可及性增加和更强的相互作用(图5i)。他们观察到两种富含rgc的tf:SOX2和LHX2,在rgc中具有高共可及性,而在IPC中则没有(图5j)。相反,NEUROG2和EOMES基序主要在IPC中共同可及,而在RGC中则不能。这些发现与之前的研究结果一致,SOX2-LHX2和NEUROG2-EOMES分别可以直接作用于RGC和IPC的染色质或共结合。
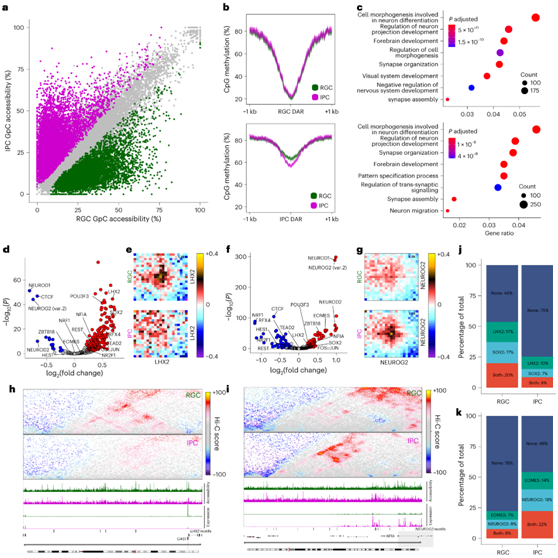
图5. 与皮质类器官表观基因组重塑相关的转录因子。
(a) 散点图显示 RGC 和 IPC 中各个 GPC 峰的 GPC 可及性水平。 (b)平均 CpG 甲基化水平。 (c) GO分析。(d) 火山图显示 RGC DAR 中 TF 基序的富集。(e) 含有 LHX2 基序的 RGC DAR 对的接触富集。 (f-g) 与(d-e)相同,但分别适用于 IPC DAR 和具有 NEUROG2 基序的 IPC DAR。 (h-i) GAS1和NFIA基因座的接触图、GpC 可及性水平以及 RGC 和 IPC 的基因表达。(j) 包含与 RGC 峰重叠的 LHX2 和/或 SOX2 基序的配对读数(间隔 100-300 bp)的单分子共可及性水平。 (k) 包含与 IPC 峰重叠的 EOMES 和/或 NEUROG2 基序的配对读数(间隔 100-300 bp)的单分子共可及性水平。
05
类器官中转座元件的表观基因组动力学
为了研究转座元件 (TE) 对人类大脑发育中表观遗传重组的贡献,他们首先关注染色质循环。接下来,他们确定了两类te:MER130和UCON31,它们在ipc中更可及(图6a-d)。然而,这两类TE总体上仍然高度甲基化(图6b-d),这表明这两种模式存在部分解偶联效应。为了确定调节这两个 TE 类别的因子,他们进行了基序分析,发现神经源性 TF 大量富集,例如 NEUROG2、NFIA 和 NFIX,这些神经源性 TF 与 IPC 分化有关(图6e-f)。他们发现与神经元分化和成熟或大脑发育相关的通路显著富集(图6g),但没有证据表明存在差异基因表达(图6h)。
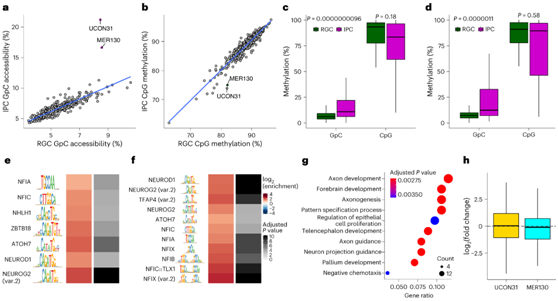
图6. MER130 和 UCON31 重复元件与可及性的变化相关,并富含神经源性 TF 基序。
(a-b) 散点图显示不同类别重复基因组元件的可及性或 DNA 甲基化水平。 (c-d) 代表 MER130和 UCON31 重复序列的 GpC 可及性和 DNA 甲基化水平的箱线图。 (e-f) TF 基序富集 MER130和 UCON31 重复元件。 (g) GO分析。(h) 箱线图显示与 UCON31或 MER130相互作用的基因中的基因表达变化。
06
人皮质类器官细胞类型特异性MPRA
为了剖析所识别的 DAR 是否可以驱动基因表达,他们将大规模并行报告基因检测 (MPRA)应用于皮质类器官,将电穿孔与immunoFACS 结合起来,分析细胞类型特异性调控(图7a)。他们在MPRA中纳入了5876个序列(500个对照),回收率>98%(图7b)。值得注意的是,VISTA增强子重叠的区域比对照更活跃,并且显著活跃的cre的活性与其基于细胞类型特异性可及性的分类相关(图7c)。RGC活性与多个已知在皮质发育中起作用的TF家族相关,如SOX和NR2F2(也称为COUP-TF1)。其次,他们将显著活跃的增强子聚集在一起(图7d-e),发现SOX和神经元bHLH基序分别在RGC或IPC聚类中富集。为了测试TF结合是否能直接影响活性,他们对选定的TF基序进行了突变。正如预测的那样,这导致了报告基因活性的大幅减少(图7f-g)。
他们重点研究了SOX2-LHX2、NEUROG2-EOMES37和NEUROD1-POU3F3等TF对,它们分别在RGC、IPC和神经元中与染色质相互作用或共结合。重要的是,具有两种基序的增强子在细胞类型特异性方面具有更高的活性(图7h-i),这表明了协同效应。这些复杂的调控作用可以在PCHD9位点上得到体现,PCHD9位点在IPC中强烈上调(图4c)。这种表达的增加伴随着在其启动子和基因内增强子之间形成染色质环,从而在IPC中可及(图7j)。尽管该增强子内EOMES基序的突变强烈降低了MPRA活性,但NEUROG2基序的突变完全消除了MPRA活性(图7k),这表明在TF功能中存在潜在的层次结构。
最后,他们重点关注了FBXO32位点,该位点在胚胎第14天的小鼠皮质中不表达,但存在于人类心室RGC和类器官中(图4c,图7l)。他们观察到多个假定的增强子元件(E1-E4),它们在RGC中可及并与FBXO32启动子的染色质环有关(图7l)。基于其高MPRA活性,他们重点分析了E2 FBXO32增强子。比较基因组分析显示,人类和同源小鼠的序列中度保守(64.7%),但具有不同的预测tf结合基序。重要的是,只有人类(而不是同源小鼠)序列才能够驱动表达(图7m-n)。
总的来说,将MPRA应用于皮质类器官能够直接量化细胞类型特异性增强子活性,分析关键tf在人类神经发生中的调节重要性,并验证人类RGC的增强子。
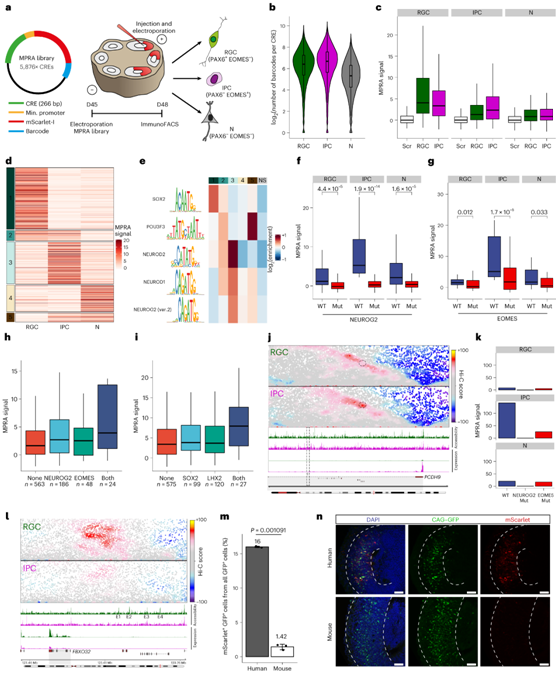
图7. 类皮质类器官中细胞类型特异性的 MPRA。
(a) 人类皮质类器官中immunoMPRA的实验概述。 (b) 小提琴图和箱线图,显示每个 CRE 获得的独特条形码的数量。(c) 箱线图显示对照和与 RGC或 IPC增强子 相关的显著活跃 CRE的 MPRA 信号。 (d) 所有细胞类型中显著活性cre的k -均值聚类。 (e) 热图显示五个重要 CRE 聚类的基序富集。 (f-g) 箱线图显示含有野生型 (WT) 或突变 (Mut) NEUROG2或 EOMES TF 基序的 IPC 中显著活跃的 CRE 的细胞类型特异性 MPRA 信号 。 (h-i) 箱线图显示包含所示基序的显著活跃的CRE在 IPC和 RGC中的 MPRA 信号。(j) PCDH9基因座上 RGC 和 IPC 的接触图、GpC 可及性和基因表达。(k) 条形图显示(j)中所示的 CRE在有或没有基序突变的情况下的 MPRA 活性。 (l) 与(j)相同,但针对FBXO32基因座。 (m) 条形图显示 通过 FACS 定量的GFP +细胞群中 mScarlet +细胞的百分比。 (n)代表性免疫荧光图像。
+ + + + + + + + + + +
结 论
本项研究开发了一种3DRAM-seq方法,并将 3DRAM-seq 与皮质类器官中的immunoFACS相结合,绘制跨多个表观遗传层的人类神经发育的细胞类型特异性调控图谱,还应用MPRA来探究类器官中细胞类型特异性增强子的活性,并从功能上评估关键转录因子对人类增强子激活和功能的作用。更广泛地说,3DRAM-seq 可用于分析稀有细胞类型和不同组织的多模式表观遗传景观。
+ + + + +



 English
English


