文献解读|Cell Rep Med(11.7):肿瘤和血浆细胞外囊泡的综合蛋白质组学分析确定了结直肠癌的诊断生物标志物
✦ +
+
论文ID
原名:Integrative proteomic profiling of tumor and plasma extracellular vesicles identifies a diagnostic biomarker panel for colorectal cancer
译名:肿瘤和血浆细胞外囊泡的综合蛋白质组学分析确定了结直肠癌的诊断生物标志物
期刊:Cell Reports Medicine
影响因子:11.7
发表时间:2025.05.01
DOI号:10.1016/j.xcrm.2025.102090.
背 景
结直肠癌 (CRC) 在癌症发病率中位居第三位,并且仍然是全球癌症相关死亡的第三大原因。早期发现 CRC 可显著提高生存率,局部病变的 5 年生存率高达 91%,而转移性病变的 5 年生存率仅为 14%。然而,虽然筛查项目取得了进展,但现有的诊断方法在可及性和可靠性方面仍然存在不足。因此,早期精准诊断的迫切需求仍未得到满足,这凸显了对非侵入性生物标志物的持续需求。此类生物标志物可以通过及时检测实现更早的临床干预并提高治疗效果。现阶段,细胞外囊泡 (EV) 已成为基于液体活检的癌症监测的有希望的候选对象。
实验设计
结 果
01
CRC 组织和血浆来源的 EV的综合蛋白质组学分析
为了系统地鉴定和验证用于CRC诊断的潜在EV蛋白生物标志物,研究团队采用了跨多个临床中心的多阶段生物标志物研究工作流程(图1 A)。在发现阶段,他们结合了组织和血浆样本中EV的蛋白质组学分析,使用基于MS的蛋白质组学和数据非依赖性采集(DIA)策略,实现了包含5603种蛋白质的深度蛋白质组学覆盖。这种整合方法鉴定出了21种CRC候选生物标志物,这些候选标志物在验证阶段使用PRM-MS进行了进一步优化。最终,他们在一个大型队列中使用ELISA验证了六种蛋白质,然后重点关注三种关键蛋白质——CTTN、HNRNPK和PSMC6——构成了ColonTrack模型。随后,他们在内部和外部队列中评估了该模型的诊断性能,证明了其潜在的临床实用性。
发现阶段包括 60 名受试者(40 名 CRC 患者和 20 名健康个体)(图 1 B)。分别使用密度梯度离心结合尺寸排阻色谱 (SEC) 柱从组织中分离 EV,并使用商业试剂盒从血浆中分离 EV。通过透射电子显微镜 (TEM)、纳米颗粒跟踪分析 (NTA) 和免疫印迹对分离的 EV 进行表征,证实了其典型的形态和大小(<200 nm)以及 EV 标志物(TSG101、CD81 和 CD9)的富集,且污染极少(无钙联蛋白)(图1C-E)。NTA 进一步表明,大多数分离的 EV 是外泌体或小微泡,直径主要在 160 nm 左右。
随后,他们使用DIA-MS蛋白质组学策略对源自组织和血浆的EV进行了蛋白质组分析。他们分别从混合样本中构建了组织EV和血浆EV的混合谱库。为了增强组织EV蛋白质组的深度和覆盖度,他们将来自多个CRC细胞系(HT29、HCT116、Lovo、SW620和SW480)的EV样本纳入组织EV谱库。混合组织EV谱库包含6949种蛋白质,血浆EV谱库包含3265种蛋白质,两个谱库共鉴定出2623种蛋白质。与Vesiclepedia数据库的比较显示,超过80%的已鉴定蛋白质包含在内,这证实了分离的EV的高纯度,并证明了本研究蛋白质组学分析的稳健性。
使用基于 DIA-MS 的非标记定量策略,他们定量了组织 EV 样本中的 5496 种蛋白质和血浆 EV 样本中的 2195 种蛋白质。其中,组织和血浆 EV 中的 2088 种蛋白质均进行了定量,这表明组织来源的 EV 可能进入血液(图1F)。定量评估显示,组织 EV 的蛋白质强度跨越 5 个数量级,血浆 EV 的蛋白质强度跨越 4 个数量级(图 1 G)。值得注意的是,在大多数血浆蛋白质组研究中,血浆中十种最丰富的蛋白质通常占总信号强度的约 80%,这凸显了蛋白质组分析对于生物标志物发现的重要性。相比之下,在本研究中的血浆 EV 蛋白质组数据中,之前报道的12种最丰富的蛋白质占总信号强度的不到 1%,这有助于进一步发现 CRC 的潜在生物标志物(图 1 G)。
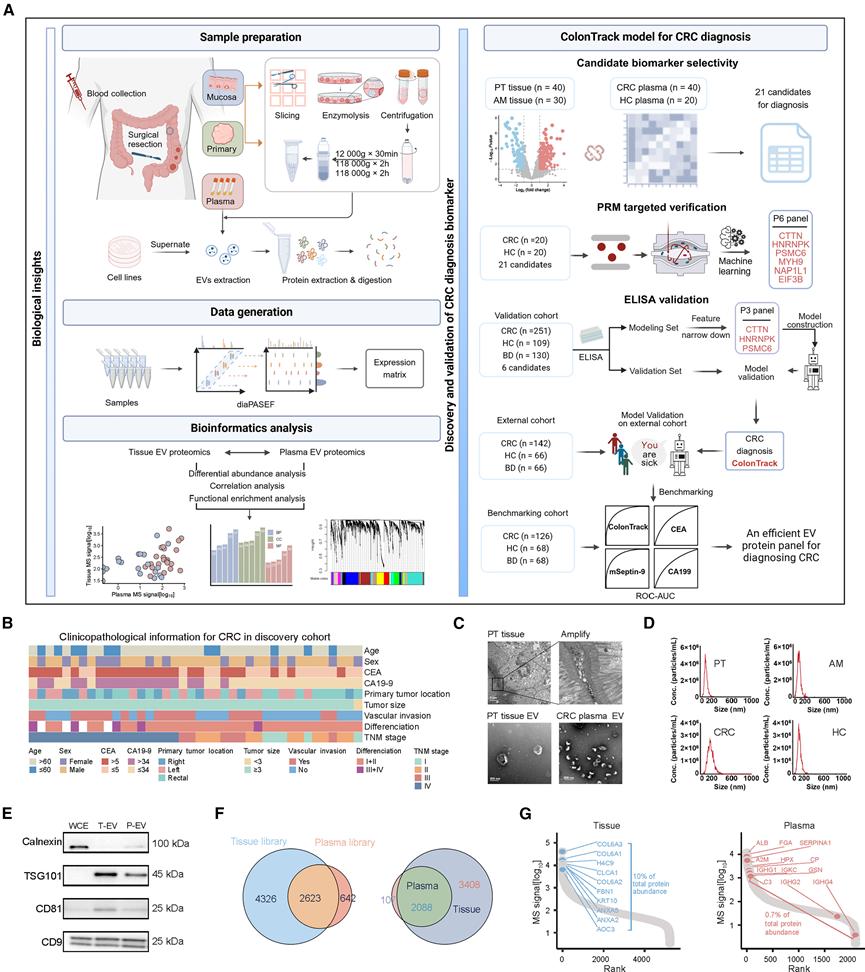
图1. 对组织和血浆来源的EV进行全面分析,以发现CRC生物标志物。
(A) 基于 EV 蛋白的结肠直肠癌生物标志物发现的工作流程。(B) 发现队列中使用的受试者的临床指标,包括 40 名 CRC 患者和 20 名健康个体。(C) 原发性肿瘤 (PT) 组织的透射电子显微镜分析。(D-E) 通过NTA和免疫印迹法对组织 EV 和血浆 EV 进行表征。(F) 两个光谱库之间已鉴定蛋白质的重叠(左)以及组织 EV 样本和血浆 EV 样本之间定量蛋白质的重叠(右)。(G) 组织EV样本(左)和血浆EV样本中定量的蛋白质按其中值强度排序。血浆中最丰富的12种蛋白质(右)。
02
组织EV蛋白质组揭示CRC中的代谢重编程
对于组织 EV 中 5556 种定量蛋白质,他们进行了加权相关网络分析 (WGCNA),并鉴定出 11 个与不同生物过程和通路相关的功能模块(图 2 A-B)。对CRC 组织来源的 EV进行全面的蛋白质组学表征表明,这些模块参与生物氧化、细胞质翻译和 mRNA 加工。值得注意的是,富含细胞呼吸过程(如氧化磷酸化和 ATP 代谢)的 ME09 模块在 PT 和 AM 样本之间存在显著差异。这些发现强调了组织来源的 EV可以反映疾病相关的生理变化,特别是在能量代谢方面,这对 CRC 的发展至关重要。
为了进一步评估 PT 和 AM 组织之间 EV 蛋白质组的差异,他们基于 Bray-Curtis 距离进行了主坐标分析 (PCoA),结果显示两组之间存在显著差异。基因集富集分析 (GSEA) 表明 CRC 相关通路在 PT 组中高度上调(图 2 C)。对 EV 蛋白质组数据进行进一步的差异分析,发现868种显著失调的蛋白质,其中 449 种上调,419 种下调(图 2 D)。这些失调的蛋白质主要参与能量代谢,包括电子传递和氧化磷酸化(图 2 E)。他们检测了失调蛋白与WGCNA模块之间的重叠情况,结果显示大多数失调蛋白归类为ME01、ME03和ME04——这些模块与能量代谢密切相关。总而言之,这些发现表明EV蛋白质组可能通过代谢重编程来调节CRC的进展。
为了进一步确定用于 CRC 诊断的 EV 蛋白生物标志物,他们分析了血浆数据。差异分析发现了 517 种明显失调的蛋白质,其中 CRC 患者血浆 EV 中 481 种上调(图 2 F)。PCoA 分析进一步显示 CRC 和 HC 患者血浆 EV 蛋白质组之间存在明显区别(图 2 G),差异比组织 EV 更明显。这种显著差异可能是由于组织来源的 EV 在血浆中的积累和富集。这些失调的蛋白质主要参与蛋白酶体活性、细胞骨架调控和翻译相关过程,在与钙粘蛋白结合和 RNA 结合相关的通路中显著富集。
通过将血浆EV蛋白质组数据与组织EV蛋白质组数据进行整合分析,他们定量了组织和血浆EV中78种差异表达蛋白质(图2 H)。将四种蛋白质(NIBAN1,CAVIN2,AOC1和P3H1)排除在外,因为它们未记录在Vesiclepedia数据库中。剩余74种蛋白质的蛋白质-蛋白质相互作用(PPI)网络分析显示其中76%是相互连接的,形成四个高度连接的聚类。这些聚类表明这些蛋白质可能在CRC增殖和发展中发挥关键作用。基于表达趋势的一致性和公开数据,他们最终选择了21种在血浆EV(CRC vs. HC)中表现出较高差异表达和在组织EV(PT vs. AM)中表现出一致表达变化的蛋白质进行PRM-MS验证。通过对组织和血浆EV中蛋白质相对丰度的分析,证实了这21种蛋白质在CRC血浆和PT组织中含量丰富(图2I-J)。因此,通过综合分析和严格的筛选标准,他们确定了21种蛋白质作为CRC诊断生物标志物的有力候选物,值得进一步研究。
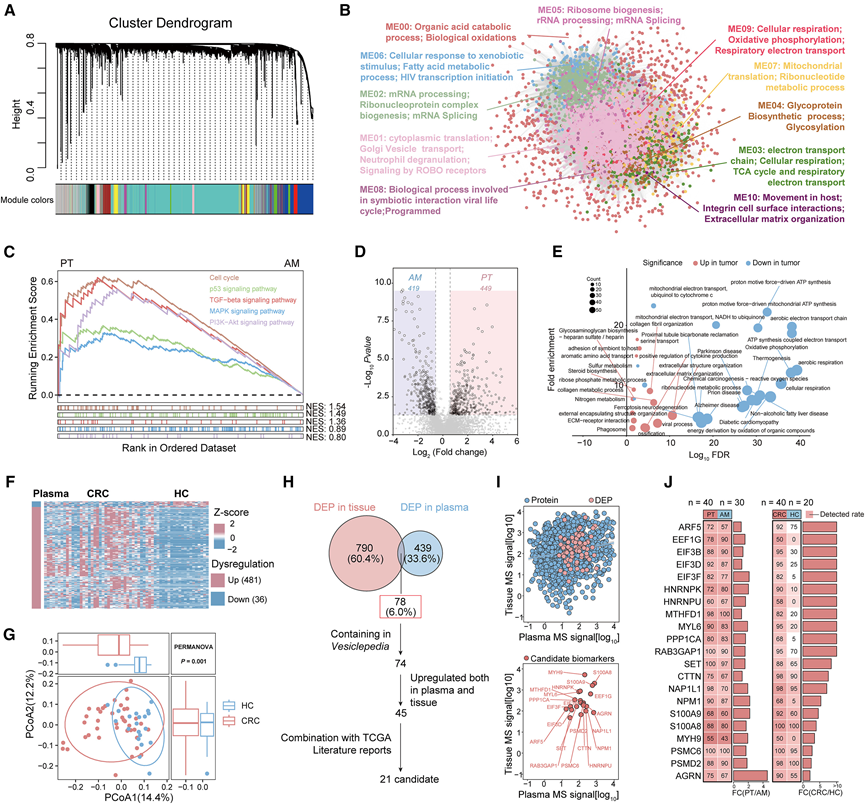
图2. 组织EV和血浆EV蛋白质组学的整合揭示了CRC诊断的候选生物标志物。
(A) WGCNA 基于组织 EV 蛋白质组鉴定出 11 个功能性蛋白质模块。(B) 11 个功能性蛋白质模块(ME00–10)的网络图,每个网络节点代表一种蛋白质,并按不同的功能模块用颜色编码。(C) 比较PT 和 AM 的 CRC 相关通路的 GSEA。(D) 火山图显示PT组织EV和AM组织EV之间差异丰度蛋白质。(E) 点图显示基于显著上调/下调蛋白质识别出的富集通路。(F) 聚类热图显示 CRC 患者和健康个体血浆 EV 中的 EV 蛋白存在差异。(G) 基于血浆 EV 样本 Bray-Curtis 距离的主坐标分析 (PCoA)。(H) 整合组织 EV 和血浆 EV 蛋白质组学以揭示 CRC 诊断的候选生物标志物。(I) 组织-血浆EV蛋白质组丰度图,显示组织EV中蛋白质强度中值(以MS强度评估)与血浆中蛋白质强度中值的关系。(J) 热图显示了组织EV样本(左)和血浆EV样本(右)中21个选定候选生物标志物的中位强度和检出率。
03
基于PRM-MS的靶向MS分析验证血浆EVs候选生物标志物
为了验证这 21 种候选生物标志物,他们采用了靶向蛋白质组学技术 PRM-MS。PRM-MS 成功定量了 40 份血浆 EV 样本中的这 21 种肽段,其中包括 20 份来自 CRC 患者,20 份来自 HC。随后,他们制定了一项策略,根据 PRM-MS 数据识别用于诊断 CRC 患者的潜在 EV 蛋白分子(图 3A)。其中,19 种候选蛋白的表达趋势与发现队列一致,而 S100A8 和 S100A9 则呈现相反趋势(图 3B)。MYH9 的高表达与 CRC 进展有关,这是通过丝裂原活化蛋白激酶/AKT 信号传导实现的,这凸显了研究 EV对于了解微环境的重要性。
对于诊断评估,13 种 EV 蛋白实现了显著的曲线下面积 (AUC) 值 (>0.75),其中 CTTN 表现出最高的 AUC 评分 (0.89) 和 Youden 指数 (0.75)(图 3C)。这些生物标志物的检测性能超过了 CA19-9 和 CEA 等传统血清学标志物。根据最小冗余最大相关性标准,表达水平高度相关的蛋白质可能会引入信息冗余。为了缓解这种情况,他们分析了 21 种蛋白质之间的相关性,并观察到某些蛋白质的表达水平是相关的。在这种情况下,只保留与其他蛋白质表达相关性较低的蛋白质(图 3D)。例如,PSMC6、CTTN 和 EIF3B 与大多数其他蛋白质的相关性较低。虽然 S100A8 和 S100A9 也与其他蛋白质表现出较低的相关性,但由于它们的约登指数(Youden index)低于 0.5,因此将其排除在候选生物标志物之外。此外,虽然PSMC2 具有良好的约登指数,但由于其与 HNRNPK 和 MYH9 等蛋白质的高相关性而排除在外。
他们利用机器学习 (ML) 技术筛选对 CRC 具有高诊断效力的血浆 EV 蛋白。首先,对 10 种最先进的 ML 模型进行了全面评估,并将它们与基于先验概率的基线模型进行了比较(图 3E)。使用准确度、受试者工作曲线 (ROC) AUC 和 F1 分数三个指标对分类器进行了全面评估。在评估了各种模型之后,选择了性能优异的随机森林算法(图 3F)。接下来,将这 21 种蛋白质用作输入变量,使用随机森林算法识别诊断模型中最重要的特征。
考虑到每个候选生物标志物的诊断潜力,包括随机森林模型中的表达相关性和特征重要性,他们最终确定了一个用于 CRC 诊断的候选血浆 EV 蛋白组合 (P6)。该组合包括 MYH9、HNRNPK、CTTN、PSMC6、NAP1L1 和 EIF3B。P6 组合的表现明显优于传统标志物,与 CEA 和 CA19-9 相比,其 CRC 诊断的 AUC 为 1.000(图 3G)。当与 CEA 或 CA19-9 结合时,P6 组合的 AUC 值分别达到 1 和 0.98。此外,他们使用随机森林模型评估了 P6 组合中单个蛋白质与 CEA 或 CA19-9 结合的诊断性能。超过 80% 的组合导致 AUC 大于 0.9,强调了 P6 组合中蛋白质的高诊断价值。总之,通过使用 PRM-MS 以及表达分析、相关性评估和基于 ML 的选择的策略组合,最终在 P6 组合中确定了六种潜在的诊断蛋白。
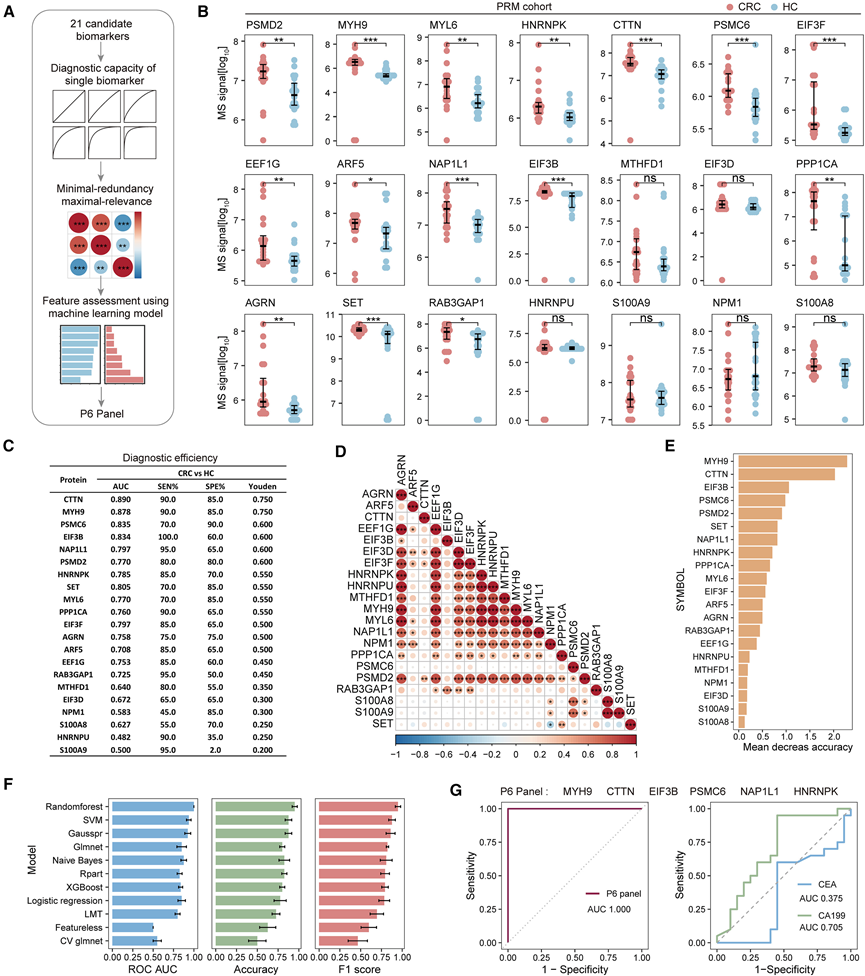
图3. 使用基于PRM-MS的靶向MS验证血浆EV候选生物标志物。
(A) 筛选 21 种候选蛋白质以选择适合开发诊断模型的蛋白质的流程图。(B)20 例 CRC 患者和 20 例健康个体中 21 个候选生物标志物的蛋白质丰度箱线图。(C) 单个候选生物标志物的诊断能力和突出显示的蛋白质纳入P6生物标志物组,用于ELISA验证。(D) 21个候选生物标志物的蛋白质丰度相关性。(E) 随机森林算法生成的变量重要性图,以每个变量的准确度平均下降来衡量。(F) 基于三个评估指标[ROC曲线下面积 (AUC)、准确率和 F1 分数]对 10 个最先进的机器学习分类器进行性能基准测试。(G) P6 组的 ROC 曲线在诊断 CRC 方面表现出比 CEA 或 CA19-9 更优异的性能。
04
用于 CRC 诊断的 ColonTrack 模型的开发
为了进一步验证 P6 组合对 CRC 的诊断潜力,他们在更大的队列中对这六种蛋白进行了 ELISA 定量分析,该队列包括 CRC 患者、健康个体以及患有炎症性肠病和晚期结直肠腺瘤 (CRA) 等 BD(良性疾病)的个体。EV 蛋白水平根据初始血浆量进行测定和校准。鉴于 PRM-MS 和 ELISA 之间数据类型的差异,他们制定了定制的特征选择和模型构建策略,以确保验证的准确性(图 4 A)。
他们首先评估了建模集中每种生物标志物的诊断潜力。在蛋白质中,CTTN、HNRNPK 和 PSMC6 在 CRC 患者血浆 EV中的浓度明显高于 BD 组和 HC 组(图 4 B)。虽然 MYH9 和 NAP1L1 在 CRC 和 HC 之间也存在显著差异,但它们无法有效区分 CRC 和 BD。在六种生物标志物中,CTTN、HNRNPK 和 PSMC6 在区分 CRC 与 HC 和 BD 方面表现最佳。使用 5 倍交叉验证的随机森林分析进一步证实了这三种蛋白质在分类中的重要性,显示准确率的平均下降幅度明显更高(图 4 C)。随后对各种生物标志物组合的分析表明 CTTN、HNRNPK 和 PSMC6 是最佳三者,在区分 CRC 与 HC(AUC = 0.967)和 BD(AUC = 0.944)方面具有较高的 AUC 值。为了进一步研究生物标志物表达与 CRC 进展之间的关系,他们根据 TNM 分期标准将 CRC 患者分为不同阶段。在四个 TNM 阶段中,CTTN、HNRNPK 和 PSMC6 在 CRC 中的表达均高于非 CRC 组。具体而言,CTTN 表达从 I 期到 IV 期呈下降趋势,而 HNRNPK 在同一阶段呈上升趋势。这些发现表明 CTTN 和 HNRNPK 的表达模式可能提示 CRC 进展,进一步支持这些生物标志物的诊断潜力。此外,CTTN从早期到晚期的表达下降可能暗示其与肿瘤侵袭和转移相关,这值得进一步研究其作为早期CRC的潜在预后标志物的作用。另一方面,HNRNPK随CRC进展而呈上升趋势,可能反映了其在细胞增殖、迁移和转移中的作用,这使得HNRNPK成为追踪疾病进展的潜在标志物。PSMC6在各期均保持稳定的高表达,提示其在CRC的早期检测和监测中具有重要意义,尤其是在DNA修复和细胞应激反应的背景下。基于这些发现,他们选择这三种蛋白作为构建CRC诊断ColonTrack检测组的关键生物标志物。
将建模集按7:3的比例随机分为训练集(123例CRC、64例BD和54例HC)和测试集(53例CRC、27例BD和23例HC),构建并验证CRC诊断的随机森林模型。ColonTrack模型在两组中均能有效区分CRC与HC和BD,表现出较强的判别能力。在训练集中,该模型对CRC vs. HC的AUC值为0.992(灵敏度=0.959,特异度=0.889),对CRC vs. BD的AUC值为0.998(灵敏度=0.959,特异度=0.969),准确率分别为93.2%和95.7%。在测试集中,该模型对CRC vs. HC的AUC值为0.990(灵敏度=0.981,特异性=0.913),对CRC vs. BD的AUC值为0.962(灵敏度=0.981,特异性=0.852),相应的准确率分别为96.1%和93.8%。总体而言,ColonTrack模型的诊断性能优于传统标志物CEA和CA19-9(图4 D-E)。总体而言,CRC患者的ColonTrack评分高于HC患者和BD患者。
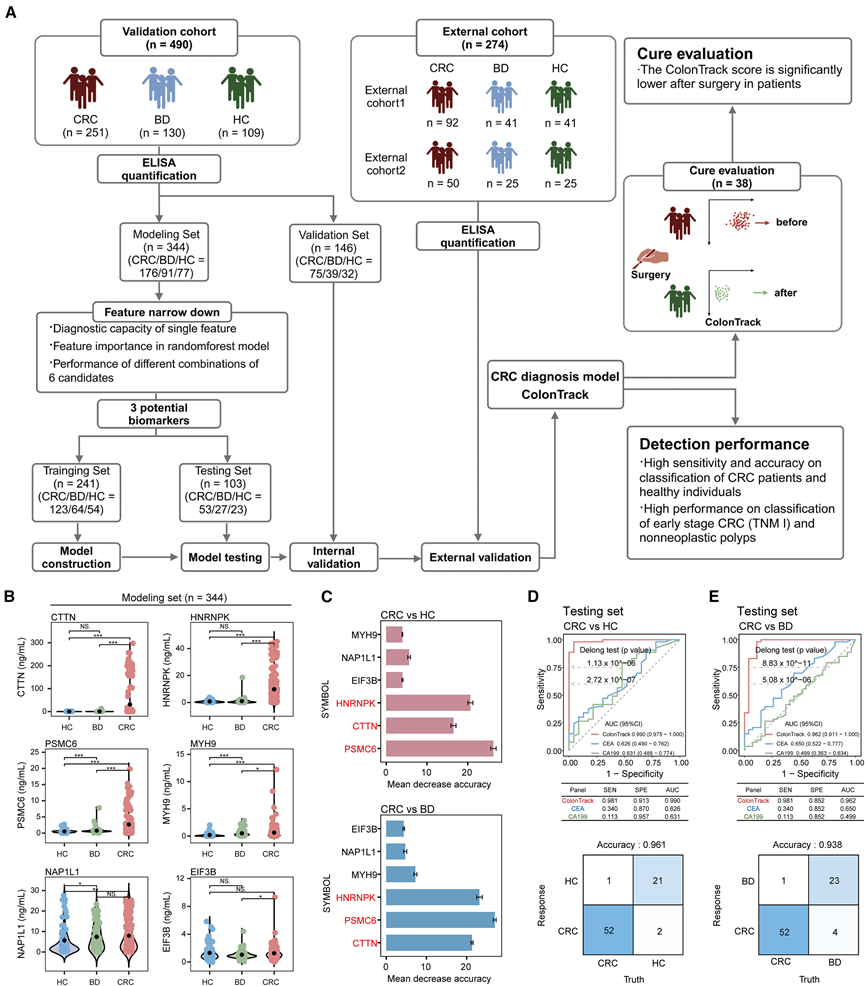
图4. 基于ELISA验证的CRC诊断ColonTrack模型开发。
(A) 基于 ELISA 定量结果的诊断模型构建和评估工作流程。(B)候选生物标志物的 ELISA 定量比较。(C) 随机森林算法生成的变量重要性图,以CRC与HC(上)和CRC与BD(下)的准确率下降平均值来衡量。(D) (上) ColonTrack 模型、CEA 和 CA19-9 在区分 CRC 患者和 HC 患者时的 ROC 曲线,(下)显示了在测试集中对 CRC 与 HC 进行分类的混淆矩阵。(E) (上)显示了 ColonTrack 模型、CEA 和 CA19-9 在区分 CRC 患者和 BD 患者时的 ROC 曲线,(下)显示了在测试集中对 CRC 与 BD 进行分类的混淆矩阵。
05
在内部和外部队列中验证 ColonTrack 模型对 CRC 诊断的有效性
为了进一步证实 ColonTrack 模型的诊断效果,他们使用独立队列进行了内部验证和外部验证。与 CEA 和 CA19-9 相比,ColonTrack 模型表现出显著增强的诊断性能,在两个验证集中区分 CRC 和非 CRC 个体均获得了最高的 AUC 值。具体而言,在内部验证集中,该模型对 CRC 与非 CRC 的 AUC 为 0.986(灵敏度 = 0.960,特异性 = 0.915),对 CRC 与 BD 的 AUC 为 0.988(灵敏度 = 0.960,特异性 = 0.923)(图 5 A)。在外部验证集中,ColonTrack 模型对 CRC 与非 CRC 的 AUC 为 0.974(灵敏度 = 0.923,特异性 = 0.947),对 CRC 与 BD 的 AUC 为 0.977(灵敏度 = 0.923,特异性 = 0.970)(图 5B)。在两个验证队列中,该模型在 CRC 患者中始终获得比 HC 和 BD 更高的评分(图 5C-D)。该模型还表现出很高的诊断准确率,内部集的准确率为 93.8%(CRC 与非 CRC)和 94.7%(CRC 与 BD),外部集的准确率为 93.4%(CRC 与非 CRC)和 93.8%(CRC 与 BD)(图 5A-B)。因此,这些结果强调了 ColonTrack 模型在 CRC 诊断中的重要临床价值。
为了评估 ColonTrack 的临床适用性,他们验证了它在早期 CRC 诊断中的表现,现有的诊断策略通常缺乏敏感性。与 CEA 和 CA19-9 等传统生物标志物相比,ColonTrack 在不同的 TNM 分期中始终表现出更高的敏感性。值得注意的是,对于 I 期 CRC 患者,ColonTrack 在内部验证和外部验证队列中的敏感性分别达到 1.000和 0.931(图 5E)。ColonTrack 评分清楚地表明,无论使用内部验证集还是外部验证集,I 期 CRC 患者的评分均显著高于 BD 和 HC。相反,BD 组和 HC 组之间的评分没有显著差异。ColonTrack 模型对早期 CRC 诊断表现出极高的敏感性,成功检测出 34 名 I 期 CRC 患者中的 32 名。值得注意的是,在 66 个 BD 样本的内部验证队列中,有三个错误归类为 CRC,而这些样本均为诊断为 CRA 的患者。
良性肠道疾病,尤其是 CRA,通常会导致 CEA 水平升高,使 CRC 诊断变得复杂。然而,ColonTrack 可有效区分早期 CRC 与 CRA,96.4% (185/196) 的 BD 病例检测呈阴性,而 23.5% (46/196) 的 CEA 检测呈阳性。在内部和外部验证队列中,区分 I 期 CRC 与 CRA 的 AUC 分别为 1.000 和 0.969,突显了 ColonTrack 在早期 CRC 诊断中的强大预测价值(图 5 F-G)。此外,这三种关键蛋白的水平在手术后显著下降(图 5 H-I)。患者的 ColonTrack 检测结果通常为阴性,表明其不仅可用于初步诊断,还可作为手术反应监测的有用生物标志物。这些发现支持使用 ColonTrack 进行早期检测和术后监测,为 CRC 管理提供个性化方法,符合精准医疗的目标。
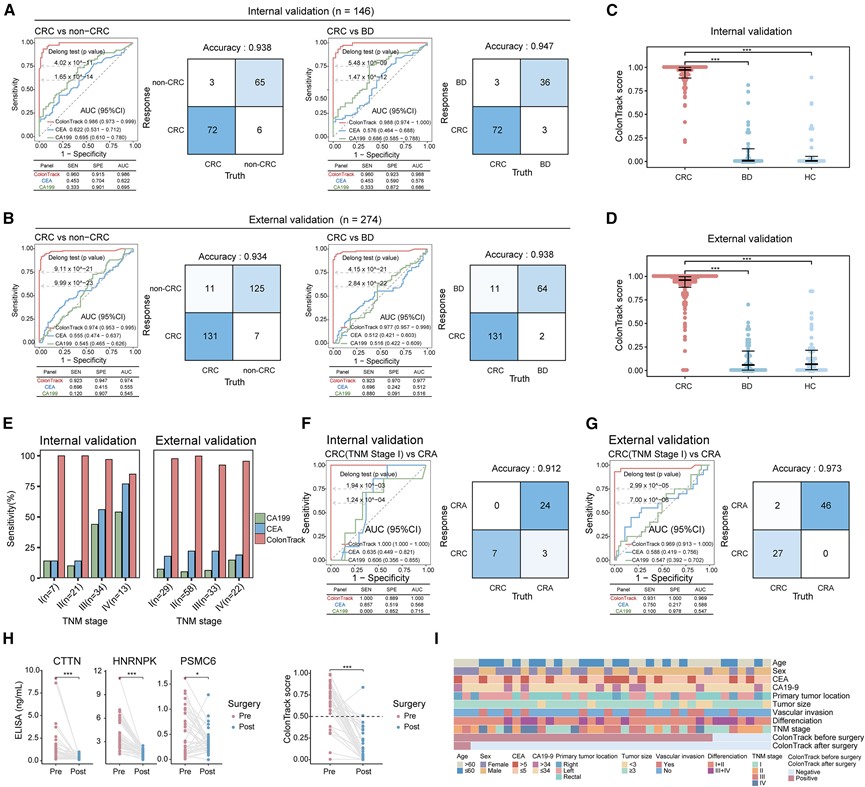
图5. ColonTrack模型对大样本群体的诊断性能。
(A)ColonTrack 模型、CEA 和 CA19-9 在区分 CRC与 HC和BD患者时的 ROC 曲线,以及在内部验证集中对 CRC 与 HC 或 CRC 与 BD 进行分类的混淆矩阵。(B) ColonTrack 模型、CEA 和 CA19-9 在区分 CRC与 HC和BD患者中的 ROC 曲线,以及在外部验证集中对 CRC 与 HC 或 CRC 与 BD 进行分类的混淆矩阵。(C-D) CRC、BD 和 HC 的 ColonTrack 分数。(E) ColonTrack模型、CEA和CA19-9对内部验证集和外部验证集中不同TNM分期CRC患者的检测灵敏度。(F) ColonTrack 模型、CEA 和 CA19-9 在区分早期(TNM 分期 I)CRC 患者与 CRA 患者时的 ROC 曲线,以及在内部验证集中对 CRC 与 CRA 进行分类的混淆矩阵。(G) ColonTrack 模型、CEA 和 CA19-9 在区分早期(TNM 分期 I)CRC 患者与 CRA 患者时的 ROC 曲线,以及在外部验证集中对 CRC 与 CRA 进行分类的混淆矩阵。(H) 通过比较手术前后的 ELISA 检测结果,发现 CRC 患者血浆 EV中的 CTTN、HNRNPK 和 PSMC6 表达水平在手术后显著下调。(I) 术后患者临床指标。
06
使用附加队列对 ColonTrack 性能进行基准测试
mSeptin-9 DNA 以其高灵敏度而闻名,已成为一种有前途的 CRC 诊断工具,并在 CRC 检测的实验室阶段得到越来越多地应用。为了更有效地证明 ColonTrack 模型的诊断价值,他们收集了一个额外队列,其中包括 126 名早中期 CRC(I-III 期)患者、68 名 CRA 患者和 68 名健康对照者。特意选择这个队列是为了确保所有 CRC 患者之前都接受过标准诊断测试,包括 CEA、CA199 和 Septin9,这些测试用于对 ColonTrack 模型的性能进行基准测试。随后,按照前面描述的方法,他们使用 ELISA 测量了该队列参与者的三种生物标志物蛋白水平。然后使用 ColonTrack 模型对样本进行诊断(图 6 A)。通过将 ColonTrack 与这些已建立的标记物的诊断能力进行比较,他们旨在评估其对早期 CRC 检测的相对有效性和潜在优势,从而对其临床适用性进行更全面的评估。
在区分 CRC 和非 CRC 组时,ColonTrack 的 AUC 达到 0.984(灵敏度 = 0.960,特异性 = 0.949),准确率为 95.0%(图 6B)。在区分早期 CRC 和 CRA 时,该模型的表现同样出色,AUC 为 0.979(灵敏度 = 0.962,特异性 = 0.912),准确率为 92.6%(图 6C)。这些结果进一步证实了该模型的稳健性和多功能性,表明其能够承受样本异质性和检测批次间差异。这表明该模型具有更广泛的临床应用潜力。
然后,他们将 ColonTrack 的诊断性能与几种成熟的 CRC 诊断指标(包括 CEA、CA199 和 mSeptin-9)进行了比较。在另外 126 个 CRC 样本中,ColonTrack 的检出率最高,为 95.2%,优于 mSeptin-9,后者的 CRC 检出率为 89.7%,与之前发布的数据一致。ColonTrack 和 mSeptin-9均表现出比 CEA 和 CA199 等传统标志物更高的检测灵敏度。值得注意的是,将 ColonTrack 与 mSeptin-9 结合起来可将检出率提高到 99.2%,表明这两种诊断方法具有互补性(图6D)。这表明,整合这些生物标志物可以增强 CRC 检测,提供更全面、更可靠的方法。
进一步比较 ColonTrack 和 mSeptin-9 对不同分期 CRC 的诊断效果,发现早期诊断结果相似,检出率分别为 94.3% 和 96.2%。然而,ColonTrack 在诊断 II 期和 III 期 CRC 方面表现略好,检出率分别为 98.2% 和 95.7%,而 mSeptin-9 的检出率分别为 78.6% 和 86.9%。由于新增队列主要由早中期 CRC 患者组成,无法评估这些指标在 IV 期 CRC 中的表现。为了提供更全面的基准数据,他们整合了验证队列、外部验证队列和手术队列的临床数据,共计 431 名 CRC 患者。其中,154 名患者接受了 mSeptin-9 检测,包括 23 名 I 期患者、56 名 II 期患者、53 名 III 期患者和 22 名 IV 期患者。在这一组中,mSeptin-9 的 CRC 检出率为 90.3%,与之前报告的数据一致,而 ColonTrack 显示出更高的 96.1% 的总体检出率。在 I 期和 II 期 CRC 中,ColonTrack 的表现优于 mSeptin-9,检出率分别为 95.7% 和 98.2%,而 mSeptin-9 的检出率分别为 86.9% 和 78.6%(图 6 E)。对于 III 期 CRC,两种生物标志物的检出率均达到了 100%。然而,在 IV 期 CRC 中,mSeptin-9 显示出 100% 的完美检出率,而 ColonTrack 的检出率略低,为 81.8%。这些结果突出了 ColonTrack 在早期 CRC 检测中的潜力,而 mSeptin-9 可能对检测晚期 CRC 更敏感。
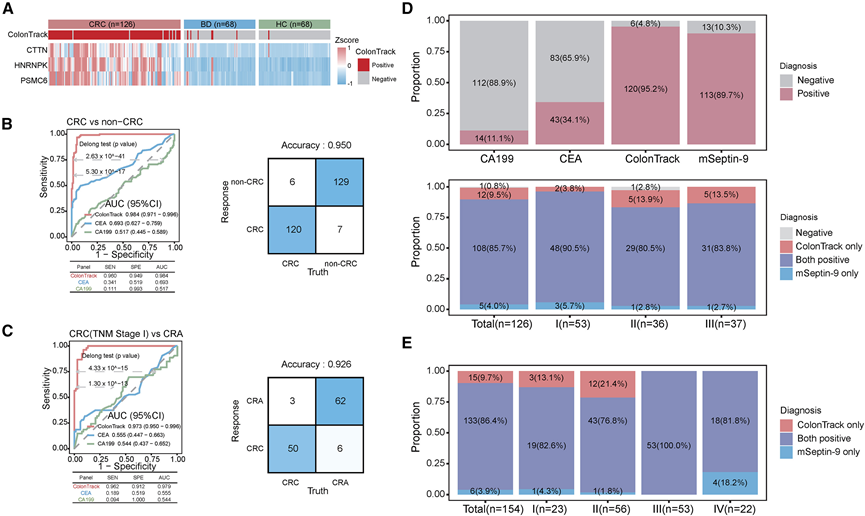
图7. 使用附加队列对 ColonTrack 性能进行基准测试。
(A) 热图显示CTTN、HNRNPK和PSMC6的表达水平及ColonTrack的诊断结果。(B) ColonTrack 模型、CEA 和 CA199 在区分 CRC 患者与非 CRC 患者时的 ROC 曲线,以及在附加队列中对 CRC 与非 CRC 进行分类的混淆矩阵。(C) ColonTrack 模型、CEA 和 CA199 在区分 CRC患者和 CRA患者中的 ROC 曲线,以及在附加队列中对 CRC 与 CRA 进行分类的混淆矩阵。(D) 不同指标的 CRC 阳性率(上)以及附加队列中 CRC 不同阶段的 ColonTrack 和 mSeptin-9 检测率(下)。(E) 回顾性队列中不同 CRC 阶段的 ColonTrack 和 mSeptin-9 CRC 阳性率。
+ + + + + + + + + + +
结 论
本研究提出了一个综合的工作流程,将基于分阶段质谱 (MS) 的发现和验证与基于 ELISA 的验证相结合,以识别 CRC 的 EV 蛋白生物标志物。应用于 1272 名受试者,构建了一个机器学习模型 ColonTrack,该模型整合了 EV 蛋白 HNRNPK、CTTN 和 PSMC6。ColonTrack 能够有效区分 CRC 和非 CRC 病例,并以较高的准确率识别早期 CRC(合并曲线下面积 [AUC] >0.97,灵敏度 ∼0.94,特异性 ∼0.93)。本亚军对组织和血浆中 EV 蛋白谱的分析表明,ColonTrack 有望成为一种用于 CRC 诊断和早期检测的强大的非侵入性生物标志物组合。
+ + + + +



 English
English


