文献解读|J Med Virol(12.7):多组学数据揭示人乳头瘤病毒整合对宫颈肿瘤发生的表观基因组和转录组特征的新影响
✦ +
+
论文ID
原名:Multi-omics data reveals novel impacts of human papillomavirus integration on the epigenomic and transcriptomic signatures of cervical tumorigenesis
译名:多组学数据揭示人乳头瘤病毒整合对宫颈肿瘤发生的表观基因组和转录组特征的新影响
期刊:Journal of Medical Virology
影响因子:12.7
发表时间:2023.05.20
DOI号:10.1002/jmv.28789
背 景
人乳头瘤病毒 (HPV) DNA 整合到人类基因组中可能会逐渐导致宫颈癌的发生。HPV DNA 在持续感染过程中经常整合到宿主基因组中,这是宫颈癌发生中的重要分子事件。研究表明,HPV 整合常常会破坏 HPV E1 和 E2 基因的开放阅读框,从而导致癌基因 E6 和 E7 的上调。E6 和 E7 都有多个促进恶性转化的细胞靶点。E6 结合并降解抑癌基因 p53 和促凋亡蛋白 Bak,而 E7 抑制抑癌基因 pRB 释放 E2F 转录因子,从而促进细胞增殖。由于超过 80% 的宫颈癌含有 HPV 整合事件,阐明 HPV 整合事件对导致宫颈癌的机制的影响至关重要。
实验设计
结 果
01
宫颈癌患者基因组中HPV整合特征
在本研究中,研究团队收集了 50 例 HPV 阳性宫颈癌患者的 97 份宫颈组织,其中包括 47对肿瘤和癌旁组织样本、1 份肿瘤组织样本和 2 份癌旁组织样本。为了表征 HPV 在人类基因组中的整合,他们分析 HPV 捕获测序数据以确定 HPV 整合位点,鉴定了 1470 个 HPV 整合位点,包括来自 42/48 肿瘤组织的 985 个整合位点和来自 41/49 肿瘤旁组织的 485 个整合位点。他们在每个肿瘤和肿瘤旁组织样本中分别鉴定出20.5和9.9个断点(图1A)。肿瘤样本中HPV整合位点的数量明显高于癌旁组织样本(图1B)。为了研究HPV整合位点的特征,他们分析了HPV整合位点在人类基因组中的分布。HPV整合分布在整个人类基因组中(图1D)。然而,在肿瘤组织中,发现HPV整合在染色体2、13、19和22中显著富集,特别是在染色体22的末端(图1C)。然而,他们发现在肿瘤组织和配对的肿瘤旁组织之间,CpG岛中HPV整合的分布没有显著差异(图1E)。
有趣的是,他们发现HPV在肿瘤和肿瘤旁组织中的整合倾向于彼此靠近,这表明HPV片段倾向于在人类基因组的附近位置整合(图1F)。他们还观察到HPV整合在基因间区域富集(图1G)。综上所述,这些发现表明HPV整合到宿主基因组并不是随机事件。
接下来,他们分别在肿瘤和肿瘤旁组织中鉴定了627和424个包含HPV整合的基因。在肿瘤组织中共发现48.4%的整合位点位于基因区域,这与癌旁组织相比有统计学差异。在本研究中,在≥3例患者中显示HPV整合的基因则定义为高频HPV整合基因。他们分别在肿瘤和癌旁组织中鉴定了9个和6个高频HPV整合基因。除已报道的KLF5、LINC00392 和MIR205HG外,他们发现了几个在肿瘤组织中整合频率较高(n≥3)的新基因,包括LINC00486、LINC02425、LLPH、PROS1和NRG1(图1A-D)。发现6个基因在瘤旁组织中含有HPV整合体。有趣的是,六个基因中的三个基因(ATP6V1G3 、NEK7和MIPOL1)的HPV整合仅存在于瘤旁组织中。LINC02425和LLPH在肿瘤组织中的整合率显著高于在瘤旁组织中的整合率。此外,MIR205HG在肿瘤组织中具有更高的整合率,而MIPOL1在肿瘤旁组织中具有更高的整合率。
GO功能和KEGG通路富集分析显示,在“肝素结合”、“细胞外基质”、“酶联受体蛋白信号通路”、“蛋白质修饰过程负调控”和“ERBB4信号通路”中,HPV整合度≥1的整合基因富集(图1H)。
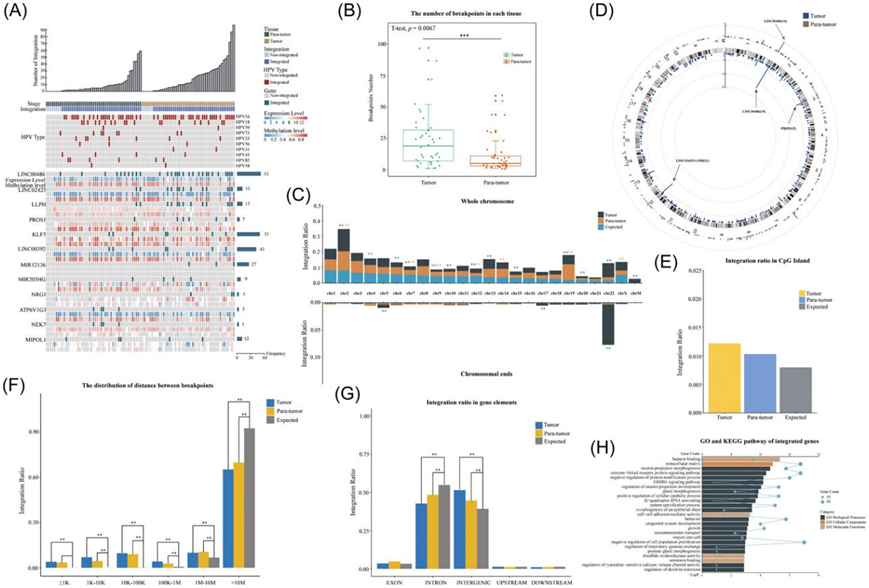
图1. HPV 整合到人类基因组中的特征。
(A) 宫颈癌和配对的邻近肿瘤旁的 HPV 整合事件概况。(B) 每个肿瘤和肿瘤旁组织中的断点数量。(C) 每个人类染色体和染色体末端中 HPV 整合位点在整个基因组中所有 HPV 整合中的比率。(D) 人类基因组中断点的分布。每个条形代表人类基因组 (hg38) 中特定基因座处 HPV 整合断点的样本频率。(E) CpG 群岛的整合率。 (F) 断点之间的距离分布。 (G) 肿瘤(蓝色)、肿瘤旁(黄色)和预期(灰色)条件下各种基因组元件中 HPV 整合的百分比。(H) 通过 HPV 整合对所有基因进行 GO 和 KEGG 通路富集分析。
02
HPV基因组的断点
肿瘤组织和癌旁组织中鉴定出的整合断点的HPV亚型总体一致,但不同组织中每种HPV亚型的百分比有所不同。在肿瘤组织和癌旁组织的整合断点处发现的主要 HPV 类型是 HPV16(占肿瘤组织中断点的 63.6% 和癌旁组织中断点的 64.4%)和 HPV18(占肿瘤组织中断点的 20.4% 和 肿瘤旁组织中22.8%)。按整合断点计数比例排序的HPV亚型分别为:肿瘤组织中的HPV16、18、59、73、33、56、31、45、82和58,瘤旁组织中的HPV16、18、73、45、58、33、82、56和59(图 1A、图 2A)。
除了整合的HPV DNA片段外,宿主细胞中还可能存在外生体HPV基因组。虽然断点可能分散在整个HPV基因组中,但主要的整合型HPV(HPV16和HPV18)呈现出不同的整合模式(图2B-C)。HPV16基因组倾向于在E1和E1^E4中断裂,但在LCR、E2、E6和E7中具有明显较少的断点(图2D)。这与之前的研究一致,研究表明整合的HPV16病毒可以在长控制区保留完整的E6和E7癌基因。E6和E7可能以完整的形式保存或整合到宿主基因组中。HPV18基因组中的断点在E1中富集(图2E)。这些结果表明,不同的HPV亚型以亚型特异性的方式整合到人类基因组中,这可能以不同的方式驱动癌症。
接下来,他们将人类基因组中发现的整合位点与HPV16和HPV18基因组中的断点进行了比对,发现人类基因组上的一个位点可以由多个HPV DNA片段整合,特别是在肿瘤组织中(图2F)。数据还显示,HPV16倾向于整合到肿瘤旁组织的2、13号染色体,而HPV18倾向于整合到肿瘤旁组织的2、3、19号染色体和肿瘤组织的2号染色体。然后,他们将肿瘤组织中检测到的5个高频HPV整合基因的整合位点与HPV基因组上的断点进行比对分析,发现整合在LINC02425、LLPH和PROS1基因中的HPV片段来自整个HPV基因组,并且没有明显的差异。相比之下,整合到LINC00486中的HPV片段优先来自HPV16的E1^E4、L1、L2和LCR基因区域。在KLF5/LINC00393附近整合的HPV片段主要来自E1、E5、L1、L2和LCR,在MIR205HG附近整合的HPV片段主要来自HPV16的L1基因。最后,NRG1基因更频繁地由HPV16的E2和E5基因整合(图2G)。
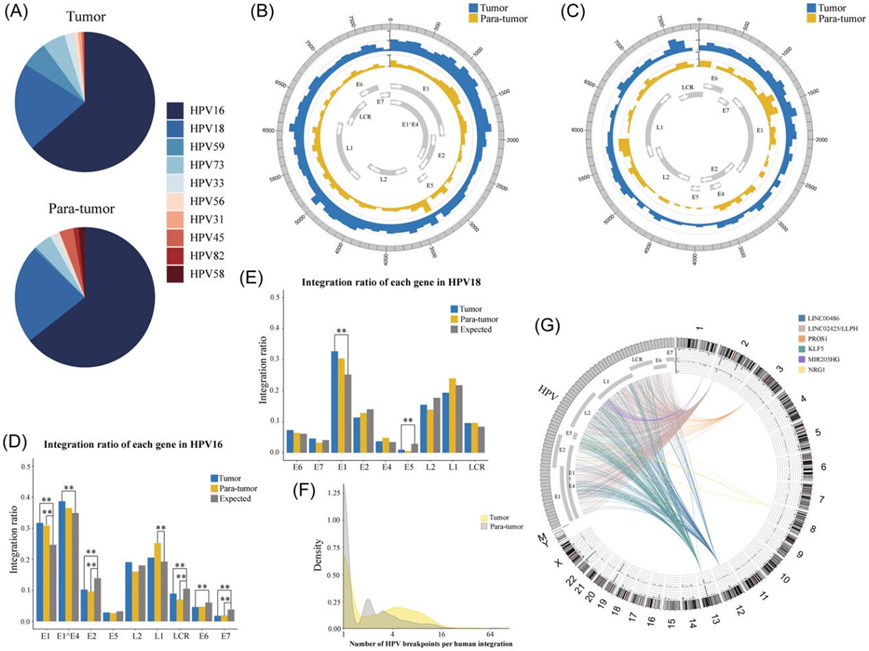
图2. HPV 基因组中断点的分布。
(A) 整合到肿瘤和肿瘤旁组织中的 HPV 亚型。(B-C) 分别是 HPV16 和 HPV18 基因组滑动窗口上的断点数量。(D-E) HPV16和 HPV18 的 HPV 片段分别整合到人类基因组中的比例。 (F) 肿瘤和肿瘤旁组织中人类基因组上每个整合位点的 HPV 断点数量。(G) HPV 整合频率高的基因及其在 HPV 基因组中相应的断点。
03
基因表达与HPV整合之间的关系
HPV 整合会影响宿主基因的表达,因此他们进行了转录组分析(RNA-seq) 以研究 HPV 整合事件中基因表达的新调控模式。他们鉴定了1412个差异表达基因(DEG),其中与肿瘤旁组织相比,肿瘤中747个上调基因和665个下调基因(图 3A)。这些DEG富集于“细胞外基质”、“细胞周期”、“DNA复制”和“微管结合”等通路,表明参与宫颈癌发展的细胞过程(图 3B)。在高频率整合基因中,KLF5、LINC00392、MIR205HG、NRG1、ATP6V1G3在肿瘤组织中显著上调,而PROS1在肿瘤组织中显著下调。为了进一步研究基因表达和HPV整合之间的关系,他们比较了同一基因中有HPV整合的样本和没有HPV整合的样本中各基因的表达水平。在肿瘤组织中,与相同基因中未发生HPV整合的肿瘤组织相比,发现18个有HPV整合的基因表达上调,而3个有HPV整合的基因表达下调(图3C)。在癌旁组织中,有3个HPV整合的基因显著上调(图3D)。
他们分别对每个基因进行了肿瘤/非整合肿瘤和癌旁/非整合癌旁的比较分析,然后统计不同基因组元件中包含HPV整合的上调和下调基因的数量。除了在肿瘤中与非整合肿瘤相比,包含HPV整合的上调基因之外,与HPV整合相关的所有其他类型的DEG均在基因间区域的HPV整合位点显著富集(图3E-F)。此外,只有在肿瘤中表达上调的HPV整合基因(与未整合相同基因的肿瘤相比)在内含子区域的HPV整合位点上富集(图3E),提示内含子中发生的HPV整合可能导致宿主基因上调,从而促进细胞癌变。
此外,只有来自肿瘤组织的具有HPV整合的DEG在外显子区域含有整合位点(图3E),而来自肿瘤旁组织的DEG则没有(图3F),这表明HPV外显子整合可能导致基因编码蛋白的结构改变,这些改变可能在致癌过程中起关键作用。为了进一步证实HPV整合与上述鉴定的DEG之间的关联,他们比较了肿瘤组织中具有HPV整合的DEG与匹配的邻近肿瘤旁组织的表达水平。他们鉴定出6个上调基因和4个下调基因。值得注意的是,在相同的基因中,这些基因的表达水平在肿瘤组织和配对的癌旁组织中以及在整合的肿瘤组织和非整合的肿瘤组织中受到相同的调节。他们还鉴定了1个上调基因和8个下调基因,这些基因在癌旁组织和配对的肿瘤组织的比较中,以及在整合的癌旁组织和非整合的癌旁组织的比较中,表达方向一致(图3G)。综上所述,这些基因的表达水平可能受到HPV整合的影响。
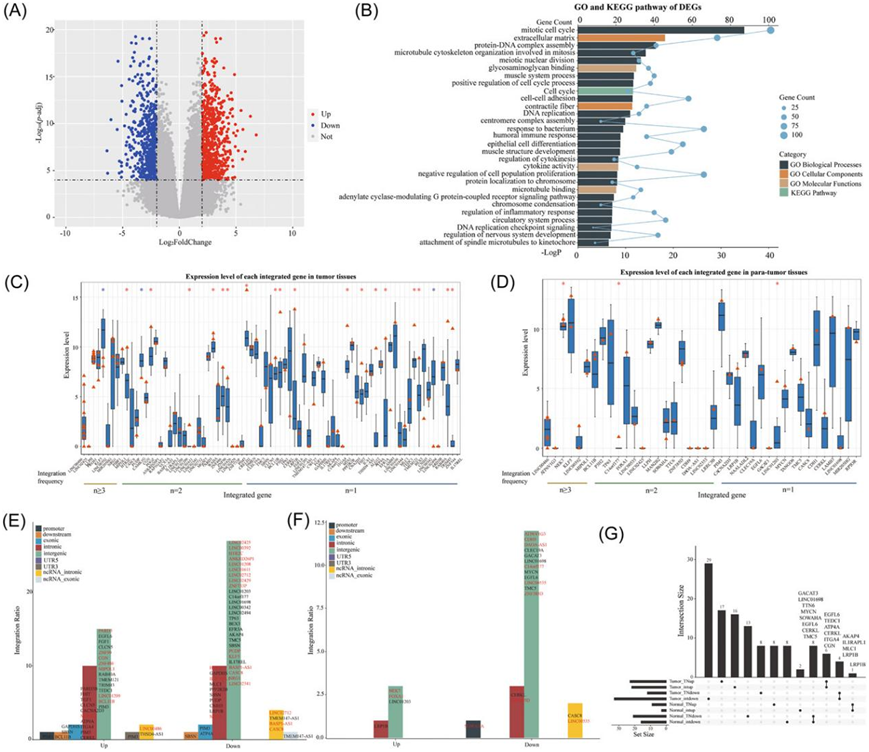
图3. HPV 整合基因的表达分析。
(A) 火山图显示肿瘤和肿瘤旁组织之间的 DEG。(B) DEG 的 GO 和 KEGG 通路富集分析。(C-D) 分别在所有肿瘤和癌旁组织中检测 HPV 整合基因的表达水平。(E-F) HPV整合基因的功能元件中HPV整合位点的分布,这些功能元件在HPV整合的肿瘤与非整合的肿瘤组织的比较中差异表达以及在HPV整合的与非整合的邻近肿瘤组织的比较中的差异表达。(G) 肿瘤和肿瘤旁组织之间以及整合和非整合组织之间具有差异表达水平的整合基因的重叠。
04
基因甲基化与HPV整合的关系
为了进一步探讨 HPV 整合是否通过改变 DNA 甲基化影响基因表达,他们通过全基因组亚硫酸氢盐测序(WGBS)研究病毒整合是否影响 DNA 甲基化,分析了人类基因组中 CpG 位点的 DNA 甲基化水平,肿瘤组织全基因组甲基化水平显著低于癌旁组织。Genomation 可以将一个差异甲基化区域 (DMR)注释为多个基因元件,这表明一个区域的差异甲基化可能会影响多个功能元件或基因(图 4A)。通过将甲基化与邻近肿瘤旁组织中发现的水平进行比较,他们将肿瘤的 DMR 分为高 DMR 和低 DMR。占据人类基因组绝大多数的内含子和基因间区域在肿瘤中呈低甲基化,且肿瘤基因组相对于肿瘤旁基因组通常是低甲基化的。他们还发现,相对于肿瘤旁组织和整个基因组的预期随机分布,上游区域(启动子和 TSS 上游 1-5 kb 区域)、外显子、CpG 岛和附近区域在肿瘤中往往高度甲基化(图 4B -C)。由于外显子和基因上游区域,特别是启动子(通常富含CpG位点),作为编码区或顺式调节区,他们推测这些区域DNA甲基化的增加可能在宫颈癌的发展中起关键作用。这些结果与之前的研究一致,即癌细胞表现出全基因组的低甲基化,但在特定的CpG岛启动子区域表现出高甲基化。
共有7120个基因在肿瘤和肿瘤旁组织的基因体区存在甲基化差异,其中271个具有HPV整合位点(图4E)。此外,1462个基因在启动子区域存在差异甲基化,其中57个基因具有HPV整合位点(图4F)。
最后,由于启动子区域的DNA甲基化对基因表达起关键的顺式调控作用,他们对启动子中具有DMR的基因进行了GO和KEGG富集分析。这些结果显示,“参与分化的细胞形态发生”、“细胞-细胞粘附”和“细胞连接组织”等几个通路富集(图4D)。接下来,他们用启动子的甲基化水平来代表基因的甲基化水平。计算高频HPV整合基因的甲基化水平,肿瘤组织样本中LINC00486、LINC00392、MIR205HG、NEK7和MIPOL1的甲基化水平显著低于癌旁组织样本,且LLPH在肿瘤组织中呈高甲基化状态。
他们接下来将进一步研究基因甲基化与HPV整合之间的关系,有5个基因(PROS1、ANKRD26P1、MIR548AG1、MTA1和MLC1)在HPV 整合的肿瘤中(相对于没有 HPV 整合的肿瘤)表现出高甲基化。此外,他们还鉴定了6个基因(BASP1-AS1、LINC02712、BCL11B、THSD4-AS1、LAMB3和ZNF544),这些基因在HPV整合的肿瘤组织样本中低甲基化(图4G)。在瘤旁组织中,他们发现三个与HPV整合的基因(NEK7、C14orf177和LINC01203)相对于缺乏HPV整合的瘤旁组织显示出显著的高甲基化,以及四个HPV整合的基因(LINC00392、P3H2、TP63和LLPH)低甲基化(图4H)。
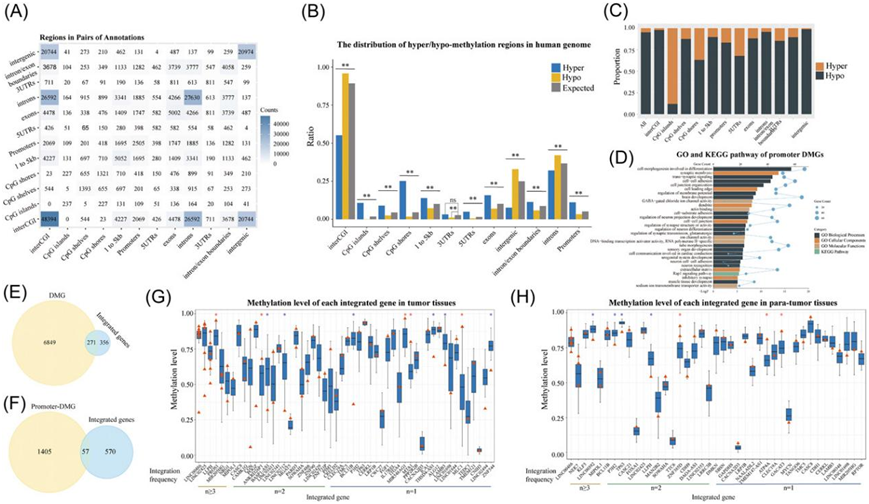
图4. HPV 整合基因的 DNA 甲基化分析。
(A) DMR 发生在多个基因组区域。(B) 人类基因组中肿瘤组织和肿瘤旁组织中低 DMR 和高 DMR 的不同分布。(C) 人类基因组特定区域中高 DMR 和低 DMR 的比例。(D) 具有差异甲基化启动子的基因的 GO 和 KEGG 通路注释的富集分析。(E) 具有差异甲基化的整合基因的维恩图。(F) 绘制具有差异甲基化启动子的整合基因的维恩图。(G-H) 分别是肿瘤组织和肿瘤旁组织中 HPV 整合基因的甲基化水平。
05
利用多组学数据对高频HPV整合基因进行综合分析
为了探索HPV整合基因在基因组、表观遗传和转录组水平上是如何调控的,他们结合了三个数据集——包括HPV整合数据、转录组数据和DNA甲基化数据——进行综合分析。他们比较了在这些基因中有HPV整合和没有HPV整合的样本之间高频HPV整合基因的表达和DNA甲基化水平。
当对肿瘤和肿瘤旁组织进行整体分析时,他们发现在PROS1中整合HPV的样本中,PROS1的启动子甲基化水平显著高于其他缺乏该基因整合HPV的样本。与此相关的是,在由HPV整合的PROS1样本中,PROS1的表达水平明显低于未由HPV整合的样本。这一结果与以往的研究一致,表明启动子的甲基化水平与相应基因的表达水平普遍呈负相关。
PROS1是本项研究数据中样本频率第三高的HPV整合热点,在10.4%的肿瘤中,在其转录起始位点上游约40 Kb处包含6个HPV整合,所有六个HPV整合都位于卫星DNA中。三个RNA-seq样本中PROS1的RNA剪接模式非常相似,这可能是由于这三个样本具有非常相似的HPV整合位置(图5A)。这些结果表明HPV整合可能通过调节PROS1启动子的DNA甲基化水平来影响基因表达。他们还发现DNA甲基化与MIR205HG基因表达呈负相关。与未整合的样品相比,HPV整合的样品中MIR205HG表达水平上调。此外,在整合该基因的HPV样本中,MIR205HG的甲基化水平显著低于未整合该基因的样本。值得注意的是,MIR205HG上游的整合位点位于其增强子中(图5B),这表明HPV可能通过整合到MIR205HG的增强子区来调节MIR205HG的表达和甲基化水平。
与PROS1相比,他们观察到MIR205HG不同的剪接模式,这可能是由于HPV整合位点的不同位置(图5B-D)。这些结果表明,HPV整合、基因表达、剪接和基因甲基化之间存在密切的关系。HPV整合可能通过改变DNA甲基化来调节PROS1和MIR205HG的表达,这一过程可能在宫颈癌的发展中起关键作用。
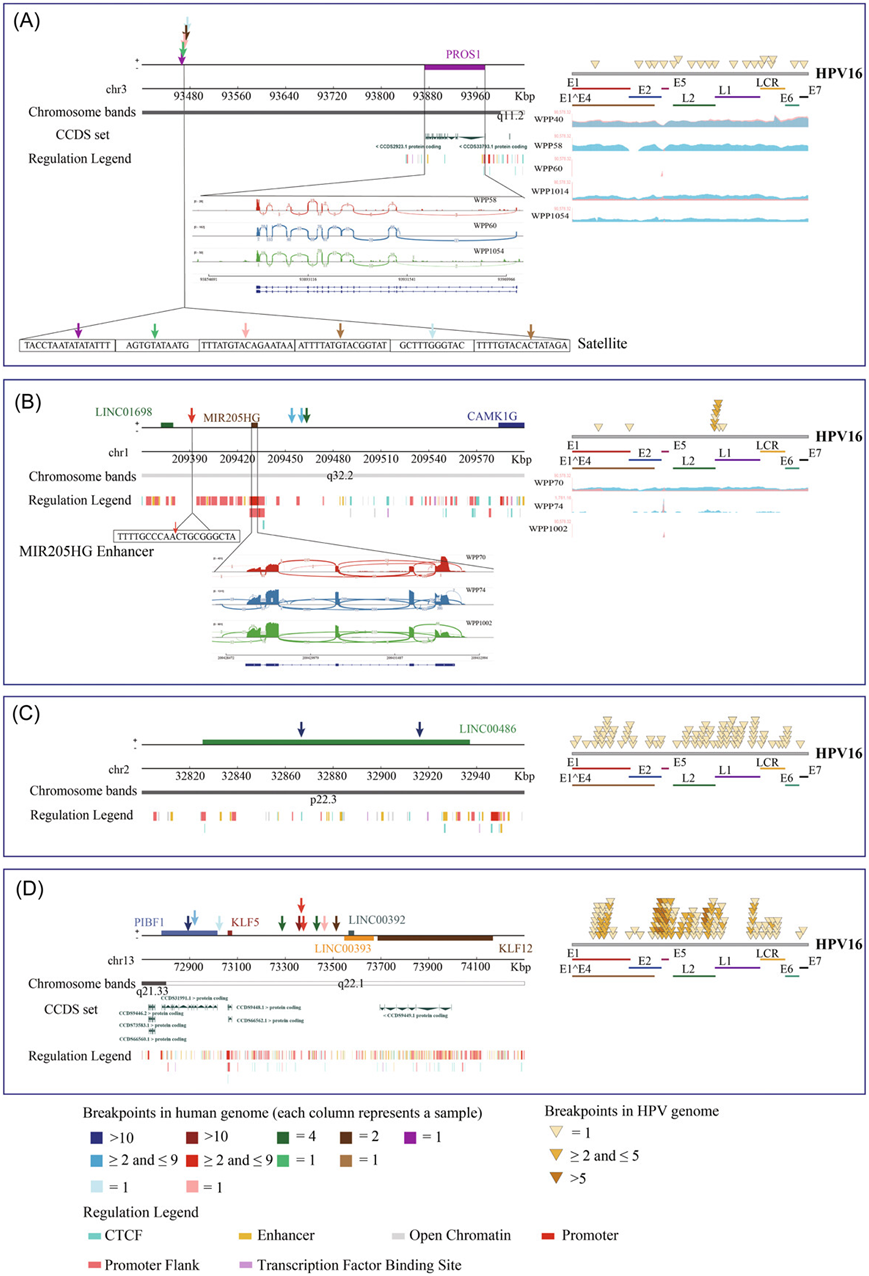
图5. HPV 整合的热点及其与多组学数据的关系。
(A) PROS1;(B) MIR205HG;(C)LINC00486;(D) KLF5;显示了 HPV 整合热点附近的基因位置。
为了探索通过多组学分析确定的HPV整合热点的功能,他们选择了MIR205HG进行进一步验证(图6A)。MIR205HG在ME180和CASKI中的表达高于SiHa和HeLa。然后,他们设计了三种siRNA来干扰MIR205HG在ME180和Caski中的表达(图6B),发现MIR205HG敲除后,细胞的增殖能力和迁移能力显著降低(图6C-H)。细胞增殖(图6I-J)和损伤愈合实验(图6K-L)也显示MIR205HG促进宫颈癌细胞的增殖和迁移。此外,他们还注意到MIR205HG敲低后,几个关键蛋白(如E-cadherin、Snail1、P53和CyclinD1)的丰度发生了变化(图6M),这些结果证明了MIR205HG在宫颈癌中的致癌作用。
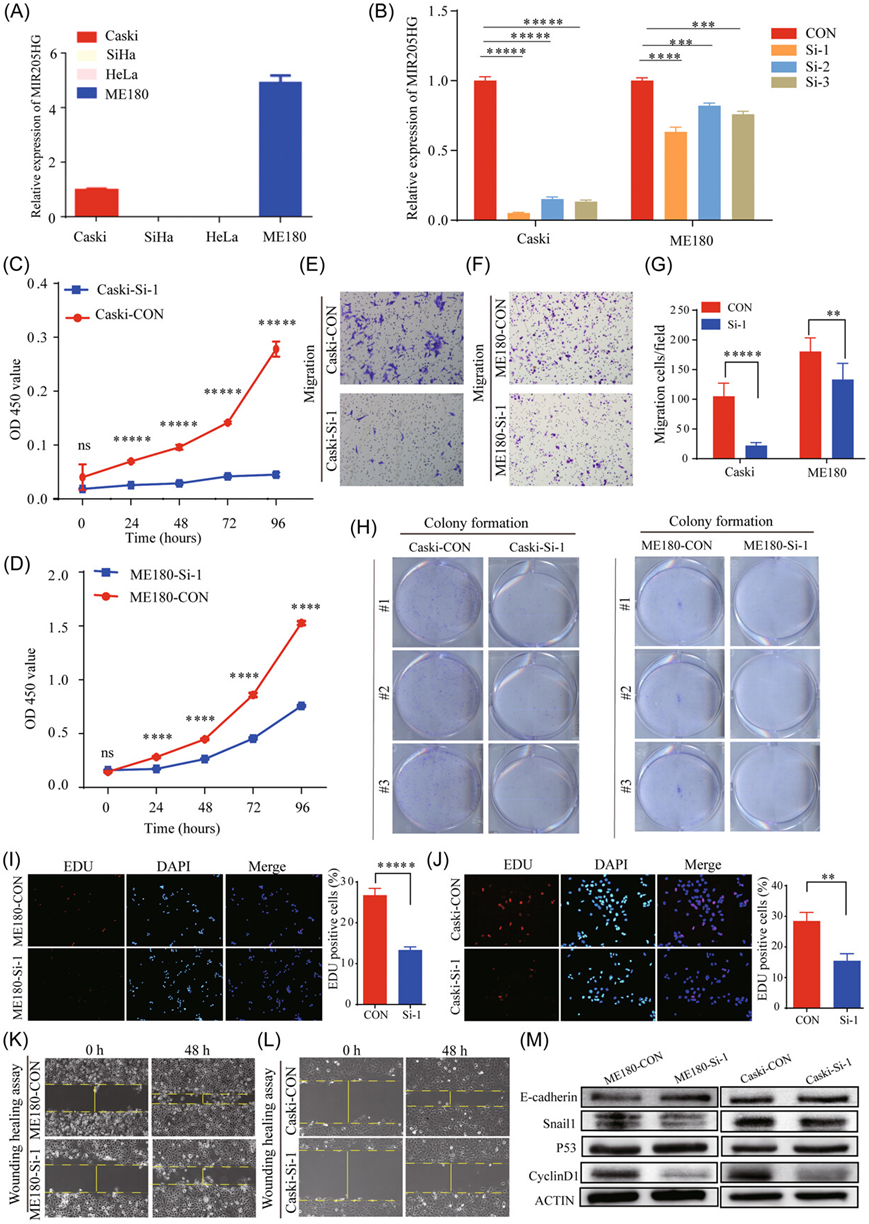
图6. MIR205HG的上调促进了宫颈癌的增殖和迁移。
(A) MIR205HG在 SiHa、HeLa、Caski 和 ME180 中的相对表达。(B)转染不同 siRNA 后 Caski 和 ME180 中MIR205HG的相对表达。(C-D)敲除MIR205HG后Caski和ME180的增殖能力降低。(E-G)敲除MIR205HG后,Caski 和 ME180 的迁移能力显著降低。(H) MIR205HG敲低后克隆形成能力降低。(I-J) Caski 和 ME180 中MIR205HG下调后 DNA 复制能力降低。(K-L) MIR205HG敲除后,伤口愈合能力下调。(M)相关蛋白表达的变化。
+ + + + + + + + + + +
结 论
本项研究通过 HPV 捕获测序、RNA-seq和WGBS分析获得了 50 名宫颈癌患者的多组学数据。在匹配的肿瘤和邻近肿瘤旁组织中检测到 985 个和 485 个 HPV 整合位点。其中,LINC00486、LINC02425、LLPH、PROS1 、KLF5、LINC00392、MIR205HG和NRG1 鉴定为高频 HPV 整合基因,其中包括 5 个新的复发基因。临床 II 期患者的 HPV 整合数量最高。HPV16 的 E6 和 E7 基因(而非 HPV18)显示断点明显少于随机分布。发生在外显子中的 HPV 整合与肿瘤组织中基因表达的改变有关,但与肿瘤旁组织中的基因表达改变无关。报告了在转录组或表观遗传水平上调控的 HPV 整合基因列表。他们还仔细检测了两个水平上相关调控模式的候选基因。PROS1和MIR205HG的启动子甲基化水平均与其基因表达呈负相关。进一步的实验验证证明,MIR205HG的上调可以促进宫颈癌细胞的增殖和迁移能力。本项研究数据为宫颈癌基因组中 HPV 整合的表观遗传学和转录组调控提供了新的图谱。
+ + + + +



 English
English


